Polihidroxibutirato Sintasa (PhaC), ¿el nuevo salvavidas ecológico?
Realizado por: Cintia Merino Piñón y Celia Juara Díaz (1ºC Biología Sanitaria – UAH)
Ya por 1862, de la mano de Estados Unidos, encontramos los primeros tipos de plástico que se empezaron a emplear. Estos consisten básicamente en polímeros formados a partir de largas cadenas moleculares obtenidas del petróleo. Desde sus inicios, todo apuntaba al éxito que tendrían, y al cambio radical que significaría para el humano y la producción mundial, a causa de sus múltiples usos y rentabilidad económica.
Pese a todo, con el tiempo hemos podido observar que el uso excesivo y los problemas que acarrea su mala degradación, nos han llevado a consecuencias tales como: la liberación masiva de CO2, la acumulación de miles de toneladas de plástico en muchos puntos del planeta, etc. Desgraciadamente, todo apunta a que la situación se agravará con los años si seguimos así, al no disponer de una opción más sostenible.
Es en este punto, donde entra en juego la ciencia, presentando ante el mundo a las “bacterias capaces de sintetizar plástico biodegradable”, Cupriavidus necator, Pseudomonas putida, Bacillus bataviensis, Allochromatium vinosum… las posibles soluciones.
¿QUÉ ES LA POLIHIDROXIBUTIRATO SINTASA (PhaC)?
En el transcurso de la investigación, iniciada en la década de los 80, en busca de una alternativa sostenible al plástico tradicional, centraron su atención en los “polihidroxialcanoatos (PHA)”. Estos son biopoliésteres sintetizados de forma intracelular por algunos microorganismos, utilizados como reserva de carbono y energía, al igual que ocurre con el glucógeno en animales y hongos.
Cupriavidus necator, también conocida como “Ralstonia eutropha” (perteneciente a la familia de las Burkholdariaceae), es una bacteria muy versátil, localizada en suelos y aguas, con capacidad para convertirse en una solución muy efectiva al problema del plástico. Se trata de una bacteria flagelada de forma alargada, gram negativa, con libertad de movimiento. Dependiendo de las condiciones ambientales, presentaría un metabolismo heterótrofo, o autolitotrofo.
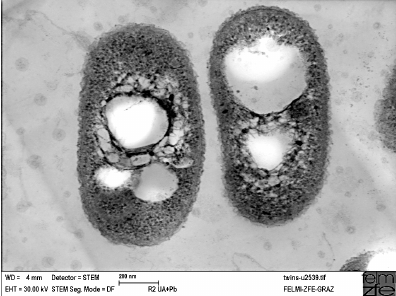
DOI: 10.5772/48737
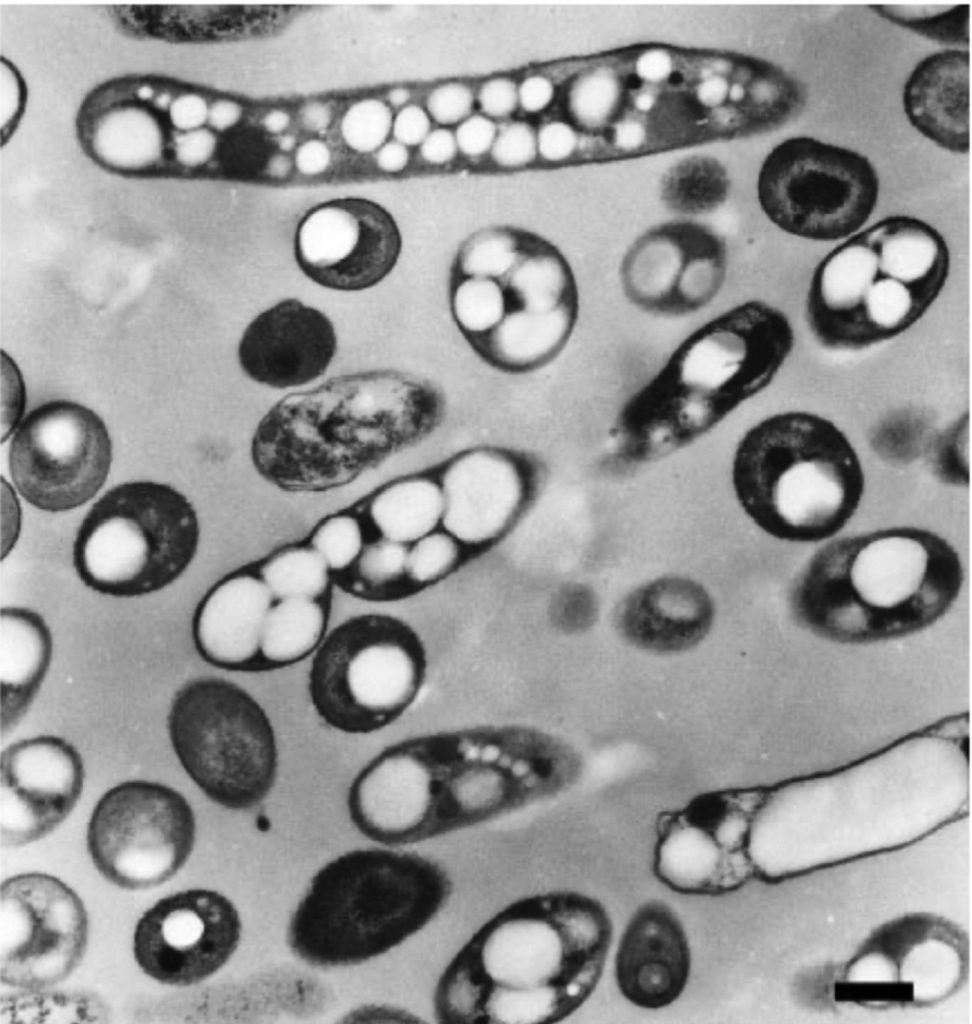
DOI: https://doi.org/10.1016/j.tibtech.2021.01.001
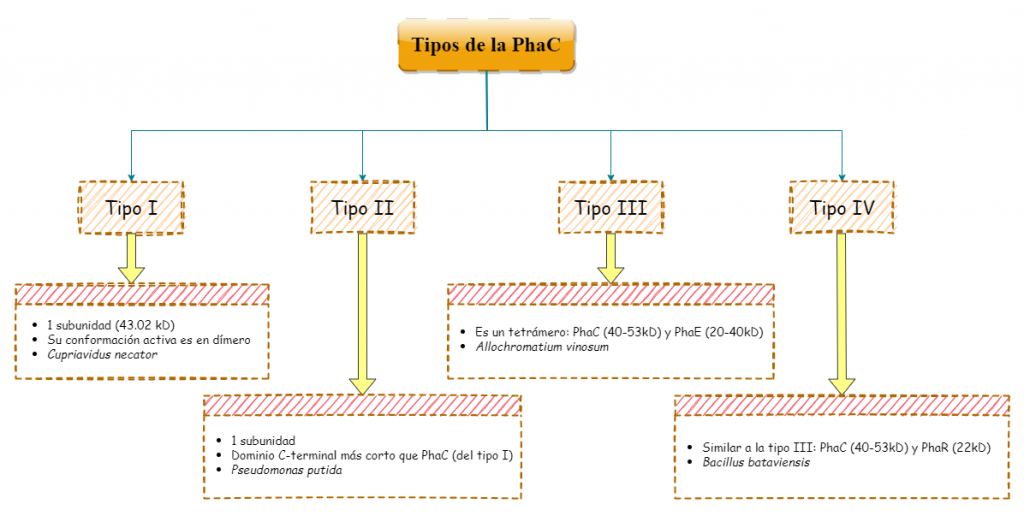
La síntesis de polihidroxibutirato (PHB), no sería posible sin la polihidroxibutirato sintasa (Pha C), responsable de la catálisis del proceso. Este grupo de enzimas presentes en muchos microorganismos sintetizadores de bioplásticos, se divide en 4 tipos distintos que difieren en su especificidad y la composición de sus subunidades (aunque todas ellas comparten el mismo mecanismo de acción y un centro activo similar al de las lipasas).
Tipos I y III
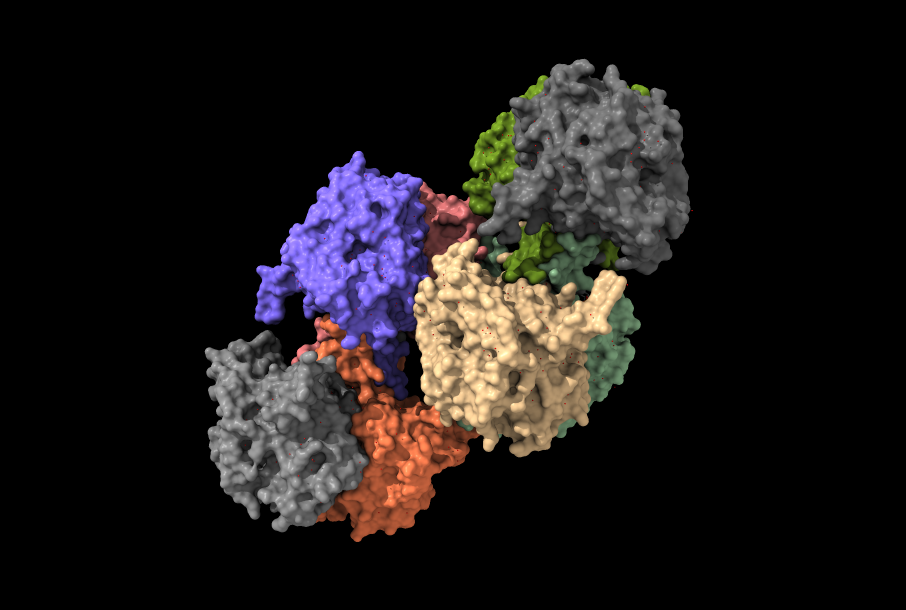
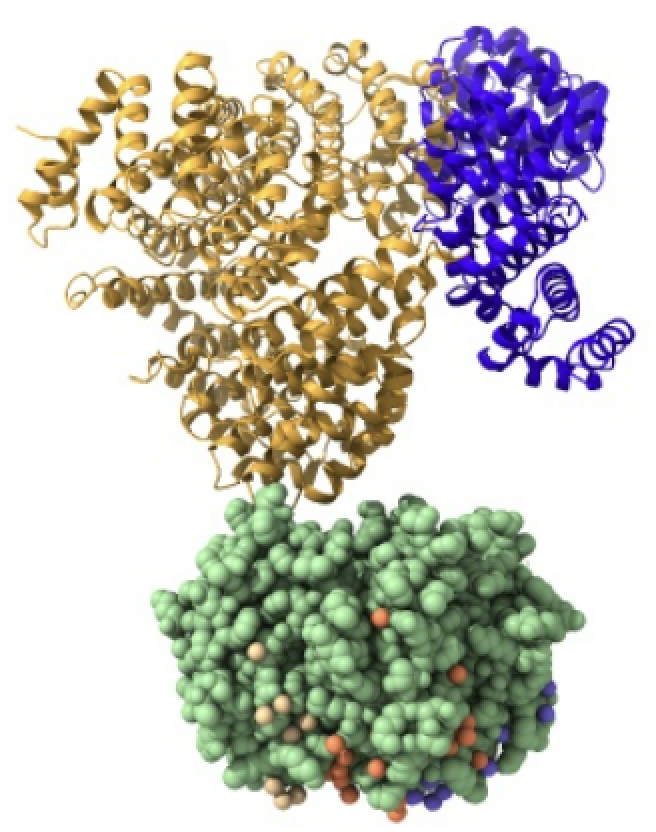
Los tipos I y III, los más estudiados hasta el momento, comparten esa especificidad por el HB como sustrato a la hora de sintetizar PHAs. El tipo I (Cupriavidus necator), se constituye únicamente de una cadena de polipéptidos de 589 residuos en total (una única subunidad, PhaC). Posee un dominio N-terminal (191 residuos) con funciones desconocidas por el momento, y un dominio C-terminal (398 residuos) encargado de la catálisis. Dicho dominio catalítico, se compone de una zona central de láminas-β, rodeada a ambos lados por α-hélices (aspecto que recuerda al centro activo de las lipasas, de ahí su parecido con esta enzima). No obstante, su conformación activa es un dímero, con sus dominios catalíticos enfrentados, que presenta una mayor actividad catalítica. El responsable de la dimerización, es un dominio helicoidal que se aleja del núcleo de la proteína, del que parte un bucle desordenado de 66 residuos que se integra a otra cadena polipeptídica, formando el dímero.
Por otra parte, el tipo III estaría compuesto de dos subunidades distintas formando un tetrámero: la PhaC (40-53 kD), responsable de la catálisis; y la PhaE (alrededor de 20-40 kD). Ejemplos de microorganismos que contienen este tipo de enzima son: Chromatium vinosum y Allochromatium vinosum.
Tipos II y IV
El tipo IV comparte una gran similitud con el tipo III: ambos son un dímero con dos subunidades distintas, aunque en lugar de PhaE como subunidad, este tipo tiene la PhaR (22kD). Esta variedad de polihidroxibutirato sintasa es propia de bacterias como: Bacillus cereus o B. bataviensis.
El tipo II está presente en muchos tipos de bacterias en la naturaleza, tales como Pseudomonas putida. Sería muy parecido al primer tipo, salvo que su dominio C-terminal presenta una secuencia más corta, y el sustrato sobre el que actúa cambia, existiendo variantes del tipo II con gran afinidad por el hidroxihexanoato (P. oleovorans).
Cabe destacar que a pesar de todas estas diferencias, todas las PhaC sintasas son enormemente similares entre ellas, presentando secuencias homólogas a las del primer tipo, en sus dominios catalíticos.
Otras enzimas
Por otro lado, la PhaC no es la única enzima que participa en la formación del bioplástico. Existen otras enzimas que desempeñan sus funciones durante la biosíntesis para formar los monómeros que componen el polímero.
Centrándonos en la Cupriavidus necator, la beta ketotiolasa (PhaA), es la enzima que inicia todo el proceso, al ser la encargada de comenzar la síntesis del hidroxibutirato. Esta transferasa, consiste en un tetrámero, de subunidades dimerizadas.
Finalmente, la síntesis de los monómeros termina tras la intervención de la acetoacetil CoA reductasa (PhaB), una oxidorreductasa que emplea NADH para reducir el acetoacetil CoA, obteniéndose el hidroxibutirato. Desgraciadamente, con respecto a su estructura tridimensional, a día de hoy sigue sin disponerse de suficiente información de los dominios que participan en la actividad de la enzimática. Aunque se cree que su estructura primaría es muy similar a una enzima que participa en la síntesis de ácidos grasos, la FabG.
ESTRUCTURA Y FUNCIONAMIENTO
El polihidroxibutirato, sintetizado por la bacteria Cupriavidus necator, es un polihidroxialcanoato de cadena corta, formado por la adición sucesiva de “n” unidades de hidroxibutirato. La biosíntesis comienza con el acetil-CoA, que puede provenir del metabolismo de carbohidratos, ácidos grasos e incluso proteínas, aunque la ruta más estudiada es la que comienza con los carbohidratos. La molécula de acetil-CoA es transferida, gracias a la enzima PhaA, a otro acetil-CoA, formándose acetoacetil-CoA.
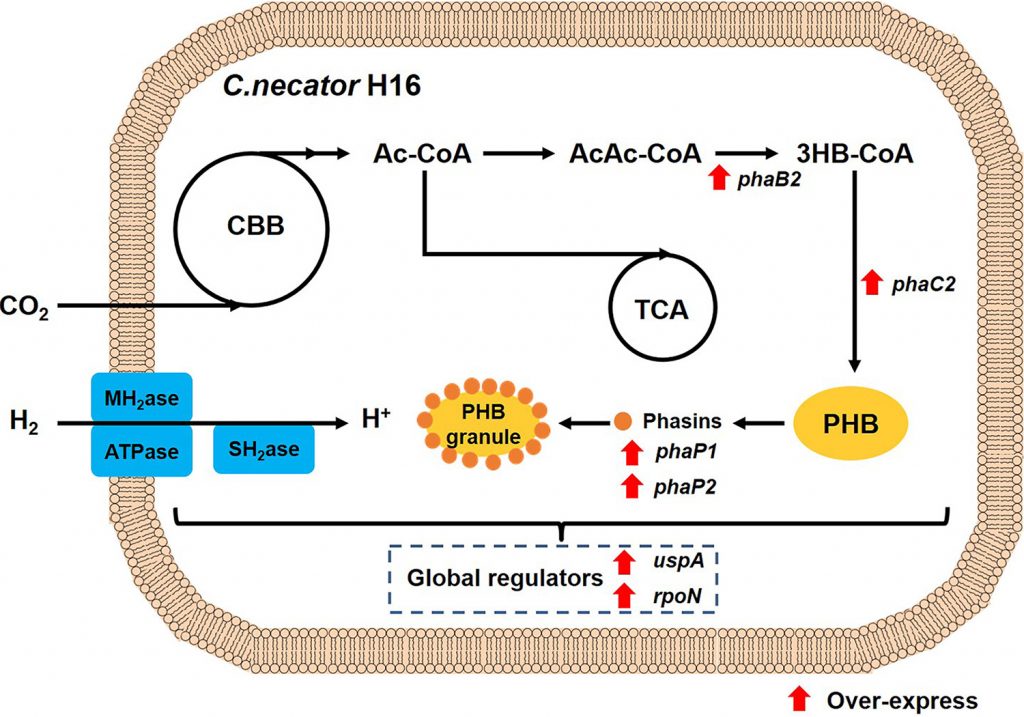
DOI: https://doi.org/10.1128/AEM.01458-21
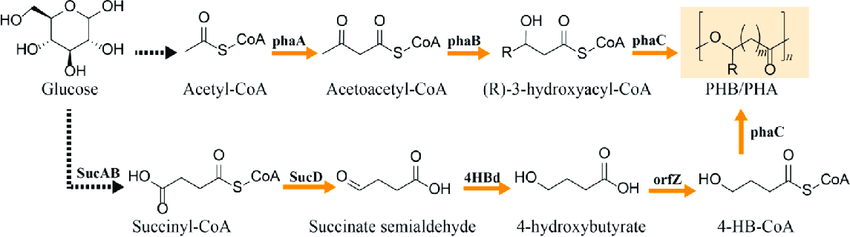
DOI: https://doi.org/10.1021/acs.chemrev.6b00804
En el siguiente paso consecutivo, la enzima PhaB reduce la molécula a hidroxibutirato, empleando para ello NADH como cofactor. Una vez obtenido el HB, comienza la formación del PHB, catalizada por la enzima PhaC.
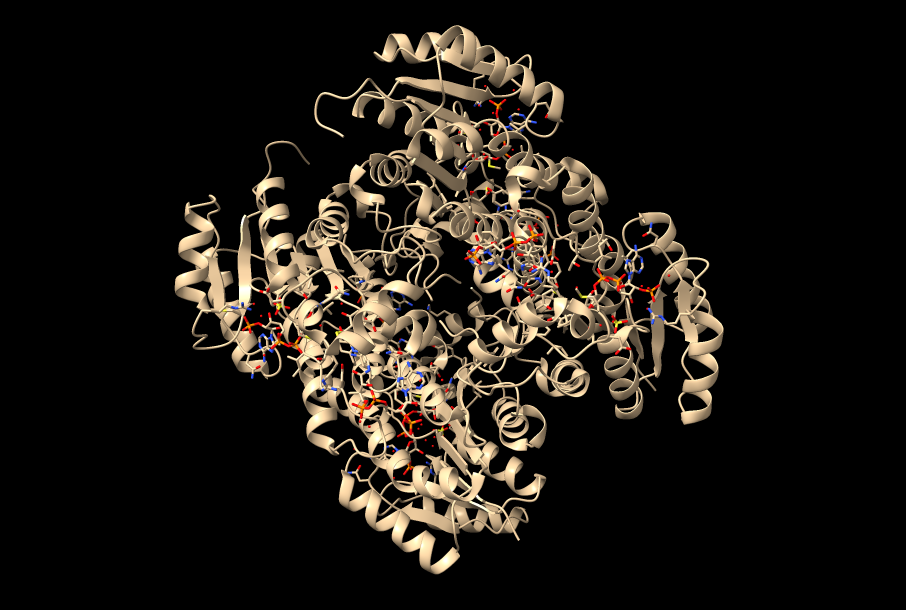
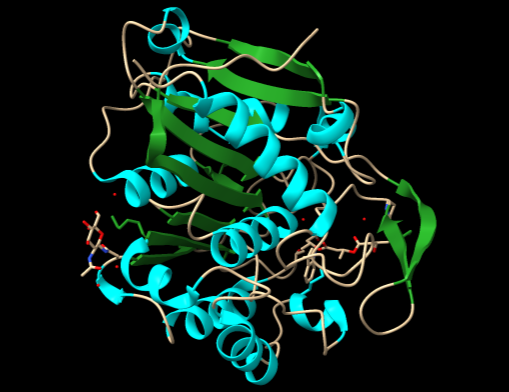
Enfocándonos, en el mecanismo de acción y la estructura de su dominio catalítico, diversas investigaciones bioquímicas llevaron a cabo la cristalización de esta enzima con el fin de estudiarlos. Con respecto al experimento, la estructura cristalizada presentaba una mutación: la cisteína del centro activo fue sustituida por alanina (Fig.10), otorgándole mayor estabilidad. En cuanto a la actividad enzimática de PhaC, se teorizaba que su funcionamiento era similar al de una lipasa, sintetizando el PHB de forma similar a los ácidos grasos. Ahora bien, actualmente el mecanismo de acción más aceptado consiste en un único sitio activo, porque la distancia que existe entre los dos centros activos del dímero (unos 33 Å) haría imposible el intercambio del bioplástico en proceso de ser sintetizado entre ambos.
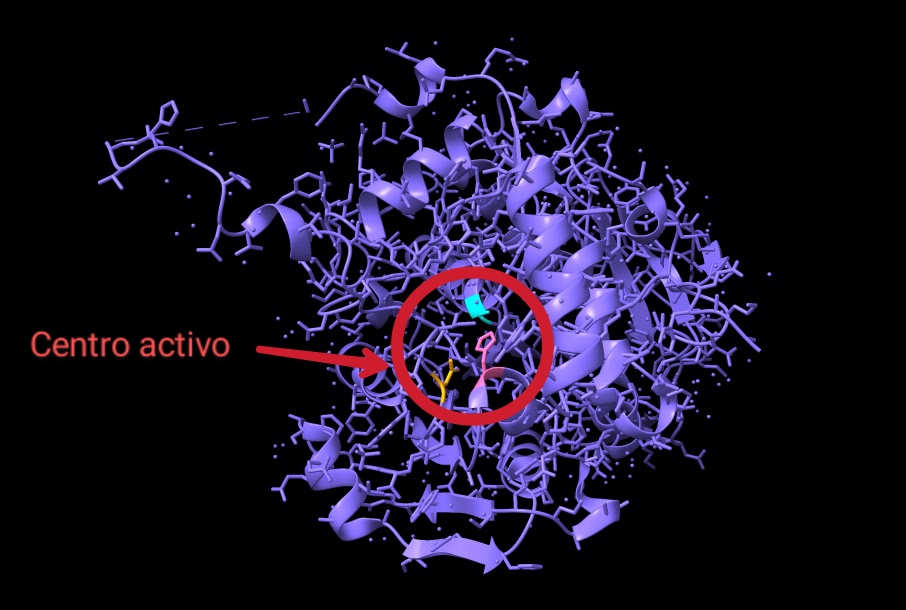
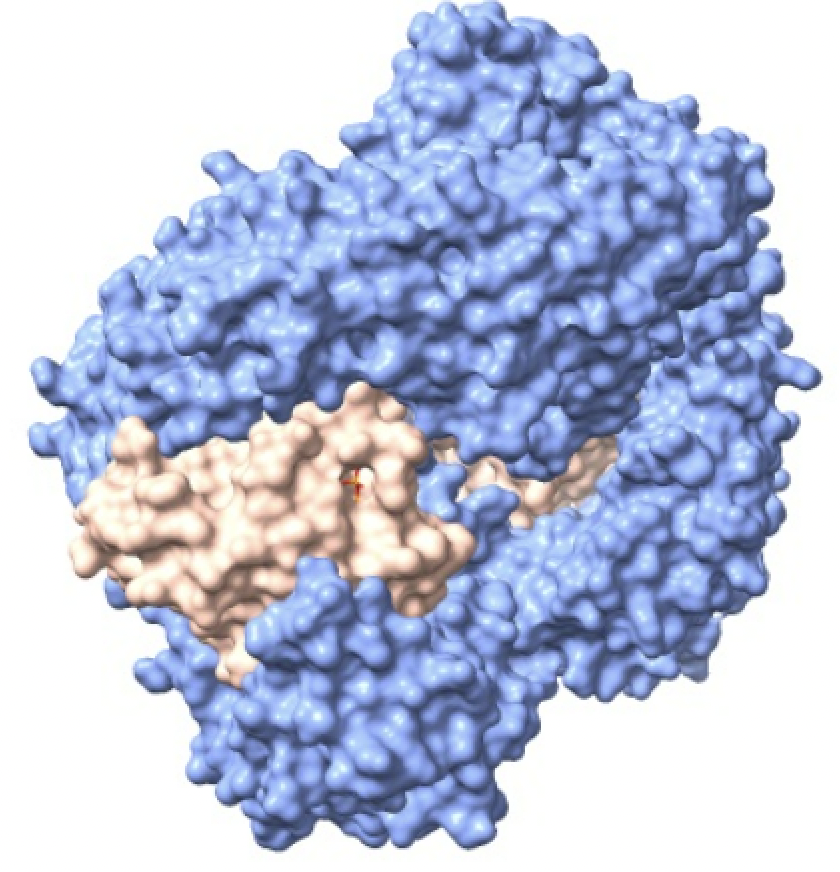
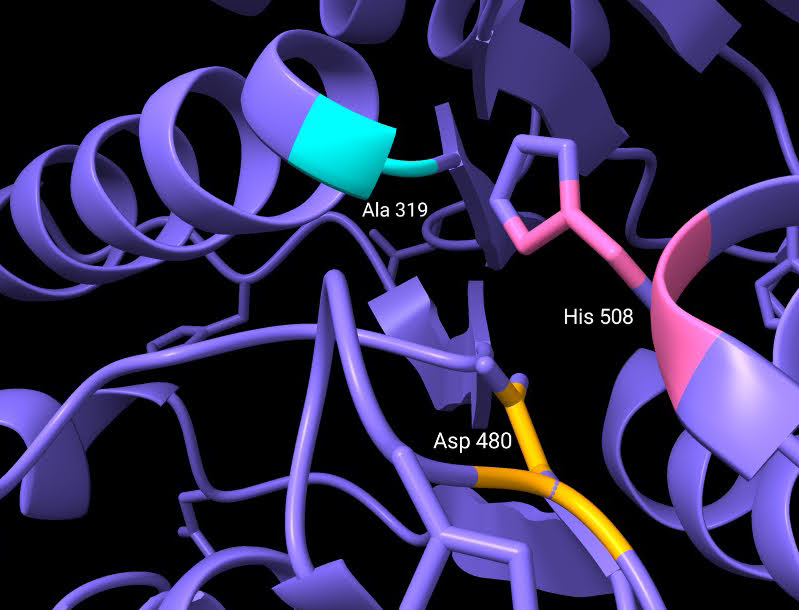
Los residuos que forman parte del centro activo de esta curiosa enzima son: Cys319, His508 y Asp480 (Fig.9). Estos se encuentran alojados en una cavidad situada a unos 10Å de la superficie proteica. La alanina del centro activo está localizada en lo que se conoce como “codo nucleófilo”, es decir, se halla entre una lámina beta y una alfa hélice. A su alrededor observamos un bucle, donde se ubica la histidina, separada por 3,5 Å de la alanina (cisteína en la enzima sin mutación) en la estructura cristalizada estudiada. Por último, el aspartato del centro activo se localiza justo detrás de la histidina, en otro bucle.
La entrada de los monómeros al centro activo de la enzima pudo observarse claramente gracias a un análisis esta estructura con un software, CAVER . Esta, consiste en un canal que accede directamente a una cavidad llena de agua en el interior de la proteína (contigua al centro activo) desde la superficie. Con unos 18 Å de longitud, este canal permite el acceso de la mitad de la molécula de HB al centro activo. Dos residuos de arginina, uno de cada monómero, están muy próximos a la apertura del canal, y su unión con el grupo CoA (del HB), es la causa del aumento de la actividad enzimática del dímero.
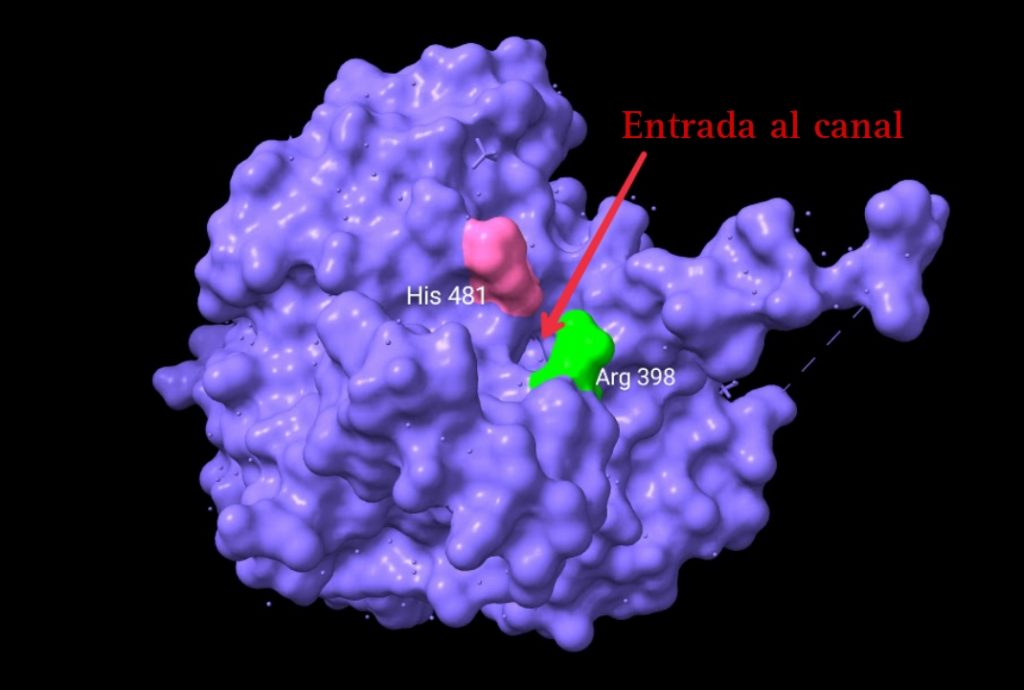
La formación del polímero comienza con la histidina (His 508) del centro activo, que se encargaría de desprotonar el tiol de la cisteína (Cys 319), mutada en alanina, gracias a la escasa distancia que las separa. De esta forma, se puede producir un enlace covalente entre la cisteína desprotonada y el primer monómero de HB de la cadena, creando una unión estable entre residuo y monómero. Es entonces cuando entra en acción el residuo de aspartato (Asp 480) que, debido al puente de hidrógeno con la His 508, participa de forma indirecta en la desprotonación de los monómeros de la cadena que se van añadiendo. La adición de un segundo monómero al “plástico” naciente requiere la correcta orientación del HB (unido covalentemente a la Cys 319), que se produce gracias a la naturaleza del propio enlace que los une, un enlace plano. En ese momento, a una distancia estimada de 2.8Å, se produce el ataque nucleofílico por parte de HB-Cys 319 a la nueva unidad en adición. Tras la incorporación de sucesivas unidades de HB obtendremos nuestro polímero de forma helicoidal levógira. Este será acumulado en forma de gránulos de tamaño considerable, 80 veces más pesados que el propio peso en seco del microorganismo.
En el modelo cristalizado y empleando de nuevo la herramienta CAVER se teorizó que la salida de esta cadena naciente tendría lugar por un canal compuesto por residuos hidrofóbicos, que se alejan en dirección a la superficie de la PhaC, hacia una cavidad aledaña al residuo N-terminal de la proteína y un residuo de aspartato, que participaría en la terminación de la cadena de PHA.
Para permitir el paso de este polímero por el canal de salida se cree que hay unos dominios conservados, dispuestos en las cercanías del canal, llevando a cabo una redistribución de esos aminoácidos hidrofóbicos que lo conforman, ensanchándolo hasta lograr el tamaño adecuado para el polímero.
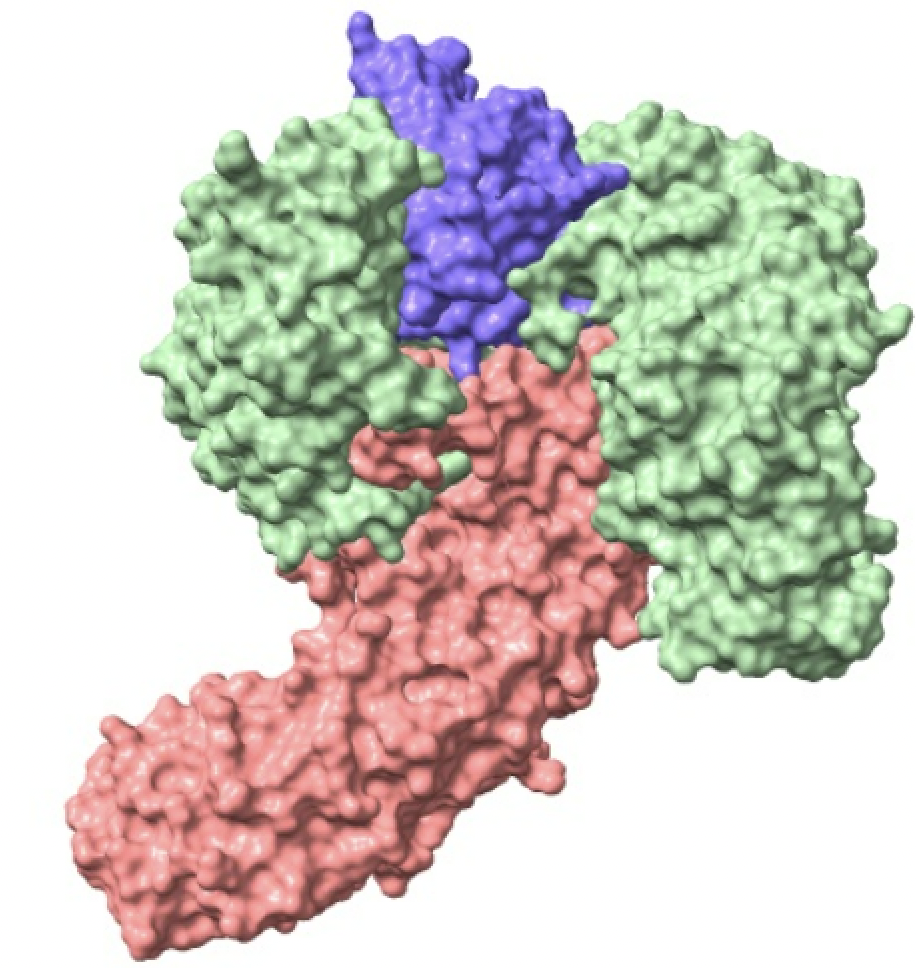
Con objeto de emplear el carbono almacenado, la bacteria usa una enzima capaz de hidrolizar el PHB, la polihidroxibutirato polimerasa. Esta hidrolasa estudiada a través de una estructura cristalina purificada obtenida de P. funiculosum, nos muestra que se compone por un único dominio, con un plegamiento alfa/beta formado por 8 láminas beta: 7 de ellas paralelas y la restante antiparalela, todas ellas rodeadas por alfa hélices. Su dominio catalítico lo forman Ser39, Asp121, y His155, residuos que se encuentran preservados en su dominio catalítico.
Cabe destacar que existen depolimerasas que poseen también la capacidad de degradar plástico extracelular, que no haya sintetizado la bacteria. En este caso la enzima presenta tres dominios esenciales: catalítico, enlazante y de unión al sustrato. Esta se encarga de romper en pequeñas unidades el bioplástico, gracias a la serina de su centro activo, de forma similar a como actúa la tripsina. En el caso de Cupriavidus necator, los monómeros resultantes de la hidrólisis son moléculas de HB, que la bacteria oxida a acetoacetato por medio de una deshidrogenasa, y posteriormente convierte en acetil-CoA (del que obtendrá energía incorporándolo a distintas vías metabólicas).
POLIHIDROXIALCANOATOS Y SU PRODUCCIÓN INDUSTRIAL
Dado que tenemos un amplio abanico de bacterias capaces de producir PHB, las técnicas y sistemas de cultivo son igualmente variados. Todas ellas pueden dividirse en dos grupos, dependiendo de las condiciones que necesitan para producir los PHAs. El primer grupo, donde encontramos a Cupriavidus necator, lo forman microorganismos que necesitan que precisan escasez de nutrientes y alta disponibilidad de carbono. Con respecto al segundo grupo, este no necesita de una situación de escasez, ya que son capaces de acumularlo en grandes cantidades durante su etapa de crecimiento.
GRUPO 1: Limitación de nutrientes
Para realizar un cultivo con las bacterias del primer grupo necesitamos un proceso dividido en 2 etapas: la primera de ellas tiene como objetivo obtener la biomasa necesaria para la producción, por lo que no existe limitación de nutrientes; la segunda etapa comienza una vez alcanzada la biomasa requerida, alguno de los nutrientes esenciales está limitado, promoviendo que las bacterias sinteticen el polímero (lo acumulan en gran cantidad formando esos gránulos tan llamativos).
GRUPO 2: Sin limitación de nutrientes
En el segundo grupo la estrategia que se sigue para obtener el bioplástico consiste en una única etapa, sin limitación de nutrientes. Los microorganismos disponen de ellos en grandes cantidades en el medio de cultivo, lo que les permite mejorar su rendimiento y aumentar de tamaño.
Obtención, proceso industrial
Una de las primeras empresas en tratar de producir el bioplástico en masa fue ICI, una empresa inglesa. Para el proceso de producción finalmente se decidieron por Cupriavidus necator, que lo sintetiza en dos etapas. El cultivo tiene a su disposición glucosa y fosfato limitado, suficiente para alcanzar una biomasa considerable y pasar a la segunda etapa, en la que las bacterias comienzan a almacenar el bioplástico. A la hora de recuperar el polímero empleamos metanol caliente para retirar los restos celulares (lípidos, fosfolípidos, etc.), y se extrae el PHB con cloroformo o cloruro de metileno. Por último, se filtra la mezcla, se enfría y precipita, luego es secada al vacío.
Tras diversas pruebas, finalmente se determinó que para obtener 1g de PHB con la cepa de la bacteria empleada, eran necesarios 3g de glucosa para permitir su desarrollo y proporcionarle la fuente de carbono para formar el plástico.
Una propiedad muy importante, a parte de su relativamente sencillo proceso de obtención, es el hecho de que es un plástico completamente biodegradable. A pesar de la gran cantidad de monómeros posibles capaces de conformar los polihidroxialcanoatos, existen diversas bacterias, e incluso hongos, capaces de degradarlos bajo cualquier condición y sin generar ningún tipo de residuo tras el proceso. Mediante las reacciones de hidrólisis citadas anteriormente en las que participan las PHB depolimerasas de esos microorganismos, se produce la ruptura del bioplástico (que se emplea como fuente de energía dentro de la bacteria descomponedora). Este proceso tiene lugar a una velocidad considerable, que depende de diversos factores (pH, temperatura, dimensión del polímero, etc.).
¿NOS AGUARDA UN FUTURO CON ELLOS?
A día de hoy, tener a nuestra disposición este polímero totalmente biodegradable y que no genera residuos con su producción, nos permitiría llevar un estilo de vida mucho menos dañino para el planeta, puesto que todos aquellos problemas que encontramos con la producción y degradación de los plásticos tradicionales desaparecerían.
Presentan muchas aplicaciones y gracias a sus diversas propiedades pueden ser utilizados en campos muy diversos, como en: farmacia, industria, agricultura, medicina o incluso como materias primas. Es por ello, que tratamos de producirlo en masa y de la forma más económica posible, intentando buscar microorganismos capaces de producirlos, empleando para ello residuos de las propias industrias en algunos casos, como sustrato en el proceso de síntesis. “Biocycle”, una empresa brasileña, es una de las que empleó los residuos de su propia producción como sustrato, en el año 2001.
Actualmente los científicos investigan a fondo la estructura molecular de la PhaC, enzima sin la cual este proceso no tendría lugar, con el fin de conseguir modificar molecularmente el bioplástico para otorgarle funciones mucho más específicas y amplias. Y aunque queda un largo camino por recorrer en cuanto a la investigación sobre estos aspectos, los PHAs nos acercarían un paso más hacia una sociedad sostenible, en la que los residuos generados sean mínimos y lo menos contaminantes posibles. Desgraciadamente, por el momento, se sigue sin poder hacer competencia al plástico tradicional, sobre todo en el ámbito económico.
DOI: 10.1186/s12934-020-01342-z
BIBLIOGRAFÍA
1- Chek, M.F., Hiroe, A., Hakoshima, T. et al (2019). PHA synthase (PhaC): interpreting the functions of bioplastic-producing enzyme from a structural perspective. Appl Microbiol Biotechnol 103, 1131–1141. DOI: https://doi.org/10.1007/s00253-018-9538-8.
2- Eun-Jung Kim, Kyung-Jin Kim, (2014). Crystal structure and biochemical characterization of PhaA from Ralstonia eutropha, a polyhydroxyalkanoate-producing bacterium, Biochemical and Biophysical Research Communications, Volume 452, Issue 1, Pages 124-129, ISSN 0006-291X. DOI: https://doi.org/10.1016/j.bbrc.2014.08.074. (https://www.sciencedirect.com/science/article/pii/S0006291X14014971)
3- GONZÁLEZ GARCÍA, Yolanda, MEZA CONTRERAS, Juan Carlos, GONZÁLEZ REYNOSO, Orfil, & CÓRDOVA LÓPEZ, Jesús Antonio. (2013). Síntesis y biodegradación de polihidroxialcanoatos: plásticos de origen microbiano. Revista internacional de contaminación ambiental, 29(1), 77-115. (http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0188-49992013000100007&lng=es&tlng=es)
4- João M.B.T. Cavalheiro, M. Catarina M.D. de Almeida, Christian Grandfils, M.M.R. da Fonseca. (2009). Poly(3-hydroxybutyrate) production by Cupriavidus necator using waste glycerol,Process Biochemistry, Volume 44, Issue 5,Pages 509-515, ISSN1359-5113, DOI: https://doi.org/10.1016/j.procbio.2009.01.008. (https://www.sciencedirect.com/science/article/pii/S1359511309000233)
5- Mezzolla, V.; D’Urso, O.F.; Poltronieri, P (2018).Role of PhaC Type I and Type II Enzymes during PHA Biosynthesis. Polymers, 10, 910. DOI: https://doi.org/10.3390/polym10080910.
6- Tamao Hisano, Ken-ichi Kasuya, Yoko Tezuka, Nariaki Ishii, Teruyuki Kobayashi, Mari Shiraki, Emin Oroudjev, Helen Hansma, Tadahisa Iwata, Yoshiharu Doi, Terumi Saito, Kunio Miki, (2006). The Crystal Structure of Polyhydroxybutyrate Depolymerase from Penicillium funiculosum Provides Insights into the Recognition and Degradation of Biopolyesters, Journal of Molecular Biology, Volume 356, Issue 4, Pages 993-1004, ISSN 0022-2836, DOI: https://doi.org/10.1016/j.jmb.2005.12.028. (https://www.sciencedirect.com/science/article/pii/S0022283605015901)
7- The Protein Data Bank H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) Nucleic Acids Research, 28: 235-242. DOI: 10.1093/nar/28.1.235
8- Wittenborn, E.C; Jost, M; Wei, Y; Stubbe, J; Drennan, C.L. (2016). Structure of yhe catalityc Domain of the Class I Polyhydroxybutyrate Synthase from Cupriavidus necator*, J Biol Chem, 291: 25264-25277 . DOI: https://doi.org/10.1074/jbc.M116.756833.
9- Yu Jung Sohn, Jina Son, Seo Young Jo, Se Young Park, Jee In Yoo, Kei-Anne Baritugo, Jeong Geol Na, Jong-il Choi, Hee Taek Kim, Jeong Chan Joo, Si Jae Park, (2021). Chemoautotroph Cupriavidus necator as a potential game-changer for global warming and plastic waste problem: A review, Bioresource Technology, Volume 340, 125693, ISSN 0960-8524. DOI: https://doi.org/10.1016/j.biortech.2021.125693. (https://www.sciencedirect.com/science/article/pii/S0960852421010348)