C. Menor-Salvan. En construcción.
Toda la Biología se basa en un aspecto molecular conceptualmente simple: el control de la arquitectura de la molécula de DNA y su interacción con proteínas. La arquitectura del DNA posibilita que complejos proteicos interacciones con ella, posibilitando la replicación y la transcripción de la información genética. El manejo de la arquitectura de la molécula es la base de varios tratamientos farmacológicos, como la quimioterapia del cáncer.
El DNA se presenta en varias estructuras secundarias, todas ellas de importancia biológica. La transición de unas a otras es esencial en el control de algunos fenómenos clave, como la replicación o la homeostasis de los telómeros. Veamos sus tipos principales:
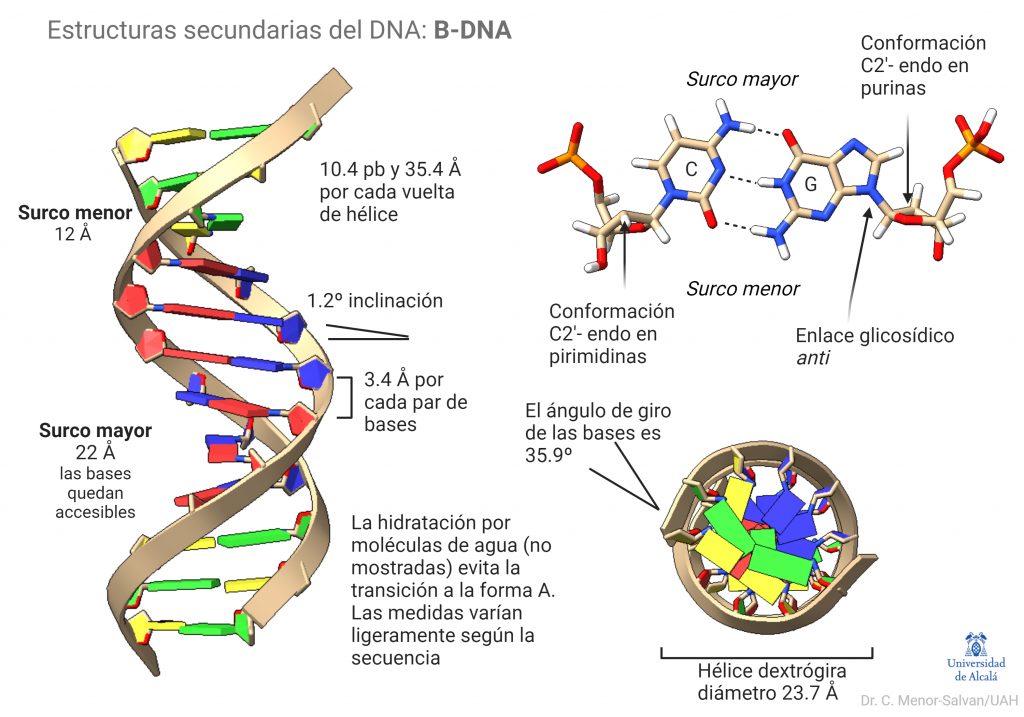
Estructura del B-DNA
Esta forma canónica del DNA es la más frecuente y relevante biológicamente. En la siguiente figura se muestra una estructura de B-DNA ideal y sus características estructurales relevantes. El B-DNA real puede sufrir pequeñas variaciones de medidas y ángulos según la secuencia, lo que juega un papel esencial en el reconocimiento de secuencias por proteínas. Además, la molécula de DNA sufre giros y torsiones al unirse a proteínas. Estos cambios topológicos son la base de la función de la molécula.
La estructura del DNA se forma por la repulsión de los fosfatos y el efecto hidrófobo, que favorecen la formación de una doble hélice. Las bases se apilan interaccionando gracias a los enlaces pi de sus anillos aromáticos, lo que estabiliza la estructura. Finalmente, algo que no suele representarse, es el papel esencial del agua. Moléculas de agua hidratan el B-DNA y forman una estructura que estabiliza la hélice y posibilita su papel biológico. Si se deshidrata la molécula, sufre una transición a la forma A-DNA. Esta transición tuvo gran relevancia en el descubrimiento de la estructura del DNA.
En la siguiente estructura en 3D podemos verlo en movimiento. Estructura propia, creada por el profesor Loren Williams (Georgia Institute of Technology)
Ideal DNA B sequence structure by cmenorsalvan on Sketchfab
Unión de oxaliplatino a DNA
Oxaliplatin bound to DNA by cmenorsalvan on Sketchfab
Obsérvese cómo, en el ejemplo anterior, el oxaliplatino actúa provocando una torsión en la doble hélice del DNA. Esta torsión, de aproximadamente 30 grados hacia el surco mayor, altera la arquitectura del DNA y bloquea la replicación.
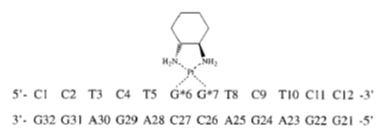
Estructura basada en PDB 1IHH
Estructura del A-DNA
Estructura del Z-DNA
Esta estructura secundaria es menos común que la B-DNA o canónica, pero se han encontrado evidencias de que existe en las células y podría jugar un papel en la regulación de la expresión de genes o en recombinación. Se diferencia claramente del B-DNA en varios aspectos: es una hélice levógira, frente al B-DNA que es dextrógiro. Los surcos mayor y menor son menos llamativos que en el B-DNA, pero en el surco mayor las bases quedan más expuestas. Esto le confiere al Z-DNA una mayor capacidad para complejar iones metálicos. El andamiaje de fosfatos o backbone se torsiona y adopta una apariencia de zigzag. La transición B->Z se da más fácilmente en secuencias ricas en alternancias de C y G y se estabiliza por unión con iones y algunas moléculas.
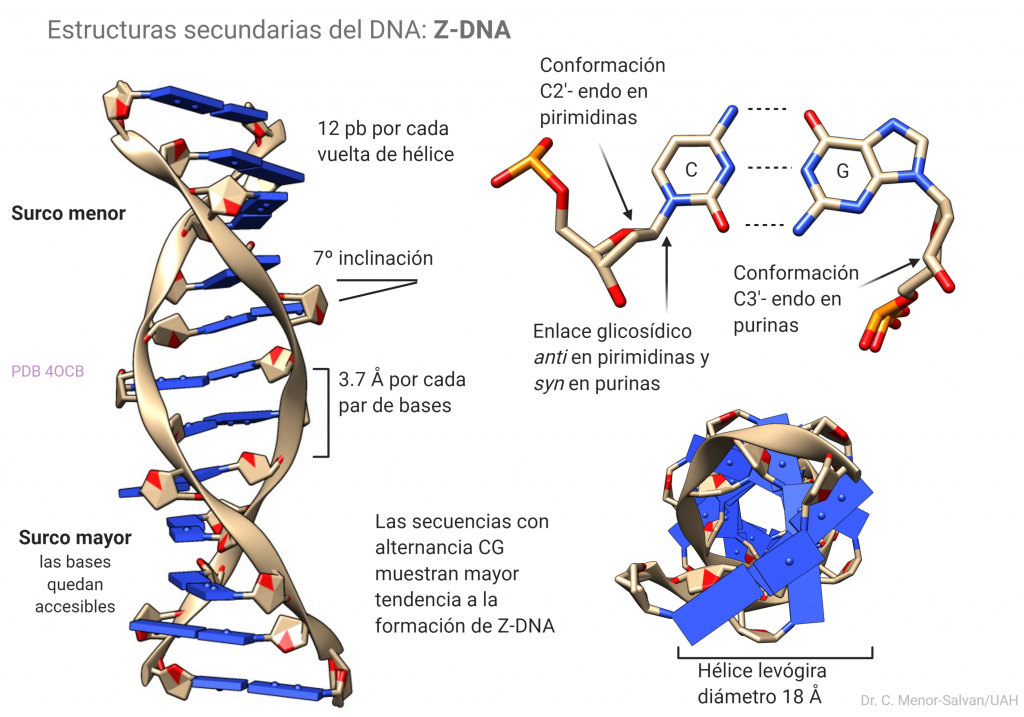
Se ha observado in vitro que la espermina se une al DNA-Z aumentando su estabilidad y favoreciendo la transición B-DNA–>Z-DNA. Es posible que esto tenga alguna relevancia biológica en procesos de regulación, pero aún queda mucho por conocer sobre el papel biológico de ésta estructura.
En el mundo de la publicidad, cine y televisión se suele representar incorrectamente el DNA y es poco común observar el B-DNA canónico, la estructura secundaria mayoritaria en la célula. Curiosamente, la mayoría de las representaciones se asemejan con mucha aproximación a la estructura del Z-DNA, como en éste caso:

G-cuadruplexo
G-quadruplex by cmenorsalvan on Sketchfab
Motivos-i o C-tetraplexos
Este tipo de estructura es complementaria a los G-cuadruplexos y su importancia biológica se va revelando gradualmente. Los motivos-i se forman en secuencias CCCBBBCCCBBBCCCBBBCCCBBB (B=timina o adenina).
El plegamiento en motivo-i requiere que las citosinas se protonen alternativamente, de modo que se establecen apareamientos no canónicos C-C+. Estos apareamientos no son ni Watson-Crick ni Hoogsteen y dependen de la protonación, por lo que ésta estructura es estable únicamente a pH 5.5-5.8. A pH superior, se desprotona la citosina y se produce la transición del DNA a estructura canónica.
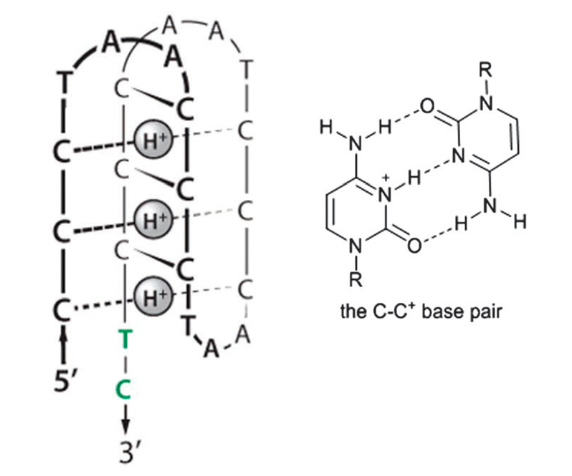
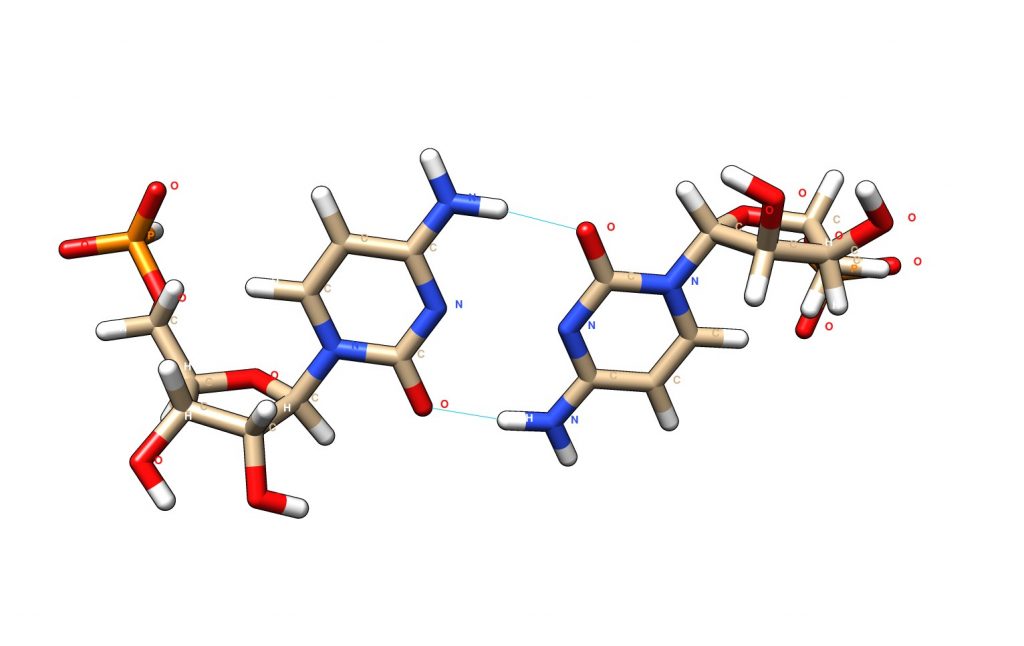
Si bien el pH ligeramente ácido que requiere la formación de los pares C-C+ no es común en el interior celular, se ha observado que los motivos-i se forman a pH neutro compatible con el medio intracelular, posiblemente por formación de estructuras híbridas o complementarias con los G-cuadruplexos.
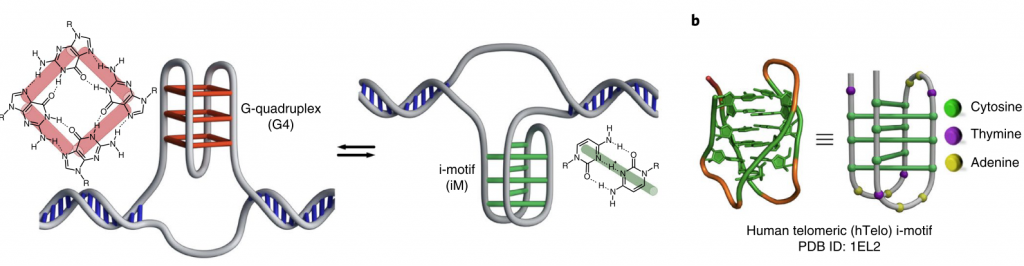
No se entiende bien aún cómo se forman y regulan los motivos-i. El pH podría ser un factor modulador clave, dado que pequeñas variaciones pueden estabilizar o inestabilizar el motivo. A su vez, los motivos-i pueden jugar un importante papel modulador en regiones promotoras y teloméricas del DNA.
Referencias
Gillingham, D., Geigle, S. and Anatole von Lilienfeld, O. (2016) ‘Properties and reactivity of nucleic acids relevant to epigenomics, transcriptomics, and therapeutics’, Chem. Soc. Rev. Royal Society of Chemistry, 45(9), pp. 2637–2655. doi: 10.1039/C5CS00271K.
Zeraati, M., Langley, D. B., Schofield, P., Moye, A. L., Rouet, R., Hughes, W. E., Bryan, T. M., Dinger, M. E. and Christ, D. (2018) ‘I-motif DNA structures are formed in the nuclei of human cells’, Nature Chemistry. Springer US. doi: 10.1038/s41557-018-0046-3.