LA PAPIROFLEXIA MOLECULAR: XCL1 y PrNp
Andrea Chiloeches Lasa y Álvaro Criado Expósito, 3º Biología Sanitaria
INTRODUCCIÓN
Las proteínas se pliegan gracias a la asistencia de las denominadas chaperonas moleculares (individuales) y chaperoninas (poliméricas). Estas proteínas actúan como enzimas acelerando el correcto plegamiento y el paso del estado mal plegado al nativo, al disminuir la barrera energética que los separa. También, previenen la agregación y disocian pequeños agregados.
La teoría inicial sobre el plegamiento de proteínas fue propuesta por el químico Christian B. Anfinsen. Tras una serie de experimentos, estableció las bases de la química de proteínas, con el dogma “A cada cadena polipeptídica le corresponde una conformación concreta única y, además, toda la información necesaria para adoptarla está contenida en su secuencia de aminoácidos”.
Esta hipótesis de Anfinsen, actualmente se mantiene valida y vigente, especialmente cuando se aplica al plegamiento de proteínas aisladas. No obstante, es cierta la existencia de excepciones a la regla, que son muy frecuentes en la Biología, en la que la individualidad juega un papel muy importante.
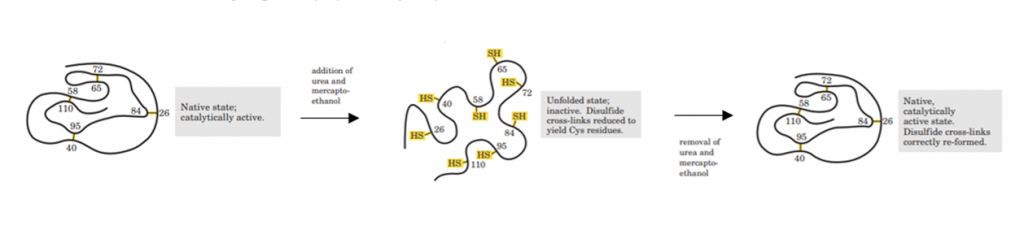
[Figura 1. Experimento de Anfinsen para demostrar el plegamiento de proteínas. La urea desnaturaliza la proteína y el mercaptoetanol reduce los enlaces disulfuro, quedando 8 residuos de cisteina. La renaturalización requiere el restablecimiento de los puentes disulfuro cruzados correctos. Fuente: Lehninger “Principles of Biochemistry”].
La mayoría de las proteínas se pliegan en una única conformación funcional. Sin embargo, algunas lo hacen en más de una estructura para desempeñar diferentes funciones cuando es necesario. Es el caso de las proteínas metamórficas que pueden estar en dos o más plegamientos distintos, reversibles, para desempeñar funciones distintas.
El estudio de las proteínas dimorfas surgió por el descubrimiento de una clase de proteínas que presentaban más de una conformación estable para llevar a cabo funciones diferentes. Así, se observó la ventaja que supone a la célula que una única proteína sea capaz de realizar dos funciones, pues así solo es necesaria la transcripción y traducción de un gen.
Estas proteínas tienen la capacidad de responder a distintas situaciones, alternando rápidamente entre sus plegamientos y es por ello, que han evolucionado como moléculas que maximizan la función idónea en el lugar y momento adecuado.
No obstante, no todo son beneficios. Algunas proteínas metamórficas pueden adquirir conformaciones alternativas, que provocan ciertas enfermedades, en general neurodegenerativas, como el Alzheimer, el Parkinson o la encefalopatía espongiforme transmisible, caracterizadas por la presencia de agregados insolubles, derivados de la degradación de la propia proteína o por las proteínas desordenadas en tejidos.
Actualmente, se conocen unas 90 proteínas metamórficas con más de 30 tipos de funciones biológicas. Para ahondar en el concepto, vamos a hablar de las dos proteínas dimorficas, XCL1 y PrNp.
XCL1
La XCL1, también conocida como linfotactina, la expresan de forma selectiva las células T activadas, en particular CD8+, y es el único miembro de la familia de quimiocinas C que presenta alternancia estructural. Es una proteína que actúa como quimioatrayente para los linfocitos, es decir, que presenta un papel regulador importante en el tráfico de linfocitos y la inflamación. Se ha demostrado que la XCL1 emite señales a través del receptor 1 de quimiocinas XC (XCR1), y se detecta en niveles más altos en el bazo, ya que es un órgano linfoide.
La XCL1 surgió como una proteína corriente, con una única conformación. Sin embargo, tras años de estudio y observación, se descubrió en la evolución de la proteína, que perdió uno de los puentes disulfuro de su conformación nativa, lo que permitió la aparición de un segundo plegamiento alternativo y de nuevas funciones.
La proteína, alterna, más de una vez por segundo, entre una conformación monomérica canónica y un dímero sándwich de láminas beta. La transición de una estructura a otra, se puede ver afectada por factores físicos como cambios de temperatura, de fuerza iónica o la entrada de compañeros de fijación, que alteran el equilibrio que hay entre sus posibles plegamientos.
Las dos funciones que desempeña la proteína:
– Actúa como una quimiocina señalizadora que se fija a receptores de los leucocitos para que combatan las infecciones (en el primer plegamiento).
– Actúa como un análogo de antibiótico, destruyendo las bacterias invasoras (en el segundo plegamiento).
De esta manera, dependiendo de la función que tiene que desempeñar la proteína, se favorece el desplazamiento del equilibrio hacia la conformación idónea. Así, cuando las XCL1 deben llevar a cabo su función destructora, se favorece la conformación que interacciona con las membranas y las permite rodear el patógeno microbiano para eliminarlo. Sin embargo, en otras situaciones y lugares del organismo se facilita con más frecuencia el otro plegamiento, que les permite anclarse a los receptores de los glóbulos blancos para movilizarlos y que sean capaces de combatir infecciones.
En definitiva, el desarrollo de la estructura alternativa de XCL1, potencia su actividad y le proporciona al organismo un control más exhaustivo de las defensas contra microorganismos, acelerando la respuesta inmune ante una infección.
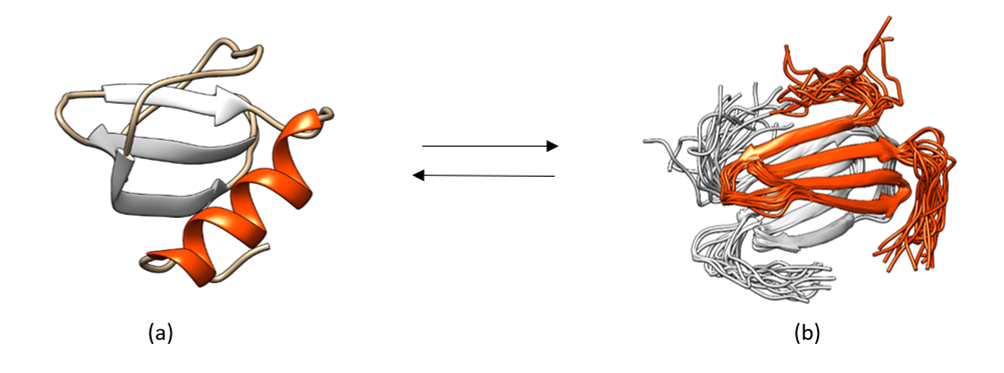
[Figura 2. Equilibrio de las conformaciones de la proteína XCL1. (a) plegamiento monomérico canónico (b) dímero sándwich hoja beta. Fuente: estructuras, obtenidas personalmente del programa UCSF Chimera, con ID: 4HED (PDB) y 2JP1 (PDB)]
PrNp
La segunda proteína metamórfica de la que vamos a hablar es la PrNp o proteína priónica.
La PrNp, es una proteína unida a azúcares que se encuentra espontáneamente en muchos tipos celulares en su forma PrPc. Esta proteína, puede sufrir una modificación en su conformación y plegarse de forma errónea, dando lugar a una isoforma patogénica (PrPsc) causante de enfermedades priónicas neurodegenerativas.
Ambas formas, son codificadas por el mismo gen, PRP, y poseen la misma secuencia, aunque distinta estructura terciaria. Por ello, no es posible que provengan de la maduración del mRNA, pues la secuencia codificante para PrP está contenida en un único exón. Así, se ha propuesto el carácter metamórfico de PrPc y que PrPsc sea un derivado de una modificación post-traduccional. Esto explica que, aunque los niveles de mRNA sean continuos, la isoforma patogénica se acumule en el tejido del animal infectado y evolucione la enfermedad.
La proteína priónica celular (PrPc) es una glicoproteína que suele encontrarse anclada a la membrana plasmática a través de una molécula de GPI (glicosilfosfatidilinositol). Se sitúa en la superficie celular y participa en procesos de transducción de señales, adhesión y reconocimiento celular. Está constituida por una sola cadena peptídica y presenta una estructura secundaria rica en hélices alfa, en una proporción 4:2, respecto a láminas beta.
A diferencia, la proteína priónica scrapie (PrPsc) presenta una estructura secundaria con un 45% de láminas beta y un 30% de hélices alfa. Es la variante de la proteína celular, que además de presentar una estructura diferente, pierde su función. El carácter patogénico de esta proteína reside en la formación de placas amiloides extracelulares, que se almacenan en el tejido cerebral, constituidas por los fragmentos de la proteína una vez ha sido degradada.
Debido al conocimiento del carácter metamórfico y de las distintas estructuras de esta proteína, hoy en día, se avanza en el estudio de su comportamiento y se investiga la posibilidad del replegamiento de la proteína PrP celular en presencia de la proteína priónica scrapie, como una posible cura para la encefalopatía priónica.
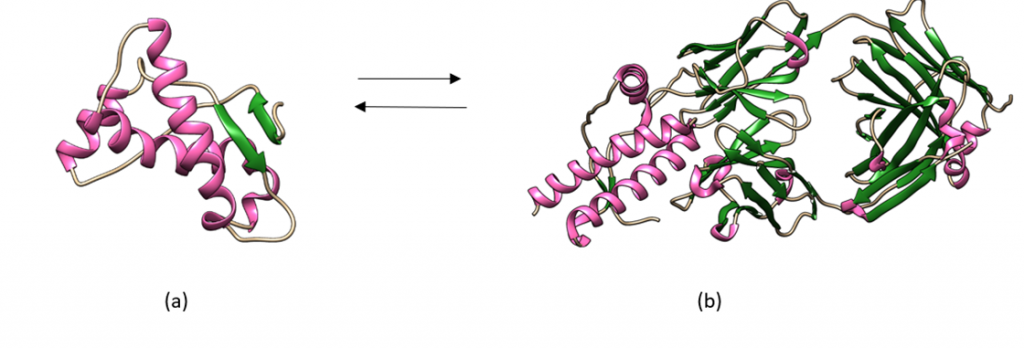
[Figura 3. Equilibrio de las conformaciones de la proteína priónica. (a) Proteína priónica celular (PrPc), con una estructura rica en hélices alfa, (b) Proteína priónica scrapie (PrPsc), con una estructura abundante en láminas beta. Fuente: estructuras obtenidas personalmente del programa UCSF Chimera, a partir de PDB con los ID: 1AG2 y 1TPX]
CONCLUSIÓN
Las proteínas metamórficas son moléculas versátiles, que han sido seleccionadas en la evolución por ser capaces de alternar sus plegamientos y regular sus funciones. Son moléculas que nos benefician (ya que desempeñan actividades esenciales para nuestro organismo) pero que también pueden derivar en procesos patológicos, lo que da pie a que se abran nuevas vías de investigación. Estas proteínas nos han permitido conocer la manera o el por qué una enfermedad nos perjudica de una manera determinada, algo que facilita poder encontrar un tratamiento a estas enfermedades.
REFERENCIAS
- Algunas proteínas cambian de plegamiento para realizar diferentes funciones. (s. f.). Investigación y Ciencia. https://www.investigacionyciencia.es/noticias/algunas-protenas-cambian-de-plegamiento-para-realizar-diferentes-funciones-19671
- Dishman, A. F. & Volkman, B. F. (2022). Design and discovery of metamorphic proteins. Current Opinion in Structural Biology, 74, 102380. https://doi.org/10.1016/j.sbi.2022.102380
- Eghiaian, F., Grosclaude, J., Lesceu, S., Debey, P., Doublet, B., Tréguer, E., Rezaei, H. & Knossow, M. (2004). Insight into the PrP C → PrP Sc conversion from the structures of antibody-bound ovine prion scrapie-susceptibility variants. Proceedings of the National Academy of Sciences, 101(28), 10254-10259. https://doi.org/10.1073/pnas.0400014101
- Just a moment. . . (s. f.). https://pubs.acs.org/doi/10.1021/acschembio.5b00542
- Lymphotactin/XCL1 Mouse ELISA Kit – Invitrogen. (s. f.). https://www.thermofisher.com/elisa/product/Lymphotactin-XCL1-Mouse-ELISA-Kit/EMXCL1X5
- Perfil, V. T. M. (s. f.). Proteínas metamórficas, camaleónicas, o mejor metamorfas. http://mgclaros.blogspot.com/2021/05/proteinas-metamorficas-camaleonicas-o.html
- Prion Review: Chapter IV (PrP). (s. f.). http://www.biologia.edu.ar/el_prion/prion4.htm
- Sarnataro, D., Pepe, A. & Zurzolo, C. (2017). Cell Biology of Prion Protein. Progress in Molecular Biology and Translational Science, 57-82. https://doi.org/10.1016/bs.pmbts.2017.06.018
- Vila, J. A. (2020). Metamorphic Proteins in Light of Anfinsen’s Dogma. The Journal of Physical Chemistry Letters, 11(13), 4998-4999. https://doi.org/10.1021/acs.jpclett.0c01414