P53: El camino hacia una división segura
Por Álvaro Benito, UAH
INTRODUCCIÓN:
- Nuestras células funcionan como una orquesta ordenada. Cada una cumple su función y realiza sus correspondientes procesos cuando y como se le ordena, el orden y el equilibrio son pilares que nos permiten vivir.
Tanto es así, que contamos con un numeroso arsenal de procesos que nos permiten mantener el orden en nuestro organismo, actuando contra las células que pretenden romper este orden. Hablamos de células que pierden el control y comienzan a proliferar sin fin, invadiendo espacios y causando toda clase de problemas: es decir, un tumor.
Con el fin de evitar un crecimiento descontrolado, contamos (entre otras medidas) con una proteína que es bien conocida por ser el “guardián de la célula”, cuya activación y actuación va a jugar un papel vital sobre células que han sufrido daños por mutaciones espontáneas, infecciones y radiaciones entre otros.
Esta es traducida por la célula como respuesta al daño en el ADN, y está relacionada con procesos que reparan la secuencia dañada. Si esta reparación no es posible o el daño es muy profundo, también iniciará procesos de muerte celular, que acabarán de una vez por todas con el problema.
ESTRUCTURA:
- El estudio de P53 consiste en una serie de dominios que llevan a cabo diferentes funciones dentro de la propia proteína.
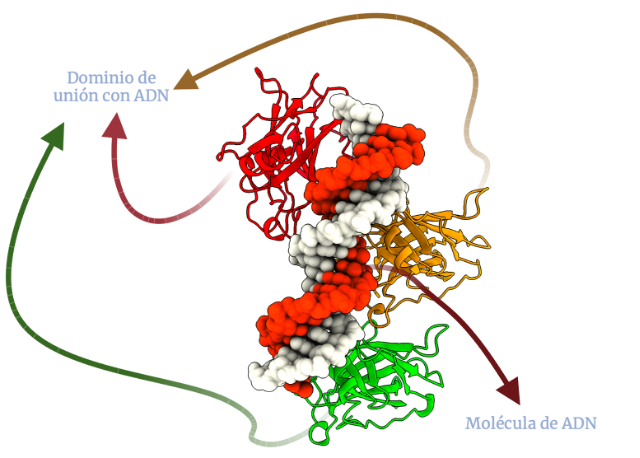
Estamos ante uno de los dominios principales de la proteína (entrada PDB 1tup), un dominio largo (rico en arginina) cuya función es enlazar p53 con la molécula de ADN (1).
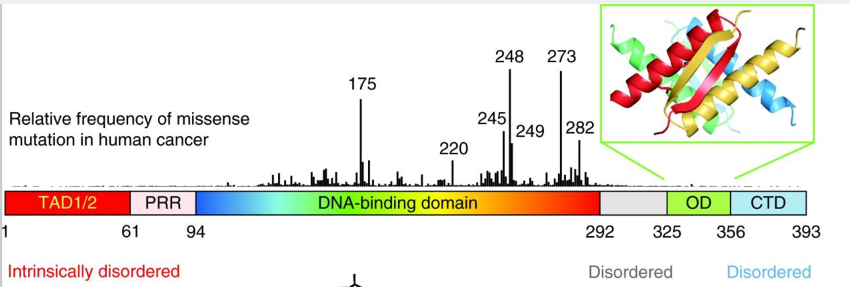
Destaca sobre el resto de los dominios estudiados al ser el origen de la mayoría de las mutaciones que se aprecian en células cancerígenas en relación con cambios aberrantes en p53 (2).
Un paso más allá…
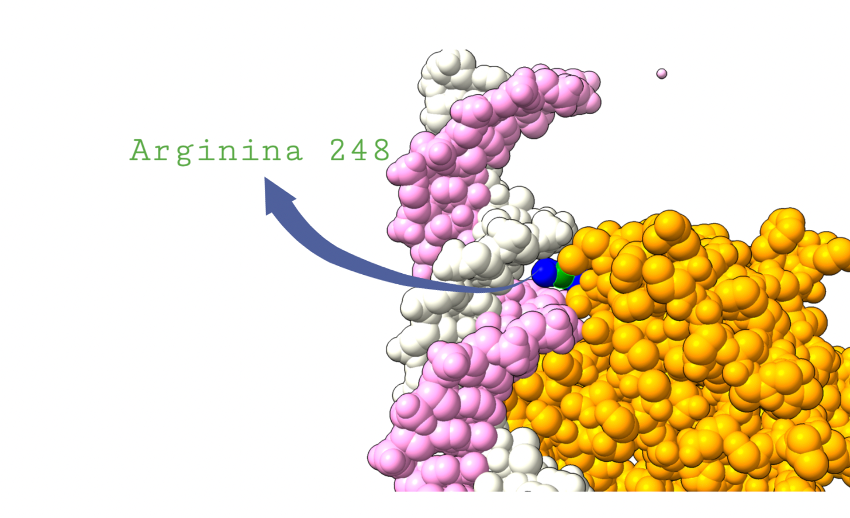
Aquí tenemos un ejemplo de una molécula de potencial mutación que podría alterar el funcionamiento de p53. Vemos como la Arginina 248 se introduce a modo de “serpiente” entre las hebras de ADN. Esto le aporta una fuerte estabilidad que permite la unión de p53 al ADN. Una mutación podría alterar esta funcionalidad, por ejemplo, cambiando el aminoácido que se ubica en este lugar. Esto generaría una pérdida de estabilidad que resultaría en una muy posible alteración funcional (1).
PROCESOS DE ACTUACIÓN:
- P53 es lo que se conoce como un regulador negativo. Se encuentra en bajas concentraciones dentro de la célula, concentraciones que aumentan en situación de estrés celular (inducido por daños en el ADN). Así, p53 sigue una serie de rutas para activarse y regular la actividad celular (3):
- Fosforilación:
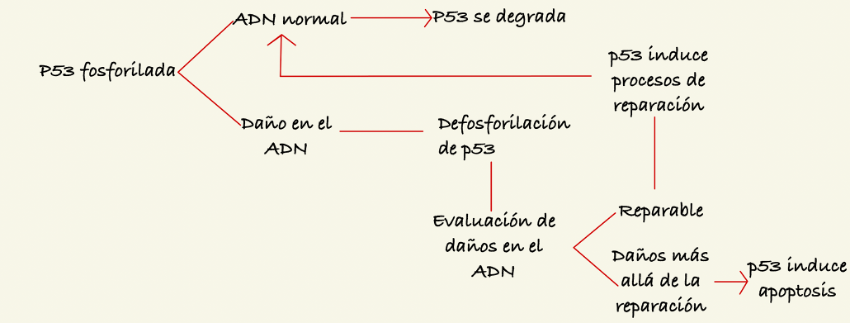
En condiciones normales, p53 se encuentra fosforilada. Esto la vuelve inestable y lleva a su degradación vía proteasoma. Sin embargo, en respuesta al daño celular, se llevan a cabo procesos de defosforilación que estabilizan la estructura de p53 y consiguen su estado de activación. A partir de aquí, p53 se une al ADN y evalúa los daños. Después, induce procesos de reparación del ADN. Si el daño no puede ser reparado, entonces p53 enfoca la apoptosis, mediante el aumento de niveles de pro-apoptóticos.
- Ubiquitinación:
Mdm2 es una de las principales ligasas E3, cuyos altos niveles resultan en la degradación nuclear de p53. Es decir, es un regulador negativo de p53. Sus niveles pueden ser alterados por el ataque de virus: el virus del Herpes (un virus relacionado con la incidencia de ciertos tipos de cáncer).
Curiosamente, p53 induce la síntesis de Mdm2, formando un ciclo que acaba con la degradación de p53 una vez esta ha llevado a cabo su función.
- Acetilación:

La acetilación de p53 consiste en generar cambios conformacionales que modifiquen la conducta de la proteína. Uno de los ejemplos estudiados es la acetilación de p53 por parte de Tip60 (una proteína de la familia MYST HAT). Cuando esta actúa sobre p53, se aumenta su afinidad hacia la expresión de genes pro-apoptóticos. De esta forma, p53 induce su aparición una vez acetilada.
Una peculiaridad del proceso es que, en esta forma, p53 no induce la parada del ciclo celular. Esto es importante, ya que se ha demostrado que en ciertas mutaciones presenten en células cancerosas, la actividad de p53 acetilada por Tip60 no llega a promover pro-apoptóticos, y como tampoco induce la parada del ciclo, p53 pierde capacidades de regulación ante daños y la célula sigue su ciclo a pesar de estar dañada.
PAPEL SANITARIO, UN VISTAZO A LA RELEVANCIA DE LA PROTEÍNA:
- Como ya se ha dicho, p53 juega un papel fundamental en la incidencia del cáncer. De sus mecanismos de actuación se obtiene la capacidad de nuestro organismo de evitar la propagación de células oncogénicas (4):
- Apoptosis: P53 posee la capacidad de llevar a cabo procesos de transducción celular que conducen a la apoptosis de la célula diana. Es decir, resulta en la destrucción de la célula problemática.
- Senescencia: Las células son capaces de entrar en un estado de “arresto” en el que ya no continúan replicándose. Este proceso se establece relación con p53 (5). Las células cuentan con cromosomas, cuyos extremos terminales (más conocidos como telómeros) se van acortando con cada replicación. Este es un proceso relacionado con el envejecimiento, ya que a medida que se vuelven más cortos, la célula pierde capacidad replicativa: hasta que se detiene por completo.
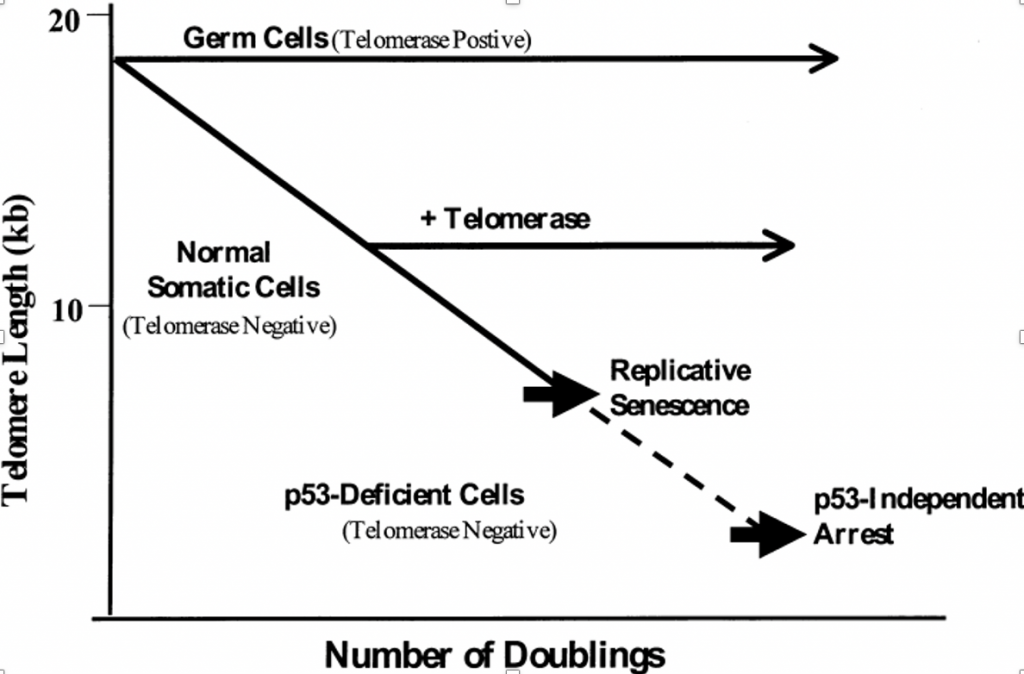
Y es que resulta que p53 es sensible a la longitud de los telómeros e inducirá el arresto celular o senescencia al bajar cierto umbral de longitud. Curiosamente, si estudiamos una célula deficiente de p53, las divisiones celulares continuarán, pero al llegar cierto punto se dejarán de dividir incluso sin la presencia de p53. Esto enlaza a su vez con el mecanismo de las telomerasas que mantienen la longitud de los telómeros a lo largo de las divisiones: que no es objeto de estudio de esta publicación.
La principal importancia que hay que recordar es el hecho de que p53 no solo interviene en procesos tumorales: también está relacionada con procesos de envejecimiento.
Es por ello, que p53 es uno de las proteínas pilar de nuestros procesos de regulación, lo que la hace una diana perfecta para el desarrollo de nuevos tratamientos contra enfermedades como el cáncer en relación con la reactivación de esta proteína en las células cancerígenas (4).
BIBLIOGRAFÍA:
- The Protein Data Bank H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) Nucleic Acids Research, 28: 235-242. Doi: http://dx.doi.org/10.2210/rcsb_pdb/mom_2002_7
- Joerger, A. C., & Fersht, A. R. (2010). The tumor suppressor p53: from structures to drug discovery. Cold Spring Harbor perspectives in biology, 2(6), a000919.
- Liu, Y., Tavana, O., & Gu, W. (2019). p53 modifications: exquisite decorations of the powerful guardian. Journal of molecular cell biology, 11(7), 564-577.
- Zuckerman, V., Wolyniec, K., Sionov, R. V., Haupt, S., & Haupt, Y. (2009). Tumour suppression by p53: the importance of apoptosis and cellular senescence. The Journal of Pathology: A Journal of the Pathological Society of Great Britain and Ireland, 219(1), 3-15.
- Itahana, K., Dimri, G., & Campisi, J. (2001). Regulation of cellular senescence by p53. European journal of biochemistry, 268(10), 2784-2791.
Molecular graphics and analyses performed with UCSF ChimeraX, developed by the Resource for Biocomputing, Visualization, and Informatics at the University of California, San Francisco, with support from National Institutes of Health R01-GM129325 and the Office of Cyber Infrastructure and Computational Biology, National Institute of Allergy and Infectious Diseases.
UCSF ChimeraX: Structure visualization for researchers, educators, and developers. Pettersen EF, Goddard TD, Huang CC, Meng EC, Couch GS, Croll TI, Morris JH, Ferrin TE. Protein Sci.2021 Jan;30(1):70-82.