Proteína 2×1, la Helicasa/Proteasa del Virus de la Hepatitis C
Realizado por Ainhoa Marín, Araceli Lucio y Nerea Mangas, estudiantes de 1º de Biología Sanitaria
1. Introducción
Actualmente, la palabra virus se encuentra en boca de todos, ya puede ser por la reciente pandemia del SARS-COV-2 o por el virus del VIH, que dejó huella en los años 80 con un aumento de casos de SIDA y la muerte de varios artistas reconocidos en todo el mundo. Sin embargo, los virus nos rodean.
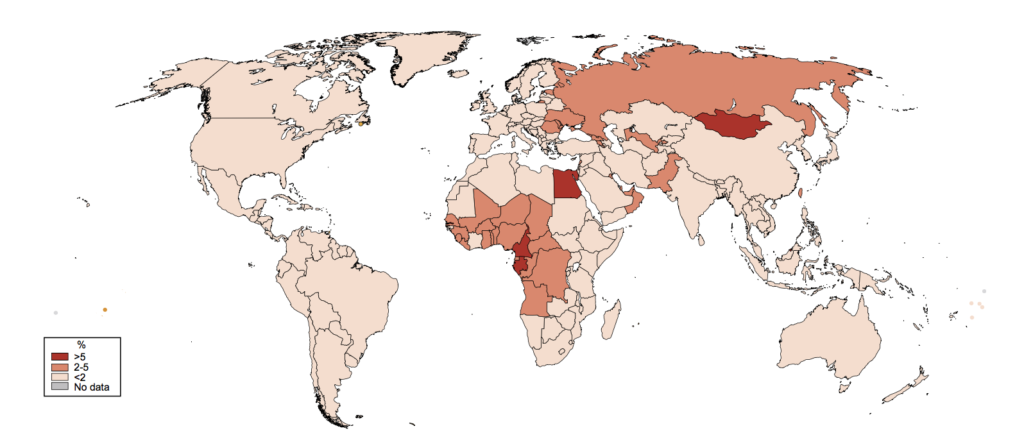
A día de hoy, el virus de la hepatitis C afecta a un 3% de la población mundial, en la que puede producir la aparición de lesiones histológicas hepáticas, además de complicaciones clínicas, por la hipertensión portal y deterioro analítico de la función hepática. Fue descubierto a finales del siglo XX. En esa época había un gran riesgo de contagio debido a las transfusiones de sangre, por lo que su descubrimiento supuso un éxito en el ámbito clínico. Descubriendo el causante de la enfermedad, esta puede ser detectada más fácilmente y con ello desarrollar terapias para combatir el virus. Este hallazgo se produjo gracias a los científicos H. J. Alter, M. Houghton y C. M. Rice, quienes fueron galardonados con el Premio Nobel de Medicina en el año 2020 en reconocimiento a su investigación del HCV (Virus de la Hepatitis C), que ayudó en su diagnóstico y búsqueda de fármacos [1] [18].
El Virus de la Hepatitis C es un virus de ARN. Por ello, se trata de un virus que ataca a los ribosomas y les fuerza a producir una gran cadena polipeptídica viral, la cual debe ser “cortada” en partes funcionales para el virus por una proteasa (lo mismo hacen el virus del VIH o los coronavirus). Esa proteasa va a ser la principal diana para los medicamentos en contra del virus, ya que es esencial para su ciclo funcional. Sin embargo, debemos entender cómo es la estructura viral para comprender su función y, a su vez, conseguir antivirales que actúen contra ella.
2. Formación de polipéptidos
Para conocer las proteínas “pertenecientes” a un virus, como puede ser la helicasa/proteasa de la que hablaremos más adelante, se debe tener claro cómo es el ciclo vital viral, lo cual haremos desde un punto de vista muy genérico.
Como se ha nombrado anteriormente, el HVC se trata de un virus de ARN, es decir, posee un material genético compuesto por una cadena de ácidos ribonucleicos. Este ARN será liberado por el virus tras la unión de este a la célula, en este caso a los hepatocitos, de modo que los ribosomas pertenecientes al hospedador iniciarán la síntesis proteica del virus.
Se sintetizará una poliproteína compuesta por, aproximadamente, tres mil aminoácidos. Esta cadena será procesada y se dividirá en 10 polipéptidos esenciales para el ciclo del virus, dando lugar a polipéptidos estructurales que formarán parte de la cápside o envoltura del virus y, lo que nos concierne, a polipéptidos no estructurales. En estos últimos se encuentra el fragmento NS3-4A-4B-5A-5B, un complejo enzimático relacionado con la replicación del genoma viral. Este fragmento se va a dividir por acción de la proteasa NS3, quien a través de un proceso de autocatálisis forma las proteínas NS3/NS4A, NS4B, NS5A Y NS5B [2].
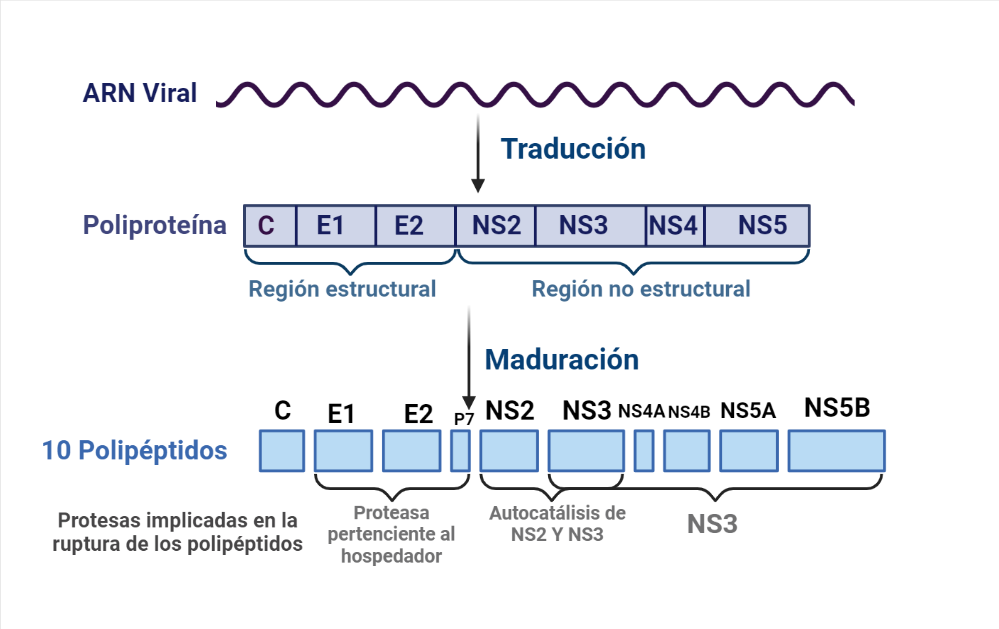
3. Estructura
La proteína NS3/NS4A se trata de una asociación entre las proteínas NS3 y NS4A (que actúa como cofactor), y gracias a ello se alinean los aminoácidos participantes en la acción catalítica de la proteína NS3.
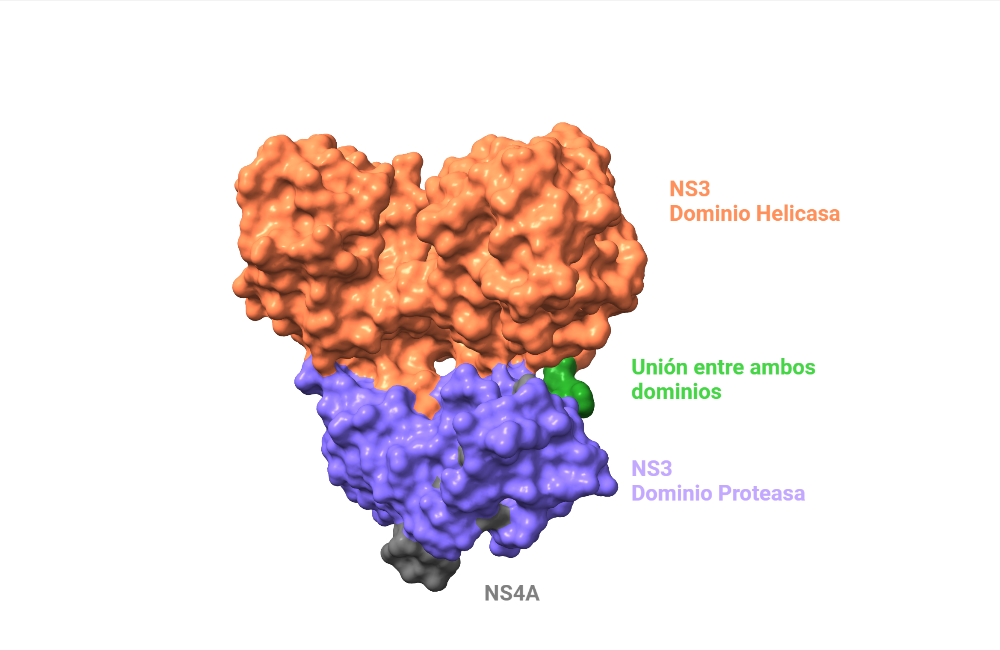
La proteína NS3 está compuesta por 631 aminoácidos con funcionalidad de proteasa y helicasa. Debido a esto, poseerá dos dominios según esas funcionalidades: dominio proteasa y dominio helicasa. Su extremo N-terminal (dominio proteasa) presenta el plegamiento típico de la familia de las quimiotripsinas, es decir, dos regiones compuestas por una estructura de tipo beta-barril que le confiere esa funcionalidad de proteasa por parte del aminoácido de serina. De esta manera la acción catalítica que llevará a cabo es muy parecida a la presente en la quimiotripsina; sin embargo, el plegamiento no es completamente igual, por lo que los inhibidores dirigidos a las quimiotripsinas no funcionan en esta proteína. Además, este dominio proteasa posee un átomo de zinc estructural que estabiliza los bucles procedentes de las láminas beta del dominio [3].
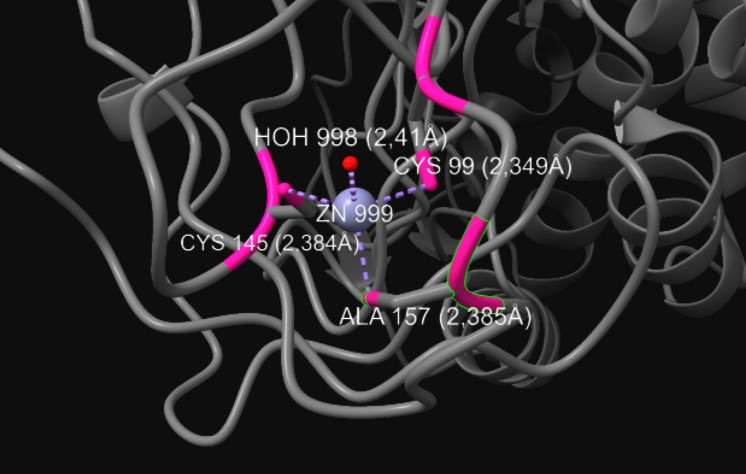
Imagen realizada por Nerea Mangas con ChimeraX a partir de PDB 1cu1
El resto de la proteína comprende el dominio helicasa (extremo C-terminal). Con esa acción es capaz de desenrollar el dúplex de ARN viral durante su replicación. El sitio de unión NTP (nucleósido trifosfato) y de ARN de una sola cadena, así como unas ranuras superficiales, conforman el centro activo de la helicasa. En ese sitio de unión NTP se presenta un grupo fosfato.
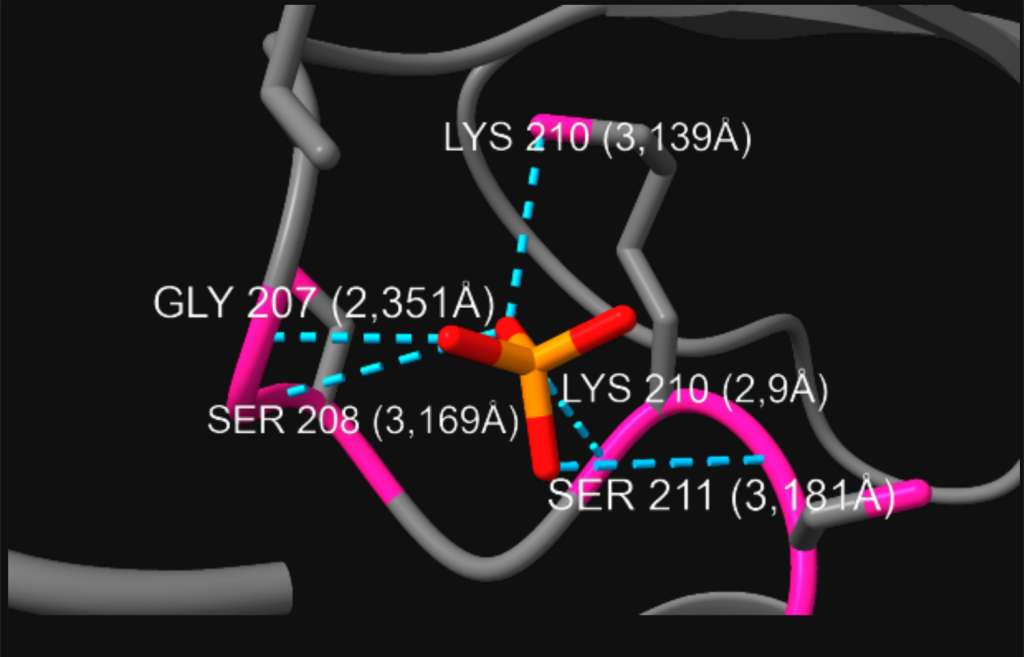
Imagen realizada por Nerea Mangas con ChimeraX a partir de PDB 1cu1
Ambos dominios de la proteína NS3 son unidos covalentemente por una cadena corta y expuesta conformada por los residuos: Pro182, Val183, Phe184, Thr185 y Asp186. A través de estudios se ha podido observar que esta cadena de unión parece flexible y adopta conformaciones ligeramente diferentes en cada copia del complejo proteico que comprende la unidad asimétrica cristalográfica.
El complejo NS3-NS4 contiene 685 residuos, abarcando de esta manera el 23% del genoma del virus y un 35% de la región no estructural, y se trata de una molécula globular con una dimensiones de 65 Å × 65 Å × 75 Å. La cadena posee seis subdominios. Como hemos dicho anteriormente, el dominio proteasa posee dos beta-barril, propio de las proteasas serinas de la familia de las quimotripsinas. En el caso del dominio helicasa posee dos subdominios estructurales beta-alfa-beta, que los relacionan con otro subdominio compuesto por siete hélices alfa y tres cadenas beta de pequeño tamaño. Finalmente, el último subdominio es conocido como helicasa alfa-helicoidal [4].
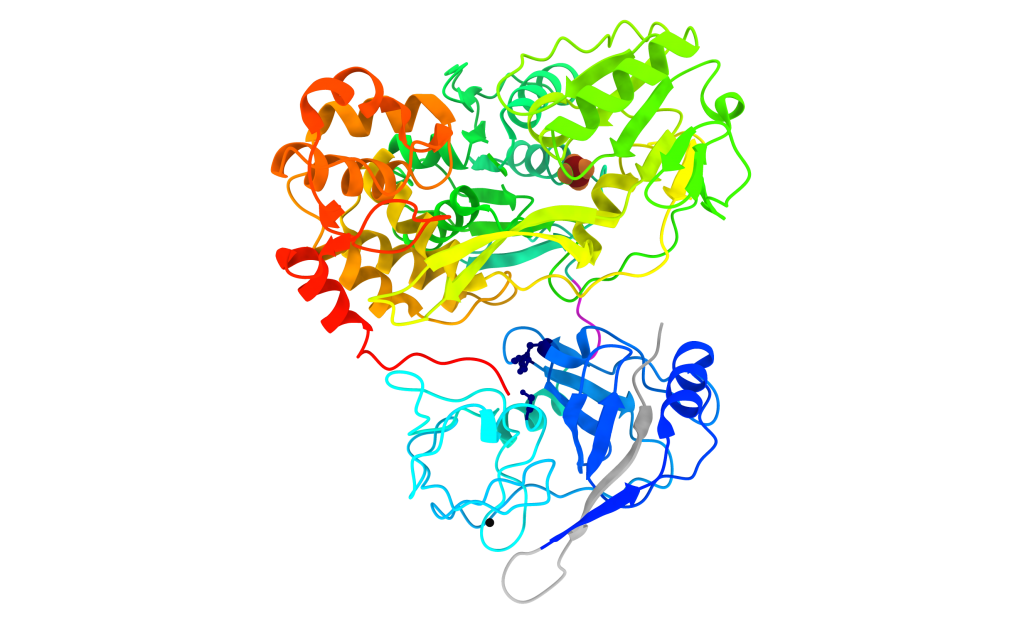
Imagen realizada por Nerea Mangas con ChimeraX a partir de PDB 1cu1
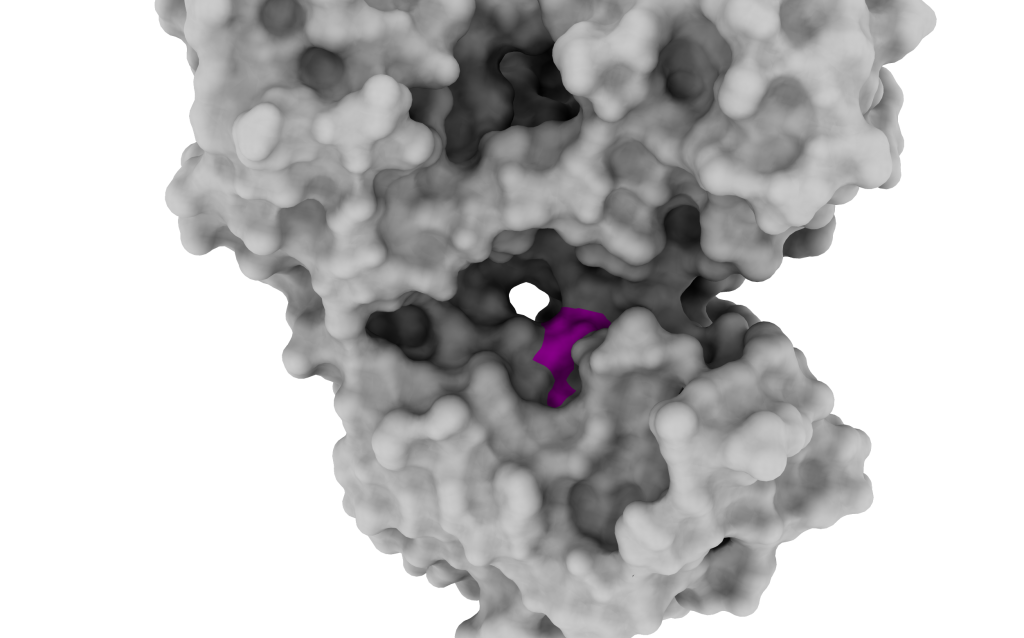
Como se he mencionado, la formación del complejo NS3-NS4A constituye el proceso que alinea el centro catalítico, por lo que esa unión forma parte del sexto subdominio. Este centro catalítico va a ser el lugar donde la proteína realizará su función de proteasa, con acción de los aminoácidos presentes en él y el cofactor que es la proteína NS4A.
En este sexto subdominio la proteína NS4A, conformada por 54 residuos y con una estructura de tipo beta-plagada, se implica en la activación por la terminación del beta-barril de la proteasa del N-terminal. De esta manera ordena los residuos de la proteína NS3 y coloca la triada catalítica del dominio proteasa serina. Sin embrago, se sabe que solo se requiere la parte central de la proteína NS4A (conformada por 14 residuos) para esta activación, pero se desconoce la funcionalidad del C-terminal de dicha proteína [4]. Aun así, el N-terminal, hidrofóbico, va a localizar al complejo proteico en la membrana del retículo endoplasmático de la célula huésped, localización donde se sintetizará la proteína viral [5].
El centro activo de la proteasa serina se conoce como triada catalítica y se compone de tres residuos: His-57, Asp-81 y Ser-139. La serina será el aminoácido que ataque de forma nucleófila al sustrato, la histidina se unirá al portón hidroxílico de la serina aumentado así su carácter nucleófilo (el de la serina), mientras que el aspartato estabilizará la carga de la histidina que se encontrará doblemente protonada [2].
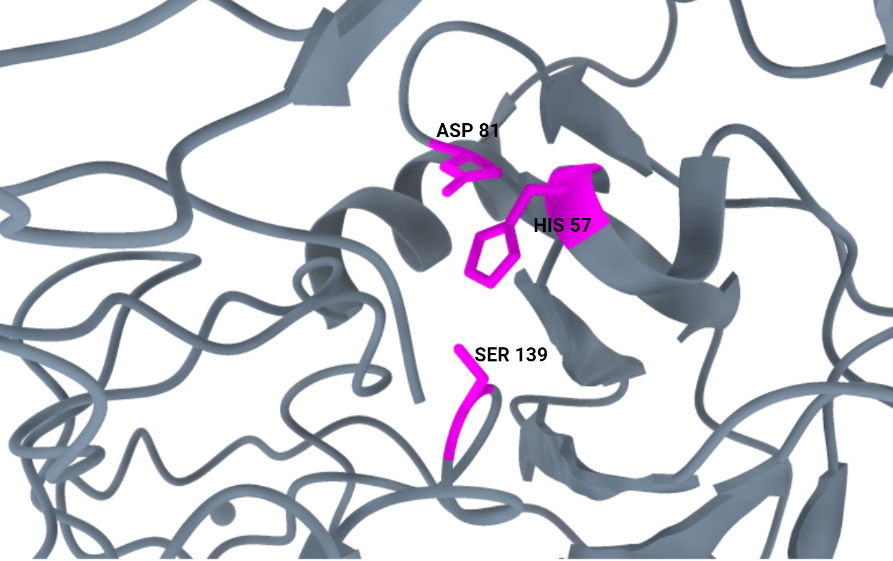
4. Función biológica
El virus de la hepatitis C está formado por varias proteínas, las cuales podemos agrupar en dos clases según sean estructurales o no.
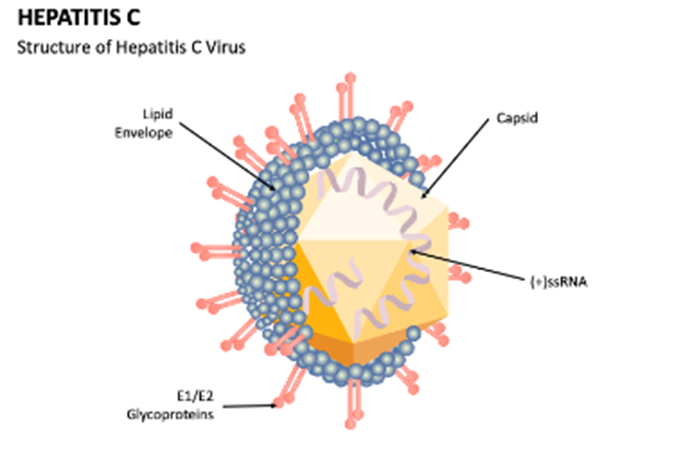
Entre las proteínas estructurales podemos encontrar las proteínas que conforman la cápside: E1, E2 y P7. Las proteínas estructurales realizan, como su nombre indica, funciones principalmente estructurales; forman la cápside del virus, interaccionan con receptores celulares de la célula que van a infectar y evaden el sistema inmunológico de la célula infectada, entre otras. Destaca la P7, la cual es esencial en la infección de nuevas células. Aunque no se tiene clara su funcionalidad completa y sus mecanismos de acción, se trata de un péptido y es clasificado como una viroporina (viroporin), es decir, una proteína hidrófoba que se encarga de realizar poros hidrofóbicos en la membrana del hospedador, llevando a la fusión entre le virus y el hospedador, y con ello a la entrada del virus en la célula, además de la secreción de viriones (partícula vírica completa) [2][15][16].
Entre las proteínas no estructurales podemos encontrar las proteínas NS2, NS3, NS4A, NS4B, NS5A y NS5B. Las proteínas no estructurales tienen distintas funciones, entre las que destacan la inhibición de la apoptosis al inhibir proteínas proapoptóticas de la célula infectada, replicación de ARN vírico para crear más virus y continuar infectando otras células, y síntesis de proteínas que conformarán nuevos virus [5].
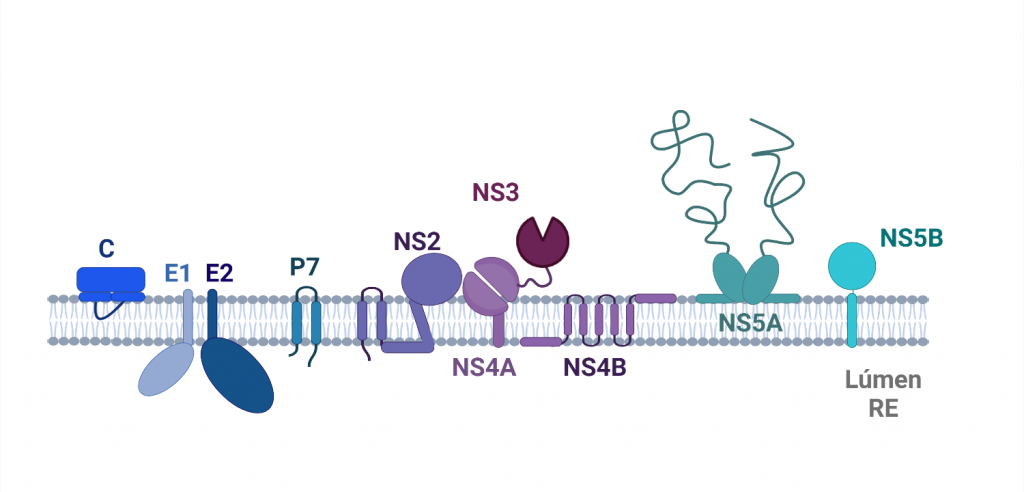
Como hemos ido viendo a lo largo de este artículo, nos centramos en las proteínas NS3 y NS4A por su importancia en la síntesis de nuevos virus. La proteína NS3 es una proteasa, helicasa y NTPasa. Posee dos dominios independientes; el dominio proteasa y el dominio helicasa/NTPasa. Aunque sean independientes, un dominio modula la actividad del otro.
A su vez, un análisis mutacional ha demostrado la importancia de la zona de unión de ambos dominios para la replicación y la inefectividad de la célula diana, pero no es necesaria para la acción helicasa y proteasa. Esta zona de unión entre los dominios modula la conformación de la proteína (alosterismo) e interacciones con otras moléculas [8].
Acción del dominio proteasa
En su dominio de proteasa, la NS3 separa dominios rompiendo los enlaces peptídicos de las proteínas para posteriormente formar proteínas no estructurales. Esta acción la podemos apreciar claramente durante la maduración de la cadena polipeptídica, nombrada anteriormente.
También se formará un heterodúplex de ARN vírico en la replicación. Un heterodúplex es aquel ADN o ARN de doble hélice que se forma a partir de recombinación homóloga entre una cadena de un cromosoma parental y una cadena de su cromosoma homólogo [7]. Para ello, necesita un cofactor, que en este caso es la proteína NS4A [2] [6].
Acción del dominio helicasa
En su dominio helicasa/NTPasa, la NS3 se encarga principalmente de desenrollar ARN y ADN durante la replicación. Además, este dominio posee cuatro motivos para su actividad NTPasa; un motivo que se encarga de unir NTP a la nueva cadena, otro que une a los nucleótidos trifosfato átomos de magnesio para hidrolizarlos, un motivo que hidroliza NTP cuando se desenrolla el ARN y un último motivo del cual se desconoce su función [2] [5] [6] [8].
Con todo ello vemos que la acción doble de la proteína NS3/NS4A del virus de la Hepatitis C es muy importante para todo el ciclo de vida del virus, con intervención tanto en la replicación del material genético como en la formación de proteínas virales. A causa de ello será un gran target para antivirales.
5. Implicaciones biomédicas
A día de hoy, como se ha mencionado, la comprensión del ciclo del virus de la hepatitis C (VHC) ha permitido la identificación de dianas potenciales en determinados puntos de su ciclo vital. De este modo, se pueden detectar e inhibir posibles dianas terapéuticas a nivel de la entrada del virus, de su replicación y de la formación de partículas infecciosas, actuando sobre ellas de modo que interrumpa directamente la reproducción del VHC. Como se ha dicho al principio, la proteasa NS3/4A del VHC es uno de los objetivos más obvios (además de la polimerasa NS5B) por su papel fundamental en el ciclo de vida del virus, por lo que es reconocido su uso como proteína diana de medicamentos en el tratamiento contra la enfermedad, permitiendo el desarrollo de tratamientos más o menos eficaces que contribuyen a la disminución de la carga viral y a disminuir o incluso revertir el daño hepático causado por el VHC [9] [10].
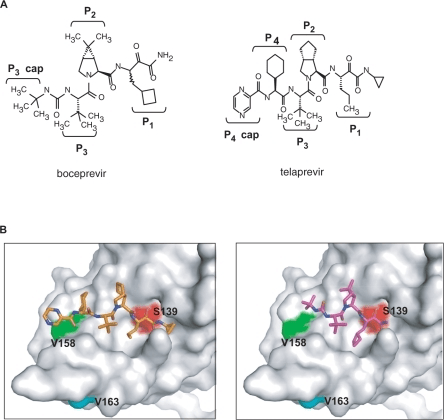
Así, tras años de tratamientos y terapias no tan efectivas, en el año 2011 se produjo un notable avance con la aparición de la primera generación de inhibidores de la proteasa del VHC: Boceprevir (BOC) y Telaprevir (TVR) que, junto a los inhibidores de la polimerasa análogos de nucleósidos o no nucleósidos, constituyen la primera generación de AAD (agentes antivirales de acción directa). Estos fármacos pueden llevar a un aumento considerable de efectos secundarios y su necesidad de ser administrados junto a PegIFN y RBV (interferón pegilado y ribavirina) en la conocida como “triple terapia” puede considerarse un inconveniente, no obstante, esta combinación aumenta considerablemente las posibilidades de éxito con respecto a la terapia dual tradicional (IFN + Ribavirina). Los efectos beneficiosos atribuidos al tratamiento de peginterferón con ribavirina son: inhibición de la replicación y traducción de proteínas del VHC, supresión de la proliferación celular, potenciación de la función fagocítica de los macrófagos, potenciación de la citotoxicidad linfocítica contra células infectadas, inhibición de la producción de colágeno y potenciación de su degradación, disminución de la carga viral del VHC, inhibición de la progresión del daño histológico, así como una probable disminución de las complicaciones hepáticas [9] [12] [14].
Foto realizada por: Andrew Karpenko, University of Washington
Acción de Boceprevir
El modo de acción de Boceprevir (autorizado para el tratamiento de infección crónica por VHC de genotipo 1) consiste en la unión selectiva, mediante un enlace covalente reversible, al sitio activo de la serín-proteasa NS3/A4 (Ser139) a través de un grupo funcional alfa cetoamida. Actuando como falso sustrato para la proteasa, emula una cadena peptídica que bloquea el centro activo, inhibiendo específicamente la proteasa NS3 implicada en el fraccionamiento de la poliproteína que es codificada por el ARN viral y, por consiguiente, impidiendo la replicación viral en las células huésped infectadas por el VHC. La resistencia del VHC al Boceprevir se asocia a mutaciones únicas localizadas en diferentes posiciones, siendo el grado de capacidad inhibitoria sobre la proteasa NS3 diferente para los diversos genotipos de VHC, y no igual de efectivo en todos los fármacos [11] [13].
Actualmente, están apareciendo nuevas generaciones de inhibidores de la proteasa del VHC, como es el Simeprevir, con el objetivo de alcanzar la RVS (Respuesta Viral Sostenida), factor que se tiene en cuenta a la hora de evaluar la eficacia del tratamiento y que se refiere a la ausencia de viremia 12 semanas después de haber concluido el tratamiento. Si nos fijamos en la terminación de los nombres genéricos de los fármacos, los inhibidores de la proteasa incluyen la terminación “previr”, destacando otros ejemplos como el Faldaprevir, un potente inhibidor de la proteasa HCV NS3/4A, así como el Vaniprevir [9] [12].
A su vez, ha habido un desarrollo de diferentes estudios para visualizar los distintos tratamientos para combatir el virus de Hepatitis C. Por ejemplo, a través del uso de bacterias E.Coli se estudia la eficiencia de la proteína NS3 en el procesamiento de la proteína sustrato. Esta bacterias se encuentran transformadas para expresar tanto la proteasa como la poliproteína sustrato. Esta técnica permite manipular la secuencia de la proteína sustrato y localizar la presencia de regiones donde la proteína viral (NS3) realiza su corte, con el objetivo de analizar las condiciones que requiere la enzima para poder reconocerla, la proteína sustrato, y procesarla. Con ello se determina esa eficiencia del proceso catalítico en presencia de compuestos antivirales.
Por otro lado, se han utilizado células eucariotas a las que se administra el virus Vaccina modificado para que exprese la proteína objeto de estudio. Sin embargo, este mecanismo ha sido sustituido por el sistema de replicón-ELISA, que genera una proteína que expresa en una sola cadena la región proteasa de la NS3 y varios dominios de corte de la poliproteína viral (NS3- NS4A-NS4B-NS5), representando una situación que puede darse in vivo en el ciclo del virus, durante el procesamiento de la poliproteína. Con este estudio se pueden determinar compuestos inhibidores de NS3.
| ANTIVIRALES | ACCIÓN |
| Ribavirina | Análogo a la guanosina (frena la síntesis de ARN viral) |
| Interferón Tipo I Peginterferón alfa2a Peginterferón alfa2b |
Citoquina |
| Alisporivir (Experimental) | Inhibidor de la Ciclofilina |
| Boceprevir Telaprevir Glecaprevir Grazoprevir Peritaprevir Simeprevir Faldaprevir Vaniprevir |
Inhibidor de la Proteasa NS3/NS4A |
| Daclatasvir Elbasvir Ombitasvir Pibrentasvir Elbasvir Velpatasvir Radalbuvir (Experimental) Sofosbuvir |
Inhibidor de la NS5A (polimerasa dependiente de ARN) |
A pesar de todo esto, se debe tener claro que el desarrollo de nuevos tratamientos se ve perjudicado por la alta variabilidad genética que caracteriza al virus de la Hepatitis C, y que recientemente ha aumentado por el hallazgo de diferencias en el dominio de la proteasa de la proteína NS3, la cual, supuestamente, era considerada invariable. Este hecho marca un punto clave para la aparición de partículas víricas genéticamente distintas capaces de no atender al efecto del inhibidor y con ello generar resistencia al medicamento [5].
Todos estos conocimientos nos muestran la complejidad de la proteína vírica en su totalidad, al mismo tiempo que nos pueden derivar al descubrimiento de nuevas alternativas para el desarrollo de antivirales contra la infección del VHC.
Antivirales dirigidos al hospedador
Con la aparición de estas resistencias a antivirales por la alta capacidad de mutación genómica, se limita la acción de los anteriormente mencionados AADs (antivirales de acción directa). Por ello se intenta generar alternativas cuyo objetivo sean partículas pertenecientes al hospedador cruciales parar el ciclo del virus.
Entre las ventajas que pueden tener estos antivirales dirigidos al hospedador (host-targeting antivirals (HTA) en inglés) frente a los AADs se encuentra la generación de una mayor barrera genética frente a la resistencia y actividad antiviral pangenotípica, es decir, que funciona en todas las variaciones del virus.
Los HTAs son menos específicos que los AADs, sin embargo, esa es realmente una de sus ventajas. Con el estudio de estos medicamentos se puede abrir un nuevo espectro de antivirales, ya que al ser su target proteínas del hospedador se puede estudiar para la replicación de otros tipos de virus, además de poder generarse a muy bajo coste, otra ventaja añadida frente a los AADs.
Con la focalización de la síntesis de lípidos y el metabolismo de la célula hospedadora se podría inhibir una replicación viral como puede ser en el caso del HCV o incluso en el virus del Dengue. No obstante, independientemente de cuan efectivos sean la moléculas inhibidoras hacia la replicación del virus, no se va a proteger a un hepatocito no infectado de infectarse. La prevención de que el virus entre en la célula es, por otro lado, un gran objetivo. Con el estudio del mecanismo de la entrada del virus de la Hepatitis C en las células, a lo largo de los años se ha podido ver un conjunto de receptores y cofactores que resultarían grandes targets.
El objetivo, la vacuna
Pese a esto, con la alta eficiencia de los AADs, cada vez es menor el interés en generar nuevos tratamientos contra el virus. Con el fin de erradicar la enfermedad está claro que la sola acción de estos medicamentos no es suficiente, además de que son de un alto coste que origina que en los países con una baja economía la incidencia sea mucho mayor. El uso de estos tratamientos puede derivar en la aparición de variantes resistentes y por consecuente en una reinfección, que en el caso de ser asintomática podría producir un mayor contagio.
Es indudable, tras conocer la historia de la medicina, que el virus de la hepatitis C requiere de una vacuna para alcanzar su erradicación. Los diferentes estudios efectuados en relación al virus han ido derivando en diferentes estrategias para la producción de una vacuna, sin embargo, sin la iniciativa que aminora la idea de que los fármacos constituyen en sí una “cura” contra el VHC, no se eliminará por completo su amenza [15].
6. Referencias
[1] Goodsell, D. S. (2020). Hepatitis C Virus Protease/Helicase. RCSB Protein Data Bank. https://doi.org/10.2210/rcsb_pdb/mom_2020_12
[2] Martínez González, J. Á. (2013). ESTUDIO QM/MM DEL MECANISMO Y CINÉTICA DE LAS REACCIONES DE LA PROTEASA NS3 DEL VIRUS DE LA HEPATITIS C CON SUS SUSTRATOS PRINCIPALES [Doctoral]. Universidad de La Rioja.
[3] Kim, J. L., Morgenstern, K. A., Lin, C., Fox, T., Dwyer, M. D., Landro, J. A., Chambers, S. P., Markland, W., Lepre, C. A., O’malley, E. T., Caron, P. R., & Thomson, J. A. (1996). Crystal Structure of the Hepatitis C Virus NS3 Protease Domain Complexed with a Synthetic NS4A Cofactor Peptide. In Cell (Vol. 87). Miller and Purcell.
[4] Yao, N., Reichert, P., Taremi, S., Prosise, W. W., & Weber, P. C. (1999). Molecular views of viral polyprotein processing revealed by the crystal structure of the hepatitis C virus bifunctional protease-helicase.
[5] Bermúdez AAD. (2008). Función de la proteasa NS3 del virus de la Hepatitis C. Rev Educ Bioquímica. 27(3):79-84.
[6] Dueñas-Carrera S, Acosta-Rivero N, Morales-Grillo J, et al. (2018). Molecular biology of hepatitis C virus. Med Int Mex. 34(3):435-442.
[7] Alquicira, J. ADN heterodúplex. (2016). Conagasi. https://conagasi.org/diccionario/adn-heteroduplex/
[8] McGivern, D. R., Masaki, T., Lovell, W., Hamlett, C., Saalau-Bethell, S., & Graham, B. (2015). Protease Inhibitors Block Multiple Functions of the NS3/4A Protease-Helicase during the Hepatitis C Virus Life Cycle. Journal of Virology, 89(10), 5362–5370. https://doi.org/10.1128/jvi.03188-14
[9] Jiménez Galán, R. et al. Nuevos fármacos en el abordaje terapéutico de la hepatitis C. Farm Hosp. [online]. 2014, vol.38, n.3 [citado 2023-01-21], pp.231-247.
[10] Asociación Catalana de Pacientes Hepáticos (ASSCAT) (Última actualización: 08/06/2018; consultado 1/2023), Fármacos disponibles para el tratamiento de la Hepatitis C.
[11] Ubago Pérez R, Castillo Muñoz MA, Navarro Caballero JA, Márquez Peláez S. Inhibidores de la proteasa (boceprevir y telaprevir) en el tratamiento de pacientes monoinfectados por el VHC: eficacia, seguridad y eficiencia comparada [Internet]. Sevilla: Agencia de Evaluación de Tecnologías Sanitarias, 2012. Informe 1/2012.
[12] Chiquete AE, Sánchez OLV, Panduro CA. Virus de la Hepatitis C. Inv Salud. 2005;7(Suppl: 1):19-25.
[13] Consejo General de Colegios Oficiales de Farmacéuticos (consultado 1/2023), BOCEPREVIR VICTRELIS (MerckSharpDohme)
[14] Grupo de Infecciosas SoMaMFYC, Hepatitis C: comparación Telaprevir y Boceprevir; Jaime Marin (6 de febrero 2013)
[15] Alazard-Dany N, Denolly S, Boson B, Cosset F-L. Overview of HCV Life Cycle with a Special Focus on Current and Possible Future Antiviral Targets. Viruses. 2019; 11(1):30. https://doi.org/10.3390/v11010030
[16]Nieva, J. L., Madan, V., & Carrasco, L. (2012). Viroporins: Structure and biological functions. In Nature Reviews Microbiology (Vol. 10, Issue 8, pp. 563–574). https://doi.org/10.1038/nrmicro2820
[17] ViralZone: a knowledge resource to understand virus diversity. Hulo C, de Castro E, Masson P, Bougueleret L, Bairoch A, Xenarios I, Le Mercier P. Nucleic Acids Res. 2011 Jan;39(Database issue):D576-82.
[18] Aurora Casanova Rituerto, Teresa Casanovas Taltavull. Hepatitis por el virus de la Hepatitis C (consultado 1/2023). https://www.seimc.org/contenidos/ccs/revisionestematicas/serologia/vhc.pdf
PDB-101: Educational resources supporting molecular explorations through biology and medicine. Christine Zardecki, Shuchismita Dutta, David S. Goodsell, Robert Lowe, Maria Voigt, Stephen K. Burley. (2022) Protein Science31: 129-140 doi:10.1002/pro.4200
UCSF ChimeraX: Structure visualization for researchers, educators, and developers. Pettersen EF, Goddard TD, Huang CC, Meng EC, Couch GS, Croll TI, Morris JH, Ferrin TE. Protein Sci. 2021 Jan;30(1):70-82