El error de Pauling y la carrera por la estructura del ADN
C. Menor-Salván. Ver. 2.5. Abril 2023
El descubrimiento de la estructura del ADN fue uno de los logros científicos destacados del siglo XX. En él participaron además de los conocidos Watson y Crick y Rosalind Franklin, una serie de científicos relevantes, cuyo nombre apenas se recuerda; sin sus contribuciones, no podemos entender la historia completa. Esta aventura nos enseña además que, en la ciencia, aunque se hagan famosos los «goleadores», el conocimiento se construye de modo colectivo y los errores pueden ser tan importantes como los aciertos. Delicioso es el fruto que surge tras el amargor del error y la ignorancia, y de ellos es desde donde se construye la ciencia. Además, los aspectos mundanos pueden ser tan relevantes como los técnicos.
El ADN se descubrió en el siglo XIX, pero se tardó más de medio siglo en revelar su estructura
No hay que confundir el descubrimiento de la estructura del ADN, con el descubrimiento del ADN en sí. Se atribuye el descubrimiento del ADN al químico suizo Friedrich Miescher, entre 1860 y 1874, siendo Phoebus Levene quien dio, en 1909, su descripción química precisa. Miescher propuso, en 1874, que, de alguna manera la «nucleína» (nombre que dió al ADN) era la «causa específica de la fertilización».
Levene acuñó el término ‘ácido nucleico’. Propuso la ‘teoría del tetranucleótido’, sugiriendo que el ADN estaba compuesto por cuatro bases, un azúcar y fosfato. Sin embargo, en aquel momento, aún no existía la tecnología necesaria para entender la arquitectura de la molécula.

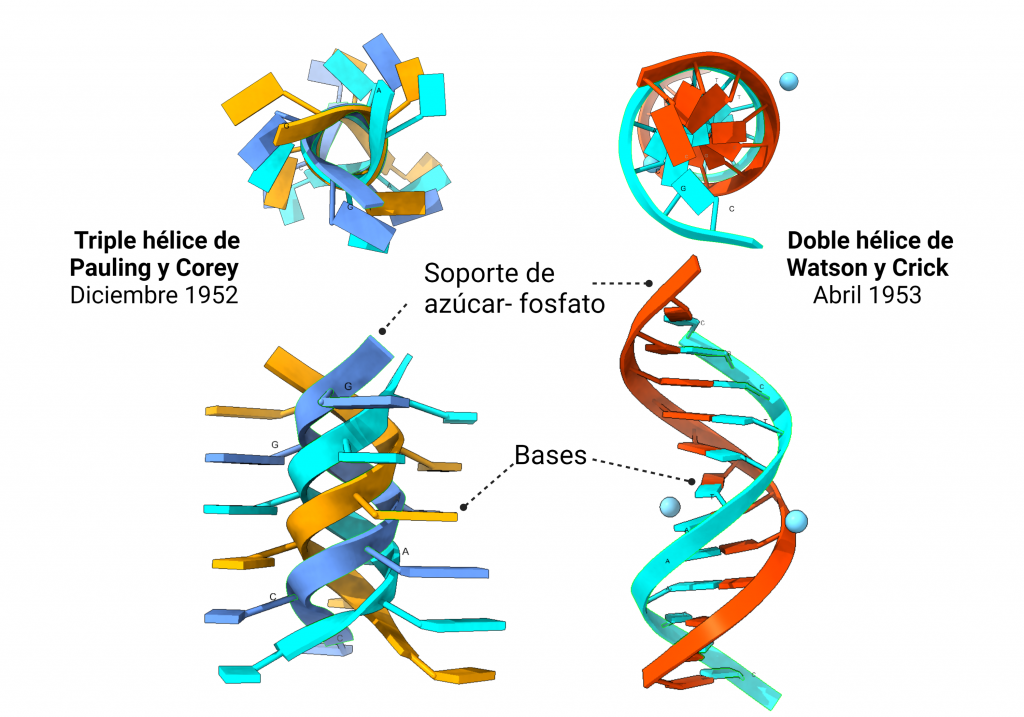
Pasó casi medio siglo hasta que se determinó su estructura. Una vez disponible el arsenal técnico adecuado, no había nadie mejor para lograrlo que el genial químico estadounidense Linus Pauling (1901-1994), quien estuvo a las puertas de conseguirlo antes de que Watson, Crick, Wilkins y Franklin publicaran su famoso artículo triple de abril de 1953.
Pauling fue uno de los científicos más relevantes del siglo XX. Recibió el premio Nobel en Química en 1954 por su contribución al conocimiento de los enlaces químicos. Para esa fecha había realizado tantos descubrimientos importantes en numerosos campos de la Química y la Biología, que, cuando le llamaron para comunicarle la concesión del premio, pidió a su interlocutor que le leyese la comunicación, pues no tenía claro por cuál de sus trabajos recibía el premio.
Pauling había resuelto otro gran problema: la estructura de las proteínas. Con Robert Corey y Herman Branson, publicaron en 1951 las estructuras secundarias que ahora ilustran los libros de texto. Este importante trabajo era ya suficiente para que tuviera fama imperecedera en el mundo de la Ciencia, pero Pauling era ambicioso y estaba obsesionado con resolver todas las estructuras de macromoléculas biológicas. Así, era el favorito en la carrera por el ADN. Él mismo estaba convencido de ello.
Astbury, Bell y la triple hélice de Pauling
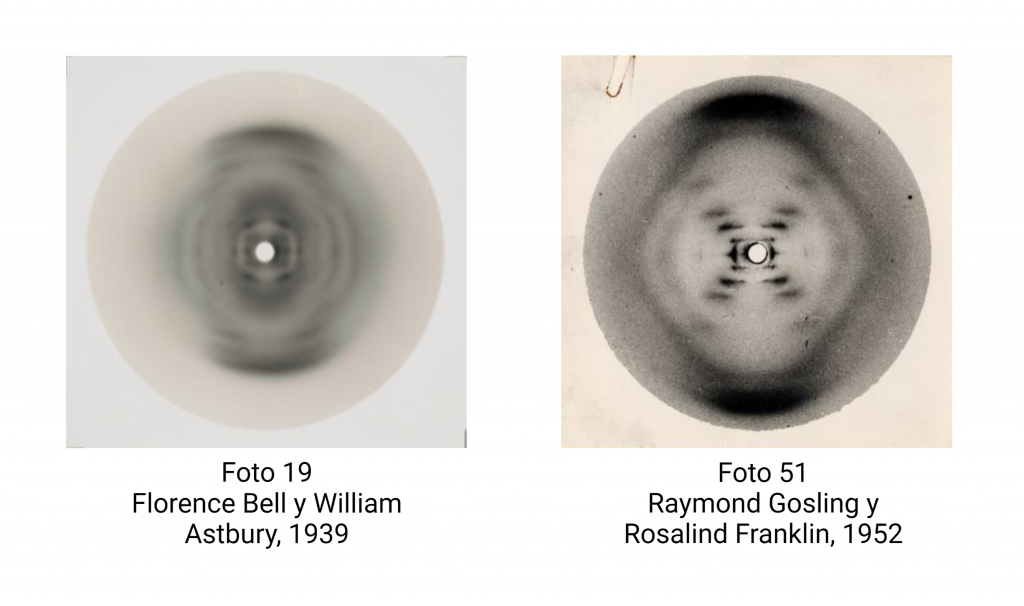
Una brillante química británica, Florence Bell, presentó, en 1939, su tesis sobre la estructura del ADN y las proteínas, bajo la dirección de William Astbury. En ella, Bell y Astbury presentaron, por primera vez en la Historia, imágenes de difracción de rayos X de ADN. Esta compleja técnica, que se aplica en campos desde la Bioquímica Estructural hasta la Mineralogía, es esencial para resolver las estructuras macromoleculares así como las estructuras de los sólidos cristalinos. Y ahí está el problema: EL ADN es muy difícil de cristalizar y tiene una peculiaridad clave, que descubrió Rosalind Franklin: puede cambiar de forma.

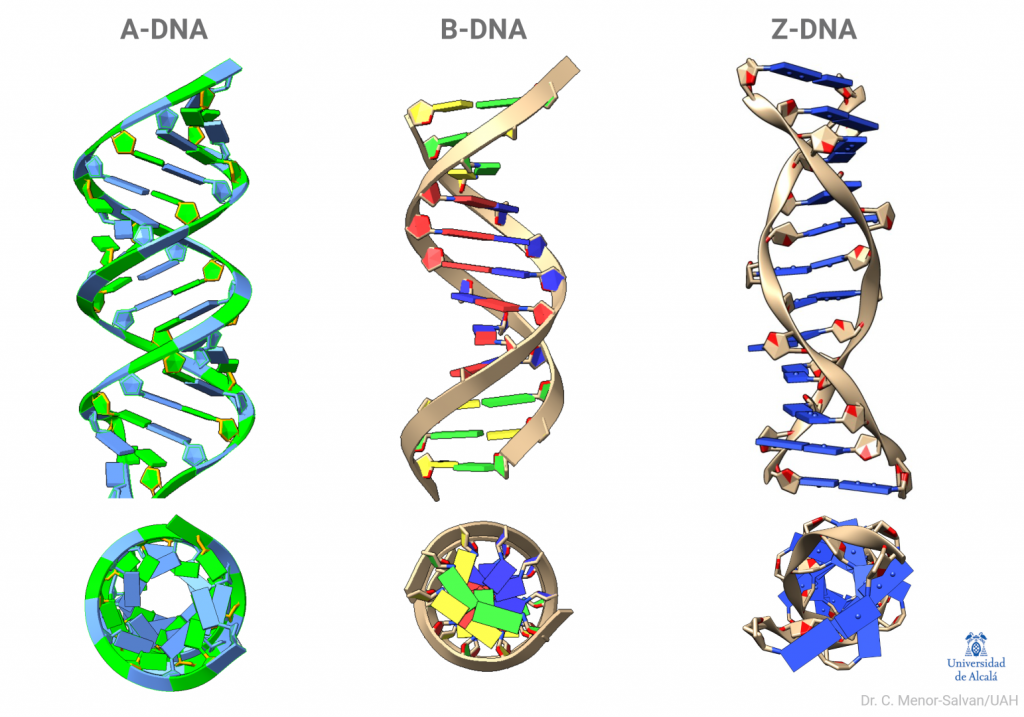
Bell logró obtener un esbozo de la estructura del ADN, formado por largas fibras con las bases colocadas en paralelo y unidas, a modo de cuentas de collar, por el fosfato; a partir de ello, no pudo avanzar más. Su tesis relata los intentos para obtener una muestra de ADN lo suficientemente cristalina, que terminaban en imágenes de difracción borrosas. El ADN se resistía a revelar su estructura. Había algo que Bell ignoraba: el ADN no tiene una única forma. Al preparar el ADN puro, se formaba una mezcla de lo que ahora conocemos como ADN B y ADN A, que hacían muy difícil interpretar los datos. Además, todavía tenía que mejorar la tecnología para la obtención de éstos.
Fue Rosalind Franklin, 15 años después, quien logró lo que Bell no pudo: obtener una forma única de ADN cristalino, en su forma canónica ADN B, con el que hizo posible la famosa foto 51, que aclaró definitivamente la estructura.
Aun así, Bell proporcionó datos importantes, gracias a una imagen particularmente clara, la imagen 19, tales como la distancia entre las bases y que los nucleótidos formaban largas cadenas.
Pauling estudió los resultados de Bell y Astbury, tomando como hipótesis que las bases del ADN se orientan al exterior, para enlazarse con otras moléculas y que los fosfatos se organizan en un patrón similar al de algunos minerales. También tuvo en cuenta otro dato fundamental: el biofísico Robley Williams había logrado, en 1952, obtener una imagen en 3D de una molécula de ADN con un microscopio electrónico. Pauling observó que ese pequeño cilindro podría ser una trenza helicoidal.
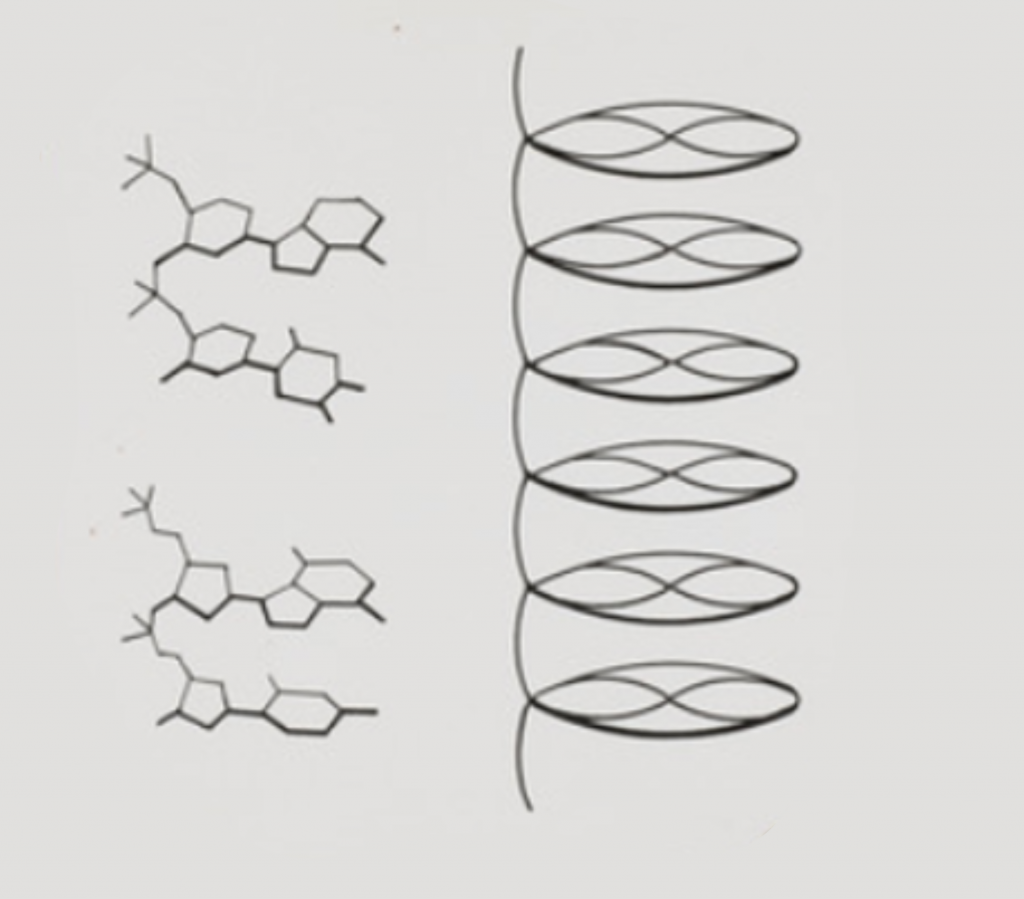
Con estas ideas, propuso un modelo en triple hélice, en un artículo enviado, no sin prisas, el 31 de diciembre de 1952. En él, publicado en febrero de 1953, Pauling sugiere que su modelo es todavía algo preliminar y requería refinado. Su colaborador Corey le avisó de que había problemas, como que el modelo no encajaba del todo bien, no se podían incluir iones de sodio, a pesar de que el ADN formaba una sal sódica, y estaba el problema de la repulsión de las cargas del fosfato; Pauling reconoció que su modelo estaba algo «apretado». Pero la prisa estaba justificada: sabía que, en Gran Bretaña, Maurice Wilkins y su equipo estaban obteniendo imágenes de difracción del ADN y que unos entonces desconocidos James Watson y Francis Crick estaban trabajando en un modelo de ADN.
Sin embargo, el modelo de Pauling resultó ser erróneo.
Pares de bases y la precaución de Wilkins
El modelo en triple hélice encajaba mas o menos bien con los datos de difracción de rayos X de los que disponía Pauling. Es más, el primer modelo de ADN que idearon Watson y Crick, en 1951, era una triple hélice similar a la de Pauling. La diferencia es que ellos contaron con Rosalind Franklin. Cuando mostraron a Franklin su modelo, ella les dio razones convincentes por las que el modelo no era válido. Franklin ya había observado la importancia del agua y de los iones en la estructura (ella estaba trabajando con la sal sódica del ADN) y de cómo debían distribuirse los fosfatos. Ellos la escucharon y volvieron a los cálculos de un nuevo modelo. En ese momento, Franklin y Wilkins, aunque no estaban a favor de determinar un modelo aún, también pensaban que se trataba de una triple hélice. Tendría que llegar la foto 51, obtenida por Franklin, a despejar las dudas: la molécula de ADN era una doble hélice.
Las claves científicas del error de la triple hélice de Pauling las explica Rosalind Franklin en su magnífico artículo de 1953, en el que muestra que tenía clara la estructura: los datos de Pauling no eran lo suficientemente resolutivos; además, una hélice con los fosfatos en el interior y las bases en el exterior no encaja, ni con las propiedades del ADN, ni con la química del fosfato; y, no menos importante, Pauling no tuvo en cuenta un hallazgo previo fundamental al que Franklin si da crédito.
En 1947, un joven bioquímico, Michael Creeth, propuso un modelo de ADN formado por dos cadenas unidas por puentes de hidrógeno entre sus bases. Los mentores de Creeth eran Gulland y Jordan, quienes habían demostrado el apareamiento entre bases del ADN. Esta observación, para Franklin, tal como ella misma cuenta en su artículo de 1953, es fundamental. Considerando los pares de bases y los datos de difracción, la famosa doble hélice emergía como la estructura correcta.

Consciente de que los datos de Bell y Astbury no tenían resolución suficiente, Pauling escribió a Wilkins, solicitando ver los suyos. Wilkins, que no deseaba que el gran Pauling tomara el control de la investigación, se negó. Suele decirse que Pauling no tuvo oportunidad de reunirse con ellos directamente, debido a que el gobierno de EEUU le denegó la renovación del pasaporte para viajar a Gran Bretaña en 1951. Lo cierto es que el impacto de esta anécdota no fue grande, pues, finalmente, Pauling visitó Inglaterra durante un mes en 1952, coincidiendo con el trabajo clave de Franklin y, durante el cual, lamentablemente, no prestó atención al ADN.
Mientras Pauling, pensando que Wilkins seguiría negándose a colaborar, se centraba en sus trabajos con las proteínas, Franklin y su doctorando Gosling obtenían las imágenes históricas que confirmaron la estructura del ADN. Franklin no tenía inconvenientes para mostrar sus resultados y, si Pauling hubiera hablado con ella directamente, la historia del ADN sería distinta. Es más, ocurrió algo extraño: Franklin mostró sus imágenes a Corey, mano derecha de Pauling. Corey sugirió a Pauling que el modelo de triple hélice no encajaba bien y que había varios problemas. Pero él insistió, pensando que, bueno, ya resolverían los problemas pendientes. No visitar a Franklin y no escuchar a Corey fue un error histórico.
No fue el único error de Pauling. Watson y Crick tuvieron en cuenta otro dato clave: el bioquímico austriaco Erwin Chargaff les explicó que las bases del ADN siguen una proporción muy sencilla, que hoy conocemos como reglas de Chargaff. Estas apoyaban la idea de que las dos cadenas estarían unidas por las bases. Pauling y Chargaff se conocieron en 1947 durante un crucero de vuelta a EEUU y hablaron de ello; pero, considerándole un tipo «molesto y desagradable», Pauling despreció sus observaciones.
El modelo erróneo de Pauling fue el impulso definitivo que llevó a Watson y Crick a publicar, sólo dos meses después, su doble hélice. Cuando el artículo de Pauling apareció en febrero de 1953, los británicos estaban sorprendidos: era un modelo ingenuo, erróneo, similar al que ellos concibieron en 1951 y que Franklin les hizo desechar, y que, como los mismos Watson y Crick comentaron, incluso violaba las reglas químicas básicas del fosfato, expuestas por el propio Pauling en su famoso libro de texto de Química General. Eufóricos por la oportunidad que el error de Pauling les brindaba, era el momento perfecto para arrebatarle la gloria de resolver la estructura del ADN; se apresuraron, entonces, a publicar su modelo antes de que Pauling tuviera tiempo de darse cuenta de su fallo y rehacer su modelo, a la vista de los nuevos datos de difracción de rayos X.

Así, en abril de 1953, tras un acuerdo con Wilkins y Franklin, se publicaron tres artículos: uno por Watson y Crick, otro por Wilkins y el tercero por Franklin y Gosling, detallando la estructura del ADN y los datos esenciales que la sostienen. Sorprende que mucha gente piense que aquel mes se publicó un sólo artículo, firmado por Watson, Crick y Wilkins, y que Franklin fue ignorada completamente. Como ocurre con El Quijote, que todo el mundo lo conoce, pero casi nadie lo ha leído. Sin embargo, la lectura de los artículos aclara cosas, como que, en efecto, no fue un artículo, sino tres, cada uno preparado por los líderes del descubrimiento: Watson y Crick eran los teóricos que desarrollaron el modelo, y Wilkins y Franklin los experimentalistas que lograron preparar el ADN puro y obtener los datos de su estructura. A priori no parece mal arreglo. Con ojos actuales, cada uno de ellos tendría su paper histórico en Nature como IP (Los problemas de índole personal que llevaron a que Wilkins y Franklin publicaran por separado, o por qué publicaron tres papers separados en lugar de uno todos juntos, no son objeto de esta entrada, y es un tema que se discute mucho, aunque para mi forma parte del gossip científico, no de la ciencia en sí)
Aceptando la derrota
Pauling aceptó sus errores con elegancia y, en la Conferencia Solvay de ese mismo abril de 1953, expresó su apoyo al modelo de Watson y Crick:
«Aunque solo han pasado dos meses desde que el profesor Corey y yo publicamos nuestra estructura propuesta para el ácido nucleico, debemos admitir que probablemente esté equivocada; Aunque se podría hacer algo de refinamiento, creo que es muy probable que la estructura de Watson-Crick sea esencialmente correcta»
En 1988, durante una conversación informal en un congreso, Pauling recapitulaba:
«Supongo que siempre pensé que la estructura del ADN era mía para resolver y, por lo tanto, no la perseguí con suficiente agresividad».
Definitivamente, no escuchar las señales de que su modelo no era correcto y su ambición, que le llevó a estar convencido de que era el único que podría resolverla, no le ayudaron.
Anexo 1: Cómo ayudó la foto 51 a revelar la estructura
La técnica de difracción de rayos X (DRX) es y ha sido esencial para la Biología. Es una técnica compleja, costosa y que requiere de una larga formación específica, por lo que los cristalógrafos, como lo fueron Rosalind Franklin, Florence Bell o el propio Pauling, expertos en DRX, son muy valiosos para la investigación y su labor no siempre es suficientemente reconocida. Prácticamente todas las estructuras que podemos encontrar en el Protein Data Bank, por ejemplo, han sido resueltas mediante DRX.
Para obtener un buen patrón de difracción, o difractograma, es necesario disponer de una muestra muy pura del compuesto que se está analizando, uniforme y cristalina (es decir, todas las moléculas de la muestra están ordenadas). Esto fue muy difícil de conseguir con el ADN, en especial por su tendencia a la transición entre ADN A y ADN B según su contenido en agua.
Rosalind Franklin y Raymond Gosling (a quien nadie recuerda, a pesar de haber trabajado en el laboratorio codo con codo con Franklin) consiguieron una imagen excepcional, como hemos visto: la famosa foto 51. Esta foto se reproduce en muchas ocasiones, pero ¿qué significa? ¿cómo se interpreta?

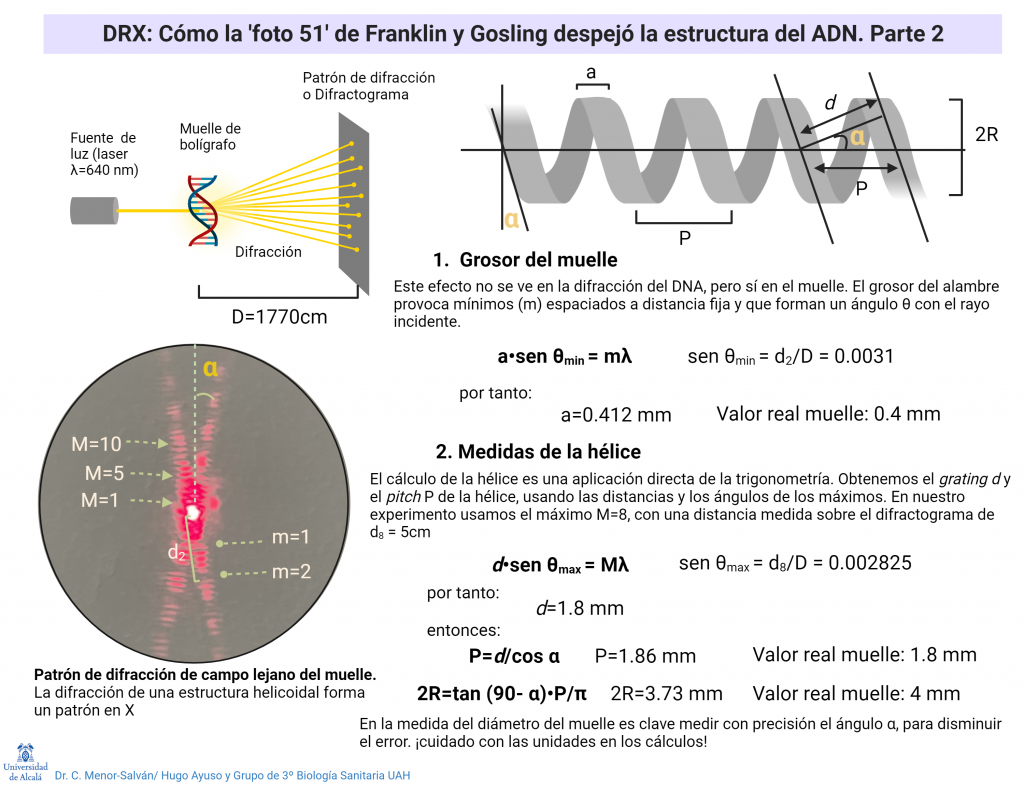
La interpretación completa es muy compleja, pero podemos entenderla de modo sencillo, en especial si usamos un experimento: si tomamos un muelle de boli o cualquier otro muelle pequeño y fino y lo iluminamos con un láser, que tenga el haz cuanto más grueso mejor (de modo que ilumine varias vueltas de hélice al mismo tiempo), el muelle va a difractar la luz laser. Como la diferencia entre la distancia de vueltas del muelle y la longitud de onda del laser es muy alta, el patrón de difracción que se produce es la difracción de campo lejano o difracción de Fraunhofer, que podemos ver al proyectar la luz a una distancia de entre 2 y 6 metros del muelle. El patrón de difracción nos permite medir con mucha precisión las medidas del muelle. Si cambiamos el muelle por moléculas ordenadas de un compuesto, y el láser por un haz fino de rayos X, muy penetrante y de longitud de onda muy pequeña, comparable a las dimensiones de la molécula, obtendremos un patrón de difracción de su estructura.
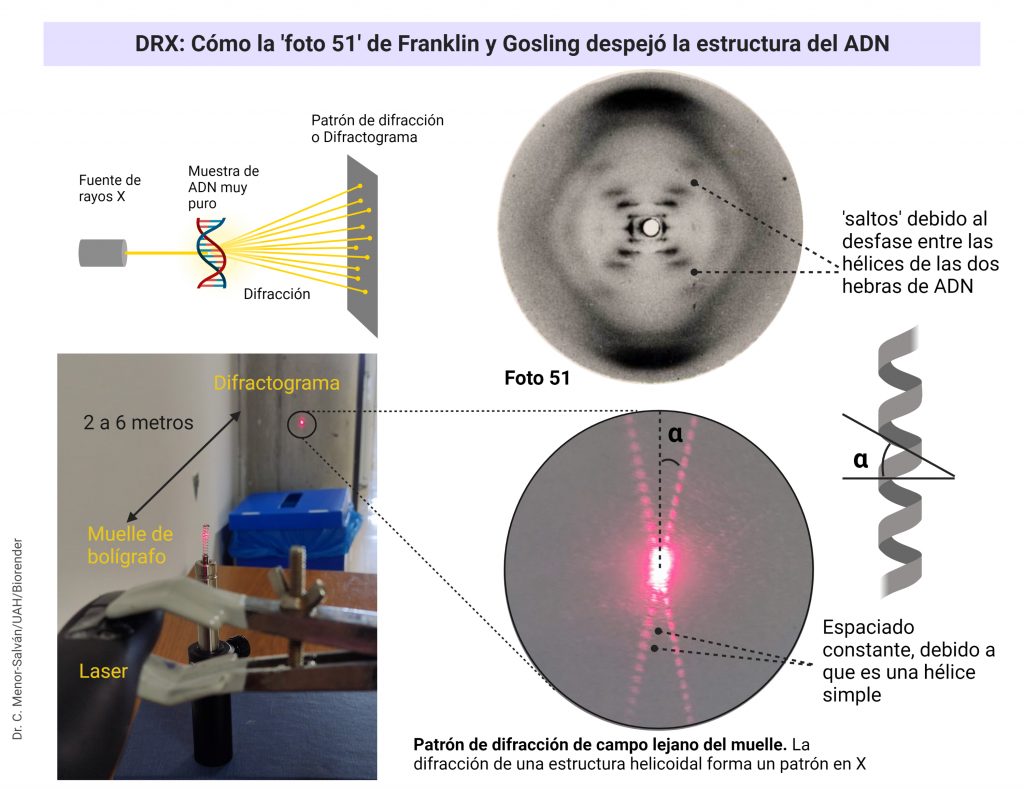
El patrón de difracción de una estructura en hélice da lugar a una «X» muy característica. El ángulo de la «X» y la distancia entre las manchas luminosas nos permite calcular las medidas de la hélice (ver mas abajo). Con el sencillo experimento del muelle y el puntero láser, podemos reproducir el experimento y los cálculos que realizaron Franklin y Gosling.
Si nos fijamos en las imágenes de Florence Bell, este patrón no está nada claro, debido a las dificultades que tuvo para preparar la muestra. Pauling supo que la molécula de ADN era helicoidal porque vio una imagen de microscopía electrónica.
Otro aspecto clave de la foto 51 es que revelaba claramente que el ADN no estaba formado por una hélice, sino por dos hélices unidas, con un desfase entre ellas. Este desfase daba lugar a los huecos que se pueden ver en la foto. Este dato confirmaba definitivamente que el ADN estaba formado por dos hebras unidas formando una hélice doble. Franklin pudo medir la molécula usando el patrón, obteniendo un ajuste muy bueno con el modelo teórico propuesto por Watson y Crick.
¿Qué habría ocurrido si el modelo de Creeth hubiera sido correcto? El patrón de difracción habría sido completamente distinto. Podemos modelizarlo también mediante la difracción de Fraunhofer de dos alambres paralelos muy juntos:

Esto nos da un patrón de difracción o difractograma lineal:

Como en el caso anterior, con el espaciado entre las manchas luminosas podemos medir la distancia entre los dos alambres. Comparemos este difractograma con el difractograma de rayos X real de un poliester, un polímero lineal formado por cadenas paralelas:
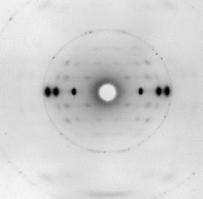
Y aquí vemos el patrón de difracción de un fragmento proteico formado por láminas beta paralelas:

Así queda demostrada la estructura de la molécula usando un método físico clave, la DRX. La verdad es que las cosas son como tienen que ser: si el DNA tuviera la estructura de Creeth, la Biología Molecular sería imposible, y por tanto la vida. Pero eso todavía no lo sabían en los años 1940-1950.
Anexo 2: Extracto de los artículos históricos de abril de 1953
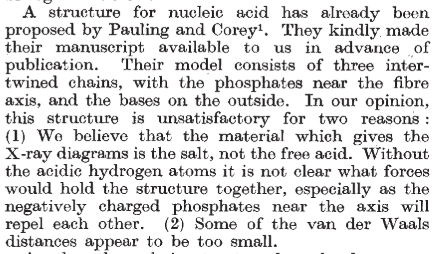



Referencias
ASTBURY, W. T. & BELL, F. O. (1938) ‘X-Ray Study of Thymonucleic Acid’, Nature, 141(3573), pp. 747–748. doi: 10.1038/141747b0.
Dahm, R. (2008) ‘Discovering DNA: Friedrich Miescher and the early years of nucleic acid research’, Human Genetics, 122(6), pp. 565–581. doi: 10.1007/s00439-007-0433-0.
FRANKLIN, R. E. & GOSLING, R. G. (1953) ‘Molecular Configuration in Sodium Thymonucleate’, Nature, 171(4356), pp. 740–741. doi: 10.1038/171740a0.
Gann, A. & Witkowski, J. A. (2013) ‘DNA: Archives reveal Nobel nominations’, Nature, 496(7446), p. 434. doi: 10.1038/496434a.
Harding, S. E., Channell, G. & Phillips-Jones, M. K. (2018) ‘The discovery of hydrogen bonds in DNA and a re-evaluation of the 1948 Creeth two-chain model for its structure’, Biochemical Society Transactions, 46(5), pp. 1171–1182. doi: 10.1042/BST20180158.
Lake, J. (2001) ‘Why Pauling didn’t solve the structure of DNA’, Nature, 409(6820), pp. 558–558. doi: 10.1038/35054717.
Levene, P. a, Hydrolysis, A. & Rockejeller, T. (1917) ‘Yeast Nucleic’, J. Biol. Chem., 40, pp. 415–424.
Pauling, L. & Corey, R. B. (1953) ‘A Proposed Structure For The Nucleic Acids’, Proceedings of the National Academy of Sciences, 39(2), pp. 84–97. doi: 10.1073/pnas.39.2.84.
Pederson, T. (2020) ‘The double helix: “Photo 51” revisited’, The FASEB Journal, 34(2), pp. 1923–1927. doi: 10.1096/fj.202000119.
Ronwin, E. (1953) ‘The Phospho-Di-Anhydride Formula and Its Relation to the General Structure of the Nucleic Acids’, Science, 118(3071), pp. 560–561. doi: 10.1126/science.118.3071.560.
Schmidt, G. (1950) ‘NUCLEIC ACIDS, PURINES, AND PYRIMIDINES’, Annu. Rev. Biochem, 19, pp. 149–186.
Thompson, J. et al. (2018) ‘Rosalind Franklin’s X-ray photo of DNA as an undergraduate optical diffraction experiment’, American Journal of Physics, 86(2), pp. 95–104. doi: 10.1119/1.5020051.
WATSON, J. D. & CRICK, F. H. C. (1953) ‘Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid’, Nature, 171(4356), pp. 737–738. doi: 10.1038/171737a0.
Linus Pauling and the race for DNA: http://scarc.library.oregonstate.edu/coll/pauling/dna/index.html
https://www.historyofinformation.com/detail.php?entryid=4425