Una mutación en la glucosa-6-fosfato dehidrogenasa y su influencia en la constante de Michaelis-Menten y la eficiencia catalítica de la enzima.
La glucosa-6-fosfato dehidrogenasa (G6PDH) es una enzima fundamental en el metabolismo central. Conecta la glucolisis con el metabolismo de los azúcares en el ciclo de las pentosas fosfato, a través de la oxidación de glucosa-6-fosfato a 6-fosfogluconolactona por NADP+. La reacción es una fuente de NADPH, que es un cofactor crucial en algunos procesos bioquímicos, como la recuperación del glutation, un pequeño péptido esencial para evitar daños por estrés oxidativo.
La recuperación de glutation es especialmente significativa en los glóbulos rojos, en los que esta reacción es su única fuente de NADPH. Si sus niveles se ven comprometidos, los glóbulos rojos se ven afectados por daño oxidativo y puede producirse anemia hemolítica
Esto puede tener lugar por deficiencia de la enzima G6PDH, que es, precisamente, una de las enzimopatías más comunes, afectando a casi 500 millones de personas en todo el mundo, principalmente en Africa, Peninsula Arábica y Sudeste Asiático. Se han descrito 230 mutaciones con relevancia clínica, principalmente hombres, al estar ligadas al cromosoma X. Las más graves producen anemia hemolítica y las menos severas producen hemolisis asociadas a tóxicos que producen estrés oxidativo o algunas toxinas, como las lectinas presentes en las judías.
La razón de esa distribución es que, curiosamente, la deficiencia de esta enzima confiere resistencia contra la malaria producida por el parásito Plasmodium falciparum. Entonces, en las zonas donde la enfermedad es endémica, las mutaciones de G6PDH proporcionan una ventaja evolutiva, prevaleciendo gradualmente sobra la enzima más activa, ya que el parásito requiere de ésta para su ciclo vital.
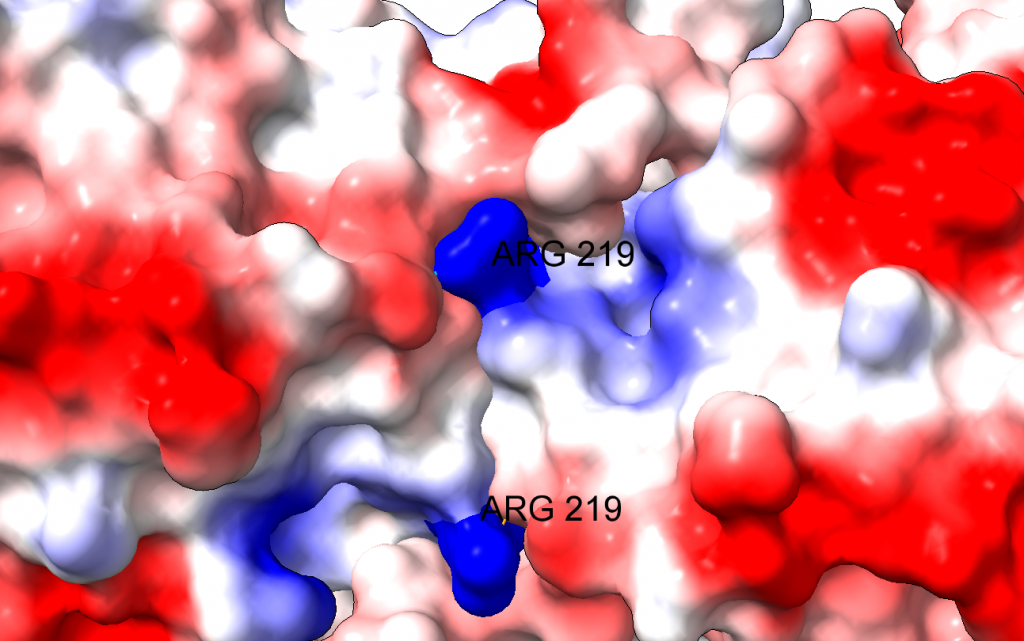
Mutación en la arginina 219
En el trabajo publicado en Nature Communications por Zgheib et al., se describe un caso muy interesante desde el punto de vista de la bioquímica estructural. Un joven paciente de 15 años sufrió hemolisis y pancitopenia tras una infección viral. Los autores estudiaron el caso y, tras ver en su historia que un tío suyo tenía deficiencia de G6PDH, llevaron a cabo el estudio bioquímico de la enzima.
Descubrieron una nueva mutación en la enzima, en la que una arginina en la posición 219 se había sustituido por una glicina. Esta mutación puede estar asociada a un cambio de un simple nucleótido, pues los codones de glicina y arginina sólo se diferencian en su primer nucleótido, siendo G para arginina y C para glicina.
El problema es que el cambio de un aminoácido con carga como la arginina por un aminoácido neutro de pequeño tamaño como la glicina puede tener importantes implicaciones en la estructura de la proteína. En efecto, la arginina 219 estabiliza la estructura proteica, que es un dímero, a través de un puente de hidrógeno y de la carga positiva, por interacción electrostática. Al cambiar por la glicina, se pierden esas interacciones electrostáticas en una posición clave, desestabilizándose la estructura de la enzima y dificultando su acción catalítica. Los autores comprueban que, en efecto, la enzima mutante es mucho más sensible a la desnaturalización, debido a la desestabilización estructural. La enzima mutante pierde actividad a temperatura superior a 44ºC, mientras que la enzima WT (wild type, la enzima funcional normal) pierde actividad a temperaturas superiores a 50ºC.
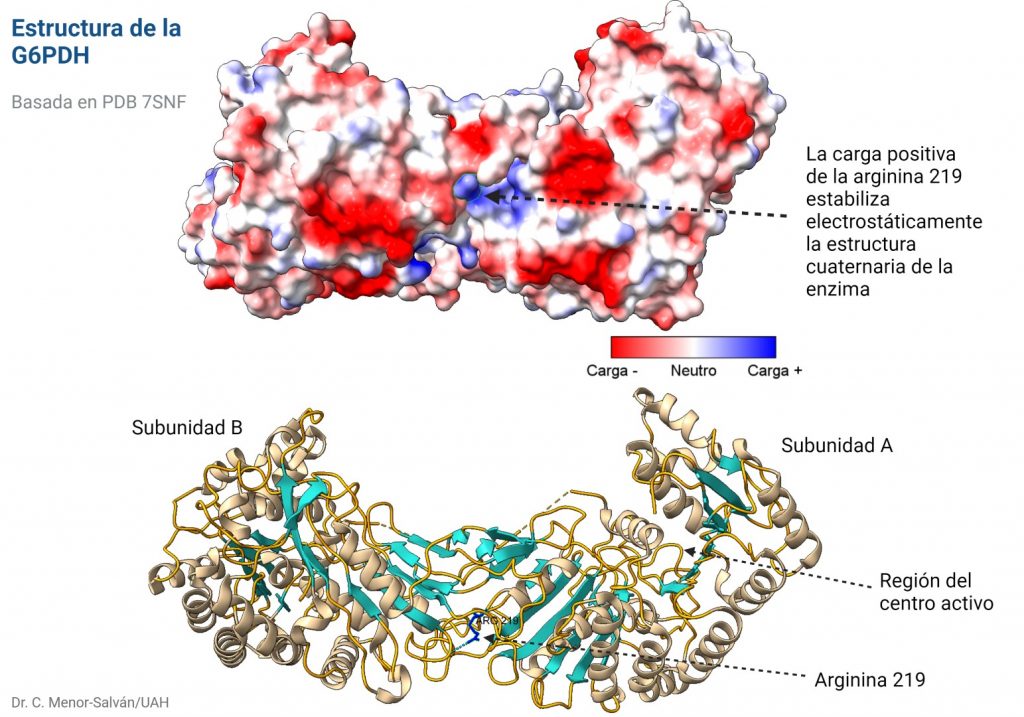
Es interesante que la arginina 219 no está en el centro activo de la enzima. Así, podemos explicar los datos experimentales de la actividad de la enzima que obtienen los autores:
- Km=49.5 µM para el sustrato (glucosa 6 fosfato) en la enzima WT
- Km=46.1 µM para el sustrato en la enzima con la mutación
- Kcat=326 s-1 para la enzima WT
- Kcat=6 s-1 para la enzima mutante.
¿que nos dicen estos resultados?. Que la enzima mutante muestra una constante de Michaelis-Menten similar a la WT para el sustrato glucosa-6-fosfato, por lo que no ha perdido afinidad por el sustrato; no es sorprendente, pues la mutación no afecta al centro activo y la unión sustrato-enzima no se ha visto afectada. Sin embargo, la enzima mutante ha perdido eficiencia catalítica. Recordemos que el Kcat o numero de turnover son los moles de sustrato que son transformados por mol de enzima y por segundo. Así, su actividad se ve enormemente reducida: la enzima WT es 54 veces más activa.
Este caso ilustra bien la diferencia de significado entre las dos constantes fundamentales de la actividad enzimática y cómo la discusión teórica que realizamos en la clase de Bioquímica Estructural tiene gran relevancia práctica.
¿Qué está ocurriendo a nivel termodinámico?
El hecho clave es que la mutación R219G varía poco la Km, pero reduuce drásticamente la Kcat. ¿hay una relación con la ΔG‡, variación de energía libre hacia el estado de transición?.
Podemos aproximar Km a la afinidad por el sustrato, es decir, a la estabilidad del complejo ES, mientras que Kcat se relaciona directamente con la barrera de activación que tiene que superar el sistema para alcanzar el estado de transición. Sabemos, según la teoría de Eyring, que ΔG‡ es inversamente proporcional a la velocidad de la reacción. Es decir, cuanta más alta sea la energía libre de transición, menor será la velocidad y la eficacia de la enzima:
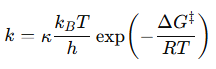
donde k es la constante de velocidad. Esta ecuación la podemos separar en sus componentes entrópica y entálpica:
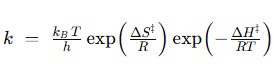
En las reacciones enzimáticas, la enzima acelera la reacción al disminuir la energía libre de activación necesaria para alcanzar dicho estado de transición, lo que consigue, bien por control entrópico, logrando que el paso del complejo ES al estado de transición sea favorable, ΔS‡>0 o próximo a cero, o bien por control entálpico, logrando que ΔH‡ se reduzca o sea negativo.
En este contexto, sabemos que kcat WT/kcat mut es 54.3, es decir, que la enzima mutante es 54 veces más «lenta». Para evaluar la diferencia entre las energías libres de activación, podemos simplificar a:
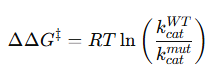
dado que
ΔΔG‡ = ΔGmut‡ − ΔGWT‡
y que podemos sustituir k por kcat si ésta refleja el paso limitante asociado a cruzar la barrera de activación.
De los datos, obtenemos que ΔΔG‡ = 2.4 kcal/mol. Es decir, que la mutación eleva la barrera impuesta por la energía de activación respecto a la enzima silvestre. Esto implica que el estado de transición es menos accesible, provocando la caída de velocidad. Como se ve, un cambio pequeño en la energía de activación tiene una consecuencia importante en la actividad de la enzima.
Haciendo un cálculo similar para la energía libre de unión obtenemos que ΔΔGB es -0.04 kcal/mol, un valor muy pequeño. La unión de sustrato es algo más favorable en el mutante, pero muy pequeño y no sabemos si esta diferencia puede ser simple error experimental, así que vamos a considerar que la mutación R219G no altera apreciablemente la energía libre de unión del sustrato o la estabilidad del complejo ES.
La reducción de Kcat sugiere que la Arg219 desempeña un papel clave en la estabilización del estado de transición. Dada la posición de la mutación, critica para la estabilidad del dímero de G6PD, podemos explicar el efecto de dos maneras:
Arg219 es esencial para estabilizar el dímero, y es éste la forma activa de la proteína. Ello favorece que la enzima mutante esté como una mezcla de monómero y dímero, mientras que la WT es sólo el dímero activo. Sabemos que la Kcat son los moles de sustrato convertidos en producto por mol de enzima y unidad de tiempo. Si disminuimos la concentración de enzima activa, aunque la concentración de enzima total se mantenga, tendremos que
Kcat aparente = factiva x Kcat
Es decir, que la enzima mutante tendrá una Kcat aparente que es la fracción de enzima activa por la Kcat de la enzima activa. Si despejamos obtenemos que factiva = 1.8%. ¡la mayor parte de la enzima estaría desactivada!
De ahí que su eficiencia baje: aunque la enzima total es constante y tiene capacidad para unir el sustrato (y por ello Km se mantiene igual), la mayor parte de la enzima es incapaz de llevar a cabo la reacción.
Por otro lado, la mutación puede haber alterado la organización estructural. Al perderse el puente salino que forma la arginina, aumenta la entropía conformacional (aumenta la flexibilidad y la «libertad» de la enzima para adoptar diversas conformaciones), penalizando el término TΔS‡. Con la mutación, el «salto» al estado de transición implica que tiene que restringir más la «libertad» de la estructura, lo que hace que ΔS‡, que antes era favorable, se haga más pequeño, o incluso negativo, elevando la energía libre de activación. Esto encaja con la pérdida de actividad a temperatura más baja en la enzima mutante.
Curiosamente, este caso también nos explica por qué el modelo llave-cerradura no es posible, ya que implica una alta estabilización del complejo ES unida a un alto coste entrópico para obtener el estado de transición.
Como se ve, pequeños cambios, como una mutación en un aminoácido o una pequeña variación en las energías del proceso tienen importantes consecuencias fisiológicas y médicas.
Referencias
Zgheib, O. et al. (2023) ‘Substitution of arginine 219 by glycine compromises stability, dimerization, and catalytic activity in a G6PD mutant’, Communications Biology. Springer US, 6(1), p. 1245. doi: 10.1038/s42003-023-05599-z.