ADN G-cuadruplexos, diana farmacológica frente al cáncer
Realizado por Ana Jiménez y Cristina Iruela – 3º de Biología Sanitaria, UAH
Los G-cuadruplexos son unas estructuras químicas que llevan años en el punto de mira por su característica estructra y localización. Cada vez se apuesta más por ellos como terapia frente al cáncer dada su interacción con estrucutras y moléculas íntimamente relacionadas con la enfermedad. A continuación se expondrá una breve revisión sobre el tema.
Estructura y función de los ADN-G cuadruplexos
Las secuencias de ADN ricas en guanina pueden plegarse en estructuras secundarias no canónicas de cuatro cadenas denominadas G-cuadruplexos (G4). Estas estructuras secundarias se forman tanto en el ADN como en el ARN. Consiste en 4 guaninas unidas por puentes de hidrógeno de tipo Hoogsteen, en los que cada guanina puede actuar como donante y aceptor de dos puentes de hidrógeno formando una estructura plana denominada tétrada G [1].
Dos o más tétradas G se pueden apilar una encima de otra para formar un G-cuadruplexo, siendo esta su unidad estructural. Esta se forman conectando 4 guaninas a través de 8 puentes de hidrógeno. En la tétrada G, se forman dos de estos puentes que emparejan guaninas adyacentes, en los que están involucrados los nitrógenos número 1,7, 2 y el oxígeno 6 de cada nucleótido de guanina [2].
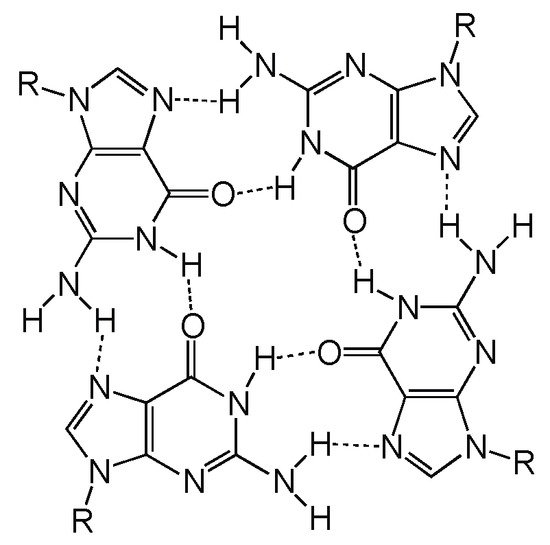
Estructura química de una tétrada G
Tomada de Kolesnikova, S., & Curtis, E. A. (2019). Structure and Function of Multimeric G-Quadruplexes. Molecules (Basel, Switzerland), 24(17), 3074. https://doi.org/10.3390/molecules24173074
Además, es necesaria la presencia de un catión metálico (Na+, K+) para estabilizar la estructura [3].
En el ARN, los G4 formados en la región 5’UTR del ARNm inhiben la traducción dependiente de cap y mejoran la traducción independiente de caperuza mediada por IRES. También influyen en otros mecanismos moleculares que tienen lugar en el ARN, como el empalme, cambios en el marco de lectura, localización del ARNm o la maduración de los miARN [3].
En base a experimentos in vitro, se predijo que los G-cuadruplexos se forman en regiones que albergan un motivo G4 específico. Sin embargo, estudios actuales muestran que también pueden formarse dentro de regiones con bucles formados por 3 o más guaninas por repetición, así como en regiones que no siguen este motivo G4 estricto [1].
No se distribuyen al azar en todo el genoma, sino que abundan en ciertas regiones, como promotores, telómeros, sitios de unión de factores de transcripción u orígenes de replicación. La estabilidad de esta estructura depende, entre otros factores, del número de guaninas por repetición y de la longitud de los bucles [1].
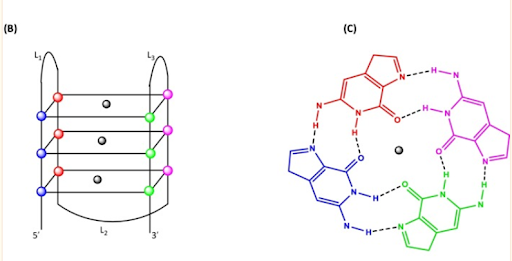
Estructura de los G-cuadruplexos
Nota: B) Una representación 2D de un pliegue G4 típico que muestra los tres cuartetos planos. Las esferas en los vértices de los cuartetos representan una guanina de cada uno de los cuatro G-tripletes. La esfera negra en el centro denota el catión metálico central (Na + , K + ) necesario para estabilizar la estructura G4. (C) Una vista superior de un cuarteto G plano que muestra los enlaces Hoogsteen (líneas discontinuas), los átomos de los mismos y un catión en la cavidad central. Las figuras no están dibujadas a escala.
Fragmento tomado de: Saranathan, N., & Vivekanandan, P. (2019). G-Quadruplexes: More Than Just a Kink in Microbial Genomes. Trends in microbiology, 27(2), 148–163. https://doi.org/10.1016/j.tim.2018.08.011
La relevancia fisiológica de estas estructuras se debe a la existencia de proteínas que pueden unirse a ellas o desplegarlas. Existen 3 clases de proteínas que interactúan con los G-cuadruplexos descritas en la literatura: proteínas de unión a G-cuadruplexos, estabilizadoras de G-cuadruplexos y desarrolladoras de G-cuadruplexos (como helicasas). Se ha descrito qué mutaciones y/o delecciones en estas proteínas conducen a cambios en la formación de estas estructuras. Lo que, a su vez, puede dar lugar a cambios en las vías biológicas (cambios transcripcionales) y aumentar la inestabilidad del genoma [1].
La formación transitoria de G4 en condiciones termodinámicamente favorables tiene funciones reguladoras importantes dictadas por su ubicación en el genoma [3]. Entre ellas se encuentran la regulación de la transcripción, traducción, replicación del ADN y localización del ARN [4]. Destaca la función de los G-cuadruplexos en relación a la inhibición de la actividad de la telomerasa [3].
Relación con los telómeros + telomerasa + cáncer
Como ya se ha mencionado, los telómeros son un ejemplo de la presencia de G4 cuadruplexos en el genoma de los vertebrados, basándose en la secuencia consenso: (5’-TTAGGG-3’) [5] que evidencia la presencia repetitiva de las guaninas (dicha secuencia es específica para los mamíferos y cambia según la especie de los mismos).
Para comprender la importancia de los telómeros, es clave entender la estructura de los mismos, cuya formación es una respuesta evolutiva al problema encontrado en los extremos 3’ cuando la maquinaria de replicación de nuestras células no puede rellenar el hueco al no tener un extremo 5’ anterior que le sirva de molde para la síntesis de la nueva cadena. Esto tiene como resultado la formación de un T-loop y un D-loop originados por la invasión de un extremo 3’ que sobresalía respecto al extremo 5’ complementario [6]. Además, se encontrará el complejo de Shelterina, el cual poseerá diferentes proteínas que regularán la actividad de la telomerasa, enzima encargada de la elongación de los telómeros por medio de la adición de unidades (TTAGGG).
Esta respuesta evita la pérdida de información en cada ronda de replicación y evitan que la célula reconozca esta región sobrante como un daño en el ADN y lo elimine. De todas maneras, estos telómeros se irán acortando igualmente con el tiempo: acortamiento telomérico de Hayflick, resultando en un punto crítico de longitud activando la llamada senescencia replicativa, siendo este proceso la base del envejecimiento celular que resulta en poner fin a su división [7]. La regulación de dicha senescencia es clave para el organismo para evitar su envejecimiento y como supresor de tumores [8].
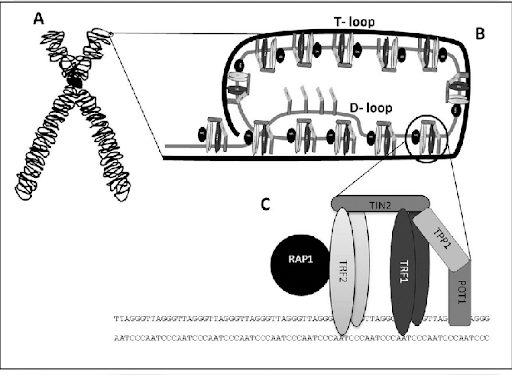
Representación de un cromosoma y terminación telomérica
Nota: A) Esquema de un cromosoma indicando la ubicación de un telómero. B) Estructura del telómero: T-loop secuestrando el extremo terminal del cromosoma, y D-loop donde se observa la triple hebra de ADN. C) Complejo Shelterina de proteínas asociadas a los telómeros.
Tomado de Mengual Gomez, Diego & Armando, Romina & Farina, Hernán & Gomez, Daniel. (2014). Telomerasa y telómero: su estructura y dinámica en salud y enfermedad. Medicina. 74. 69-76.
Los G4 tienen un papel de represión de determinados genes en células sanas impidiendo la entrada de la maquinaria necesaria para la replicación y transcripción. En células sanas, estos evitan la expresión de oncogenes como: MYC, sufriendo así un proceso de regulación negativa [5].
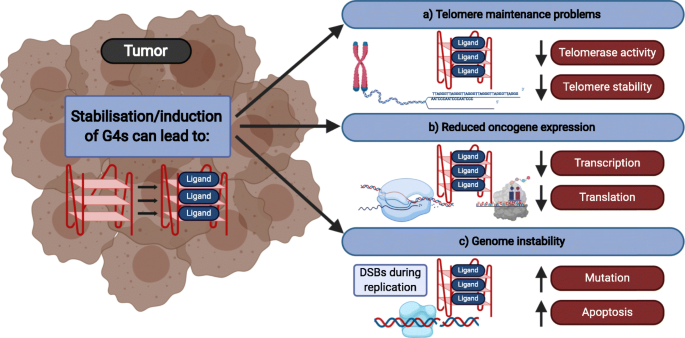
Resumen esquemático de los efectos de los ligandos de G4 en las células cancerosas
Tomado de: Kosiol, N., Juranek, S., Brossart, P., Heine, A., & Paeschke, K. (2021). G-quadruplexes: a promising target for cancer therapy. Molecular cancer, 20(1), 40. https://doi.org/10.1186/s12943-021-01328-4
Lo que ocurre en enfermedades como el cáncer es que el acortamiento de los telómeros se evita hasta tal punto que las células se inmortalizan y escapan al proceso de muerte celular. La base patológica de esto es la activación de la telomerasa la cual está además sobre expresada en los tejidos cancerosos [9], cuya activación será siempre el reflejo de una respuesta anómala. Los G4 presentes en los telómeros de sus células no tendrán la misma eficacia que en las células sanas, puesto que la telomerasa se introduce y favorece la elongación de dicho telómero. Este suceso tendrá como consecuencia el desarrollo del fenotipo inmortal que adoptarán las células del tejido afectado y que se volverán cancerosas [5]. Es importante destacar que la alteración de la unión de los G4 con la telomerasa se ha observado tanto in vivo como in vitro [1].
Cabe mencionar las regiones TERRA, región telomérica de RNA no codificante [5]. Esta, es el transcrito resultante del telómero llevado a cabo por la enzima RNA polimerasa II la cual puede aparecer como ARN nucleoplásmico libre o en forma de un nuevo loop en la estructura de los telómeros: R-loop (correspondiente a un híbrido entre ADN y ARN) [8].
Cuando el telómero se acorta hasta dicho punto crítico anteriormente mencionado, este R-loop se asocia con el resto de TERRA promoviendo la reparación dirigida por homología (denominada HDR-mediated). Este proceso va a permitir la recombinación del telómero con su propia secuencia perpetuando así su vida celular y evitando la senescencia prematura. Además, este mismo mecanismo será utilizado por algunas células cancerosas para la elongación de los telómeros en caso de no poseer telomerasa funcionando como mecanismo de alargamiento alternativo, siendo la base de los tumores ALT [8].
Paradójicamente, algunos estudios han dado evidencia de la longitud reducida de los telómeros de las células cancerígenas respecto a las células de tejidos libres de cáncer, así como un aumento del número de los G4 en las mismas [5][9]. Para esto se siguen formulando diferentes hipótesis.
Otras utilidades bioquímicas
Además de la función anteriormente mencionada, se han estudiado cada vez más aplicaciones:
- Son utilizados como sondas, solas o en complejo con hemina, una estructura de porfirina que contiene hierro para detectar la presencia de diferentes ligandos [10].
- También como transportadores, gracias a su capacidad para secuestrar ligandos, actuando como agentes de administración de fármacos [10].
- En los últimos años, se ha extendido su uso como fármacos, en concreto como aptámeros (ácidos nucleicos de cadena sencilla aislados de genotecas de oligonucleótidos por selección in vitro), interactuando con biomoléculas, como proteínas e interfiriendo con sus funciones [10].
- O como dianas farmacológicas explotando su capacidad para interactuar con ligandos específicos, lo que puede alterar funciones importantes si el G-cuadruplexo se encuentra en regiones esenciales en el genoma del virus o de la célula huésped [10].
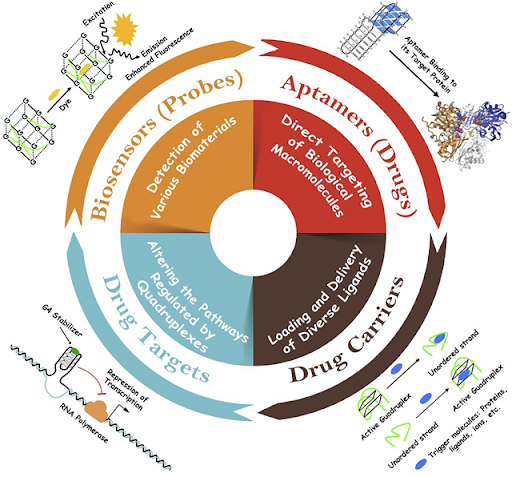
Aplicaciones de los G-cuadruplexos
Nota: Representación gráfica de las principales aplicaciones de los G-cuadruplexos.
Tomado de: Abiri, A., Lavigne, M., Rezaei, M., Nikzad, S., Zare, P., Mergny, J. L., & Rahimi, H. R. (2021). Unlocking G-Quadruplexes as Antiviral Targets. Pharmacological reviews, 73(3), 897–923. https://doi.org/10.1124/pharmrev.120.000230
Telomestatina
En múltiples estudios, se ha propuesto que las mejores dianas farmacológicas serían aquellas que solo se expresasen en las células cancerosas o aquellas que fuesen esenciales para mantener el fenotipo maligno de las mismas. La telomerasa, es una diana clave [6][7][9].
Se trata de un producto natural aislado de Streptomyces anulatus que es un ligando de los G4 teniendo una afinidad muy alta por la secuencia concreta de los telómeros: (5’-TTAGGG-3’). Al interaccionar, inhibe de manera eficaz la actuación de la telomerasa, por lo que se detiene la elongación de los telómeros de las células cancerígenas y como consecuencia suprime su proliferación. Esta actividad anticancerígena provoca que algunos de los factores claves encontrados en el complejo de Shelterina del telómero, como TRF2 y POT1, se liberen de dicho telómero, evitando así que lleven a cabo su función de retrasar la senescencia [6].
Además, la telomestatina es un ligando que tiene una mayor afinidad por los G4 intramoleculares, tanto si han sido formados a partir de un ADN telomérico dúplex, como de uno monocatenario, teniendo la función anteriormente mencionada. Esto supone una ventaja frente a otros compuestos como TMPyP4, el cual posee afinidad por los G4 intermoleculares y teniendo un efecto totalmente diferente el cual no se ha observado en la telomestatina: formación de puentes de anafase en erizos de mar [6].
A pesar de sus ventajas estabilizado los G4 cuadruplexos, arrastra algunas características que resultan contraproducentes así como sus solubilidad o inestabilidad, por lo que se empezaron a utilizar algunos compuestos análogos sintéticos [5].
Búsqueda de otros fármacos
En definitiva, la existencia de análogos sintéticos de G4s es lo que ha permitido contemplar una nueva forma de terapia para el cáncer [5][11], dado que reprime el correcto funcionamiento de las células cancerosas, llegando a conseguir la destrucción de la misma; así como análogos de la telomestatina [11], aunque estas terapias siguen en constante estudio y desarrollo.
El silvestrol es un compuesto obtenido de la corteza de los árboles de la familia flavaglina cuya estructura permite inhibir el factor de transcripción: eIF4A, tratándose de una análogo sintético. El factor posee una actividad helicasa clave para el proceso fisiológico de la transcripción al permitir deshacer las estructuras secundarias que pueden aparecer en la cadena de ADN y que impedirían la continuación del proceso. Al mismo tiempo tiene un papel clave en la carcinogénesis al facilitar la leucemia linfoblástica aguda de las células T al promover la transcripción de oncogenes como MYC, CDK6 o MDM2 al desenrollar los G4 de la región 5’ UTR de sus mRNAs. Este compuesto lo que hará, será inhibir al eIF4A [5], interfiriendo indirectamente en el mantenimiento de la estructura de los ADN G cuadruplexos.
Otro análogo que también afecta al gen MYC es: TMPyP4, anteriormente mencionado. Este se basa en la represión de proto-oncogenes de dicho gen por medio de la estabilización de los G4 cuadruplexos [5].
Los análogos “pirodistatina” y CX-3542 provocan daño en células cancerosas también. El primero, induce la formación de un nuevo loop en la estructura del telómero: “R-loop”, siendo un híbrido de DNA y RNA transcrito causando un daño en el ADN canceroso. El segundo causa daño y muerte celular con mayor eficacia en 2 tipos celulares cancerosos concretamente: células ATRX deficientes y células BRCA1/2 deficientes [5].
En relación a la función de estas estructuras como fármacos, existen secuencias cortas en los ácidos nucleicos derivadas del motivo hexanucleotido TGGGAG, denominadas “secuencias de Hotoda” que son potentes inhibidores anti-VIH. Estas secuencias cortas también se encuentran activas en otros virus como en los que aparecen secuencias de 6 nucleótidos con la siguiente estructura GGGGGT, la cual, da lugar a G-cuadruplexos. Este se une al dominio C-terminal de la proteasa del virus de la hepatitis A y es un fuerte inhibidor de la proteasa 3C de este virus [10]. Al inhibirla, impide que el virus descomponga sus proteínas para poder multiplicarse. Por lo tanto, deja de propagarse.
Un argumento notable es que estas secuencias cortas son demasiado cortas para ser específicas. Además, pueden actuar sobre otros componentes celulares del huésped, que se unen a estructuras secundarias de ADN no canónicas [10].
Otro fármaco que ha resultado ser un potente inhibidor de la telomerasa es RHPS4, tratándose de un mutante de la subunidad de la telomerasa denominada hTERT. La expresión de dicha subunidad mutante ha dado evidencias de inhibir el proceso de la telomerasa al unirse y competir por el sitio de unión. Tras estudiar su efecto en células tumorales, se concluyó que la línea celular MCF-7 de las células pertenecientes al cáncer de mama sufren una detención del crecimiento similar a la senescencia [7].
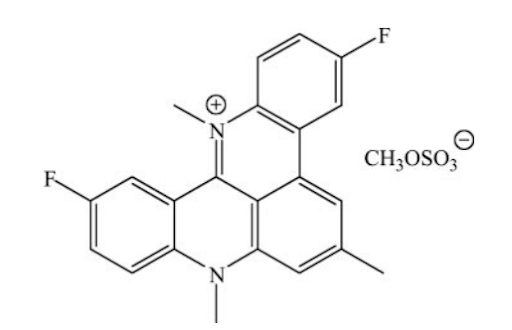
Estructura de RHPS4
Tomada de: Cookson, J. C., Dai, F., Smith, V., Heald, R. A., Laughton, C. A., Stevens, M. F., & Burger, A. M. (2005). Pharmacodynamics of the G-quadruplex-stabilizing telomerase inhibitor 3,11-difluoro-6,8,13-trimethyl-8H-quino[4,3,2-kl]acridinium methosulfate (RHPS4) in vitro: activity in human tumor cells correlates with telomere length and can be enhanced, or antagonized, with cytotoxic agents. Molecular pharmacology, 68(6), 1551–1558. https://doi.org/10.1124/mol.105.013300
Referencias consultadas
- Kosiol, N., Juranek, S., Brossart, P., Heine, A., & Paeschke, K. (2021). G-quadruplexes: a promising target for cancer therapy. Molecular cancer, 20(1), 40. https://doi.org/10.1186/s12943-021-01328-4
- Yuan, W. F., Wan, L. Y., Peng, H., Zhong, Y. M., Cai, W. L., Zhang, Y. Q., Ai, W. B., & Wu, J. F. (2020). The influencing factors and functions of DNA G-quadruplexes. Cell biochemistry and function, 38(5), 524–532. https://doi.org/10.1002/cbf.3505
- Saranathan, N., & Vivekanandan, P. (2019). G-Quadruplexes: More Than Just a Kink in Microbial Genomes. Trends in microbiology, 27(2), 148–163. https://doi.org/10.1016/j.tim.2018.08.011
- Kolesnikova, S., & Curtis, E. A. (2019). Structure and Function of Multimeric G-Quadruplexes. Molecules (Basel, Switzerland), 24(17), 3074. https://doi.org/10.3390/molecules24173074
- Nakanishi, C., & Seimiya, H. (2020). G-quadruplex in cancer biology and drug discovery. Biochemical and biophysical research communications, 531(1), 45–50. https://doi.org/10.1016/j.bbrc.2020.03.178
- Kim, M. Y., Gleason-Guzman, M., Izbicka, E., Nishioka, D., & Hurley, L. H. (2003). The different biological effects of telomestatin and TMPyP4 can be attributed to their selectivity for interaction with intramolecular or intermolecular G-quadruplex structures. Cancer research, 63(12), 3247–3256.
- Cookson, J. C., Dai, F., Smith, V., Heald, R. A., Laughton, C. A., Stevens, M. F., & Burger, A. M. (2005). Pharmacodynamics of the G-quadruplex-stabilizing telomerase inhibitor 3,11-difluoro-6,8,13-trimethyl-8H-quino[4,3,2-kl]acridinium methosulfate (RHPS4) in vitro: activity in human tumor cells correlates with telomere length and can be enhanced, or antagonized, with cytotoxic agents. Molecular pharmacology, 68(6), 1551–1558. https://doi.org/10.1124/mol.105.013300
- Pérez-Martínez, L., Wagner, T., & Luke, B. (2022). Telomere Interacting Proteins and TERRA Regulation. Frontiers in genetics, 13, 872636. https://doi.org/10.3389/fgene.2022.872636
- Kelland L. R. (2005). Overcoming the immortality of tumour cells by telomere and telomerase based cancer therapeutics–current status and future prospects. European journal of cancer (Oxford, England : 1990), 41(7), 971–979. https://doi.org/10.1016/j.ejca.2004.11.024
- Abiri, A., Lavigne, M., Rezaei, M., Nikzad, S., Zare, P., Mergny, J. L., & Rahimi, H. R. (2021). Unlocking G-Quadruplexes as Antiviral Targets. Pharmacological reviews, 73(3), 897–923. https://doi.org/10.1124/pharmrev.120.000230
- Teng, F. Y., Jiang, Z. Z., Guo, M., Tan, X. Z., Chen, F., Xi, X. G., & Xu, Y. (2021). G-quadruplex DNA: a novel target for drug design. Cellular and molecular life sciences : CMLS, 78(19-20), 6557–6583. https://doi.org/10.1007/s00018-021-03921-8