El péptido de Navidad
Este año, mi postal de felicitación de Navidad a los biólogos ha sido así:
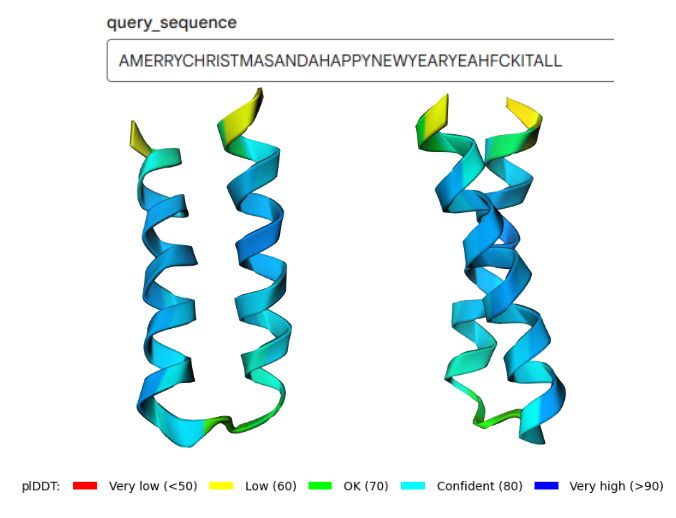
Representa la estructura del péptido imaginario
NH2-AMERRYCHRISTMASANDAHAPPYNEWYEARYEAHFCKITALL-COOH
Un péptido está formado por una secuencia de aminoácidos, normalmente 20, que forman una cadena. Esta cadena se pliega, dando lugar a una estructura tridimensional que llamamos proteína. Es decir, que un péptido contiene información usando un código de 20 letras, cada una de las cuales representa una aminoácido. Este código es:

Podría enviar un mensaje usando estas 20 letras, en forma de un péptido. El receptor del mensaje sólo tendría que ‘leer’ el péptido (usando secuenciación por espectrometría de masas, por ejemplo). En mi mensaje de navidad, he escrito la felicitación en forma de péptido. Si lo sintetizamos, ¿qué estructura tendría?
Utilizando la inteligencia artificial Alphafold 2, podemos hacer una predicción de la estructura tridimensional del péptido, es decir, la pequeña proteína que podría formar.
Análisis de la estructura del péptido
La predicción de la estructura nos dice que formaría un motivo con dos hélices alfa cruzadas:
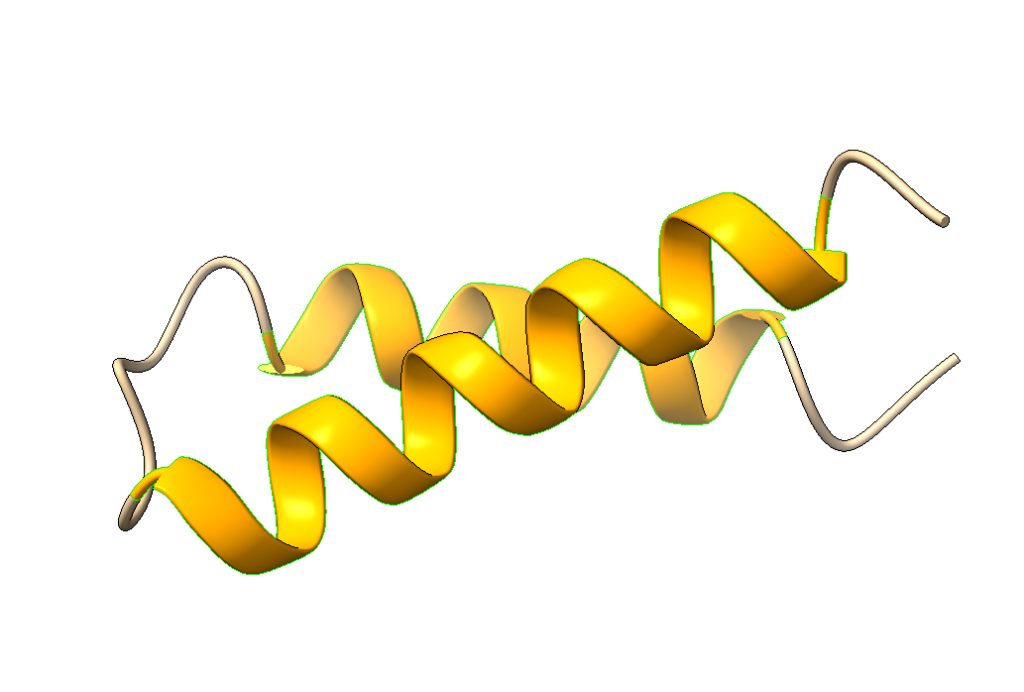

La estructura predicha es relativamente estable y, para ello, hay tres claves.
- Las cisteínas.
- Interacción hidrófoba en el punto de cruce de las hélices (similar a motivos de leucine zipper)
- Las prolinas.
Cisteína:
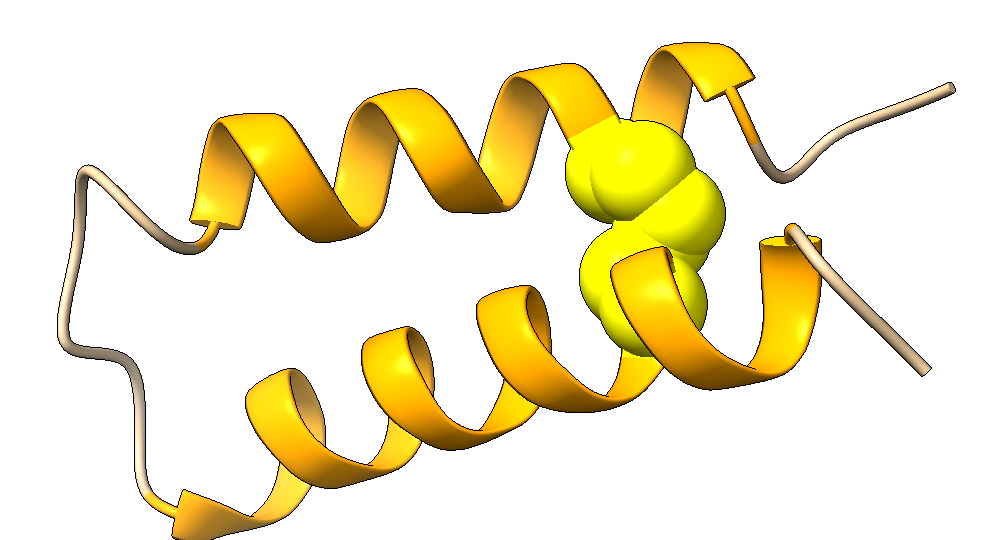
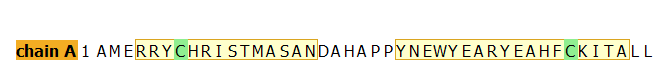
Las dos cisteínas de la cadena estan en la posición ideal para formar un puente disulfuro que va a estabilizar la estructura. Esta es la razón por la que añadí el «fckitall», además de dejar el par de prolinas en «happy» centradas en la secuencia.
Prolina:
Las dos prolinas en HAPPY son claves. La prolina es un aminoácido bastante peculiar, ya que es una amina secundaria que forma un anillo. La prolina rompe las hélices, creando una torsión que da punto de giro. Esta parte de la estructura es fundamental para «enfrentar» las dos hélices, y la torsión se asemeja a la cadena simple de colágeno.
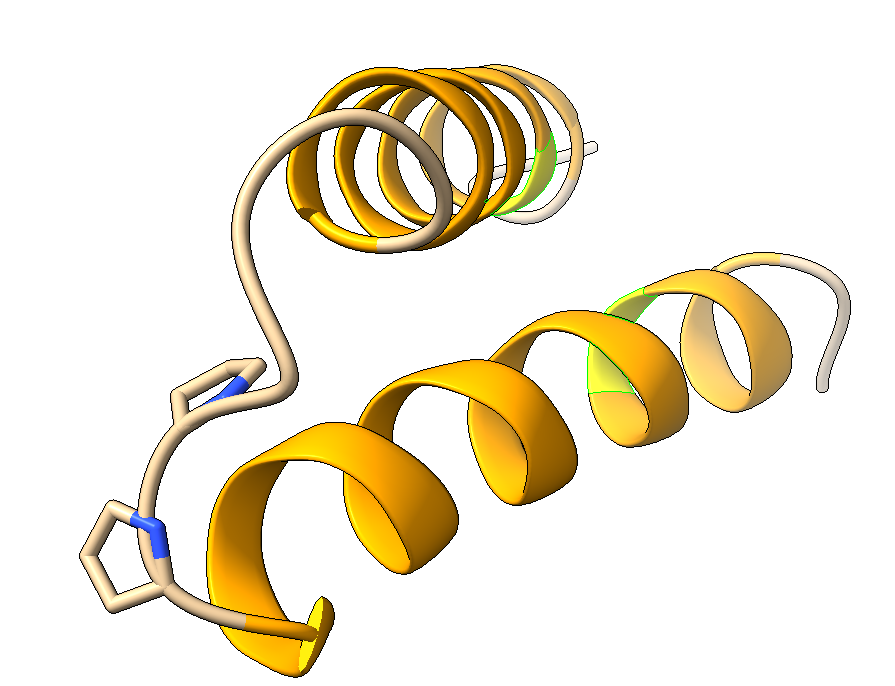
Interacción hidrófoba
Aqui juega a favor y en contra. Por un lado, la interacción hidrófoba dirige la orientación de las hélices y estabiliza el plegamiento, con una interacción intensa en la región central (en rojo):
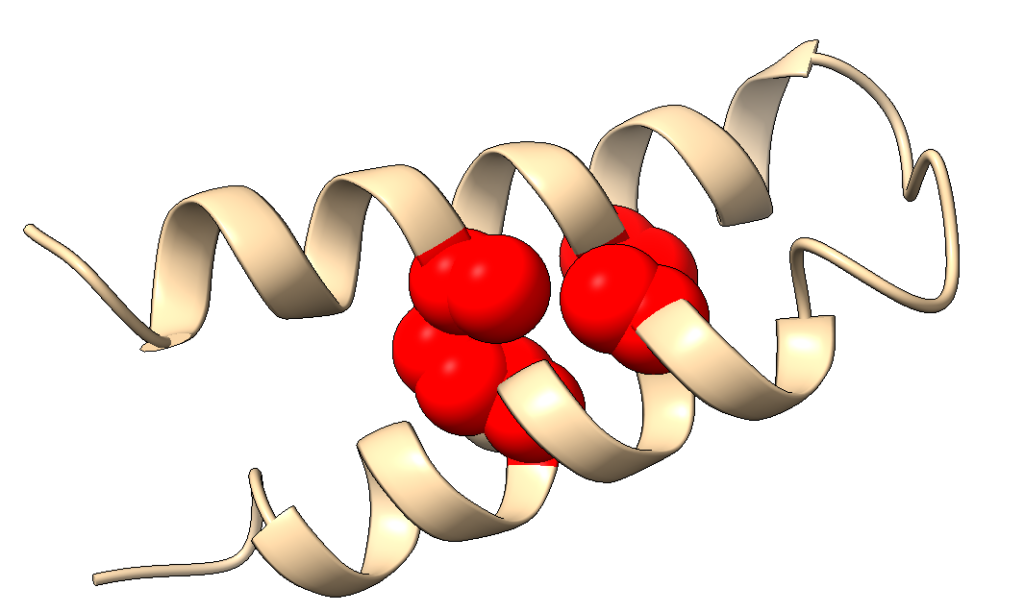
Por otro lado, hay aminoácidos hidrófobos (isoleucina, leucina, alanina) en posiciones terminales y expuestas, que podrían dar lugar a interacciones intermoleculares que generen una estructura distinta. Lo discutiremos un poco más adelante.
¿Podría existir esta estructura en realidad?
La predicción nos dice que es posible, con una probabilidad relativamente elevada (70%) de que forme esta estructura en realidad, aunque las posiciones de aminoácidos hidrófobos en los extremos de la cadena y el pequeño tamaño del péptido hacen probable que, si lo sintetizamos en realidad, forme un precipitado de agregados amorfos o bien amiloides.
Para evaluar posibles estructuras reales, tenemos la IA Alphafold 3, capaz de predecir estructuras resultantes de la interacción de varias moléculas de proteína o proteína con otros ligandos. Esto nos da perspectivas interesantes. Si evaluamos la estructura resultante de unir varias moléculas del péptido, obtenemos esto:
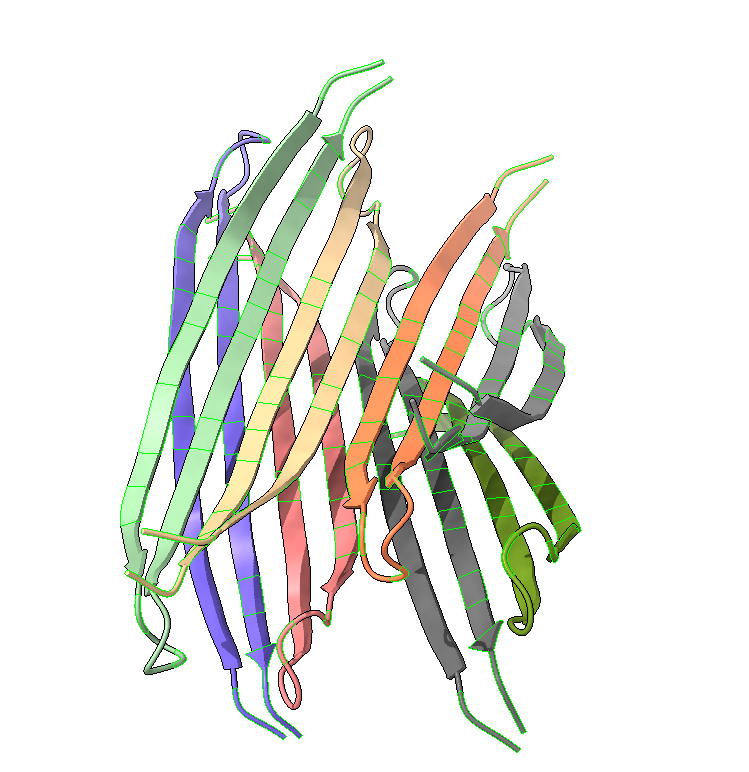
Esta estructura es un amiloide: láminas beta que se agrupan, hasta terminar formando agregados y fibras, llamadas fibras amiloides. Esta estructura está estabilizada por interacciones hidrofóbicas y electrostáticas, que favorecen el agregado.
Ligandos y estabilización de la estructura
Diversos ligandos, tales como cofactores (desde iones metálicos a moléculas como las coenzimas) y grupos prostéticos, como el grupo Hemo, son esenciales en la estabilización estructural. De hecho, es posible que el primer papel bioquímico de moléculas como los nucleótidos, fuera puramente estructural.
¿estabilizarán los ligandos nuestro péptido de navidad?
Si mezclamos el péptido con uno de los cofactores posiblemente más antiguos en la evolución, el AMP (adenosina monofosfato) ocurre algo maravilloso:
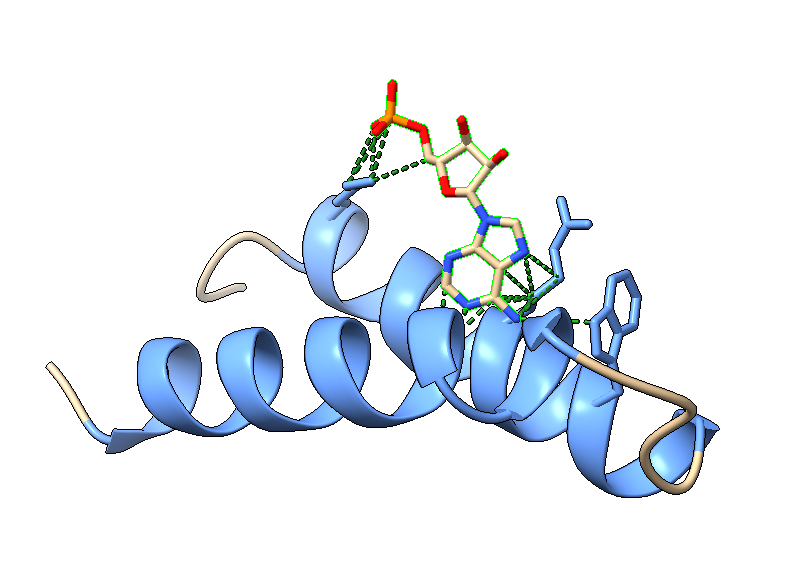
La molécula de AMP interacciona con el péptido, estabilizándolo y esta estructura alcanza una ¡probabilidad del 80%! de que se produzca en la realidad. Posiblemente, el primer papel bioquímico de los nucleótidos de adenina no fue ser parte del RNA, sino ser moléculas que interaccionaban con péptidos primordiales, dirigiendo su plegamiento y creando una fuerza selectiva hacia estructuras funcionales. Es posible que el RNA (o sus hipotéticos precursores similares, preRNA) surgieran en pasos posteriores de la evolución química.
Hablando de RNA: ¿que ocurrirá si estudiamos la interacción del péptido de navidad con un hairpin, uno de los plegamientos más simples de RNA?
La predicción nos dice que es posible, aunque con una probabilidad más baja (60%). La estructura resultante estabiliza las hélices del péptido de navidad y muestra muchos contactos con la molécula de RNA:
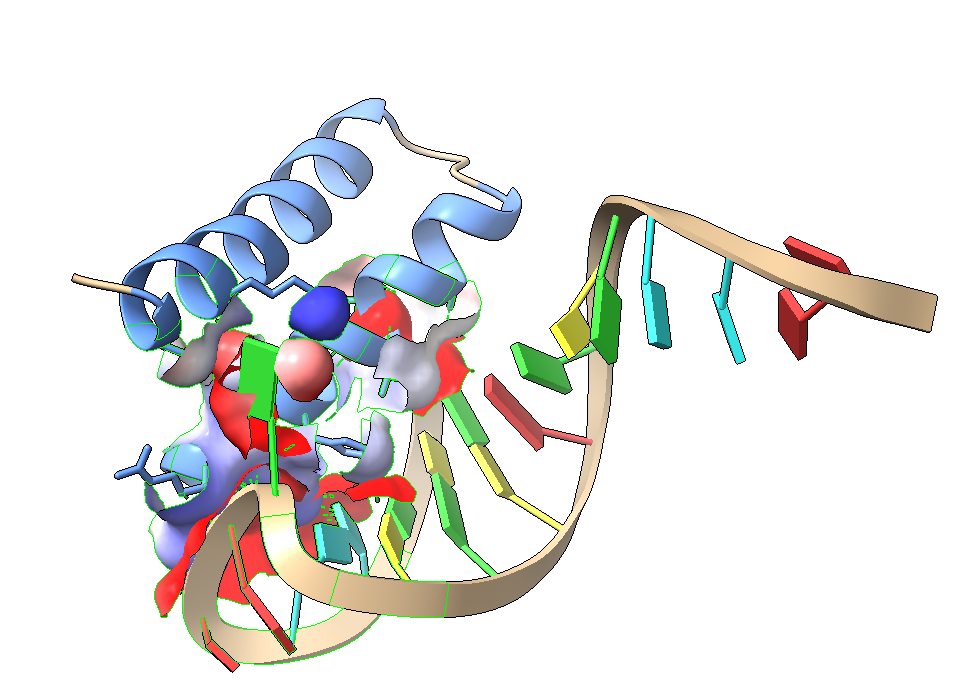
Aquí la vemos sin los contactos representados:
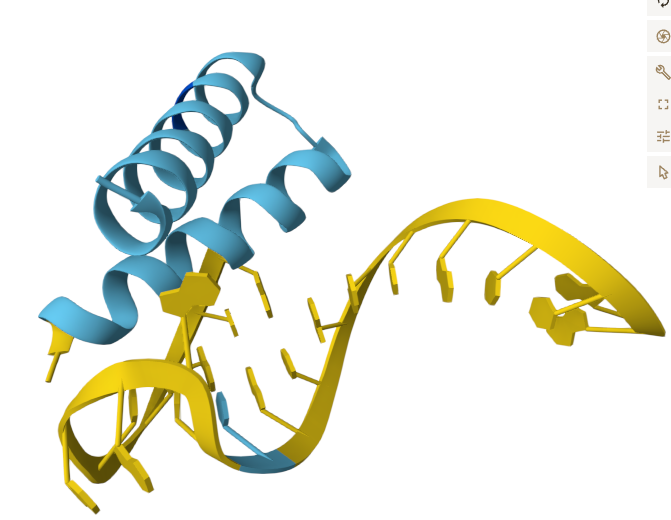
Las moléculas de ácidos nucleicos, como el RNA, tienen una gran capacidad para interaccionar con péptidos y proteínas. Esto fue y es clave en la evolución.
Los cofactores pueden dirigir el plegamiento. Es el caso del grupo hemo, un grupo prostético esencial en el plegamiento y función de proteínas como la hemoglobina y la mioglobina. En nuestro péptido de navidad, la interacción con hemo le conduce a un motivo completamente distinto, formando láminas beta:
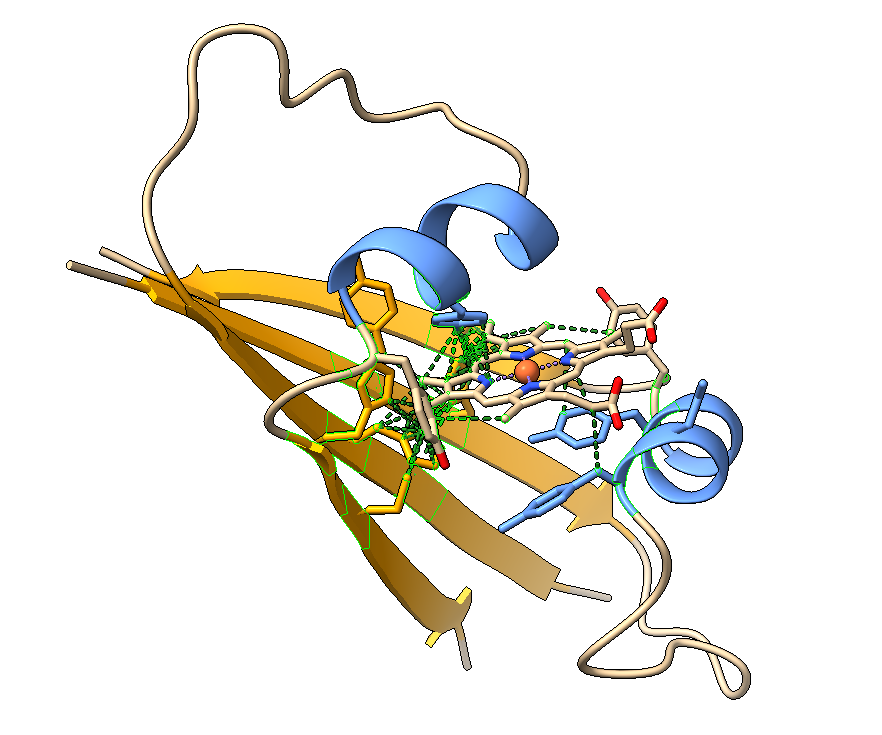
La interacción con hemo crea una estructura distinta, relativamente estable (probabilidad 70%). Un problema de esta estructura es que el átomo de hierro tiene libres las posiciones de coordinación encima y debajo del plano de hemo.
¿podrían darse estas estructuras en la realidad? Matemáticamente es posible, pero dada la complejidad de las interacciones reales, si la probabilidad de la predicción no es la más alta (>90%) lo más seguro es que un péptido generado al azar o sin tener en cuenta las reglas de los plegamientos, termine formando precipitados de agregados amorfos o amiloides. Pero, la única forma de saberlo es sintetizar el péptido y realizar los experimentos.
No obstante, para proteínas reales, es impresionante la capacidad de predicción de una IA como Alphafold. Es capaz de predecir las estructuras e interacciones y acertar plenamente, lo que la convierte en una valiosa ayuda para la investigación y la enseñanza.
Para las predicciones he usado Alphafold 2 y Alphafold 3 y para las representaciones he utilizado el software ChimeraX.



Súper interesante e ingeniosa felicitación de Navidad.
Y ya de paso me entero de algunas de las herramientas de IA que utilizáis en el laboratorio. Impresiona…
¡Enhorabuena César!
Gracias!