ATACANDO LA EPIGENÉTICA DEL CÁNCER: ROMIDEPSINA
Diego de León Oliva & Nicolás Martí Mencías
1. INTRODUCCIÓN
Desde que en 1971 Alfred Knudson propuso la hipótesis de que en la oncogénesis debían ocurrir mutaciones en el ADN, se ha invertido mucho esfuerzo en la investigación de la genética del cáncer. Pero igualmente importante es la epigenética de éste. Por eso, también salen al mercado medicamentos que tratan de restablecer el epigenoma normal, lo que induciría la muerte de las células malignas. Además, también pueden llevar a efectos sinergistas al combinarse con otros medicamentos. En este contexto, existen actualmente dos tipos de fármacos: los inhibidores de DNA-metiltransferasas y los inhibidores de desacetilasas de histonas (HDAC), y nosotros vamos a desarrollar un ejemplo de estos últimos: la romidepsina.
La romidepsina es un fármaco con actividad antineoplásica inhibidor de las desacetilasas de histonas (HDAC). En este post hablaremos de su naturaleza molecular, su mecanismo de acción y sus propiedades farmacológicas y clínicas. A grandes rasgos, la romidepsina es un depsipéptido bicíclico que interfiere en la expresión génica inhibiendo las HDAC de clase I y II, induciendo la detención del ciclo celular y apoptosis. Este medicamento también se conoce como FR901228 o FK228, y se comercializa como Istodax®.
La romidepsina fue aislada en 1993 como producto de fermentación de una cepa de Chromobacterium violaceum, en un programa de investigación japonés que buscaba compuestos bacterianos con propiedades antimicrobianas y antitumorales. El medicamento fue descubierto por Fujisawa Corporation y mostró grandes propiedades citotóxicas contra distintas células tumorales. La cepa se obtuvo de una muestra del suelo de la prefectura de Yamagata, en Japón (1,2). Posteriormente, el National Cancer Institute (NCI) de EEUU confirmó que era un potente anticancerígeno. Comenzaron los ensayos clínicos, y en 2009 la FDA aprobó el fármaco para el tratamiento contra el linfoma cutáneo de células T (CTCL), y en 2011 contra el linfoma periférico de células T (PTCL).
2. ESTRUCTURA
La romidepsina tiene estructura de depsipéptido bicíclico formado por la unión cíclica de cuatro aminoácidos y un ácido heptenoico con un grupo tiol, y presenta un puente disulfuro intramolecular. Un depsipéptido es aquel en el que algún enlace amida peptídico se reemplaza por un enlace tipo éster. Se encuentran mayoritariamente en productos naturales de origen marino y microbiano.
La romidepsina se compone de los aminoácidos L-Val, D-Val, Z-dehidrobutirina y D-Cys, y el ácido (3S,4E)-3-hidroxi-7-mercapto-4-heptenoico. Este ácido forma el puente disulfuro con la D-Cys y el enlace éster con la L-Val.
La estructura fue determinada usando una combinación de técnicas espectroscópicas, resonancia magnética nuclear y cristalografía por rayos X (3).
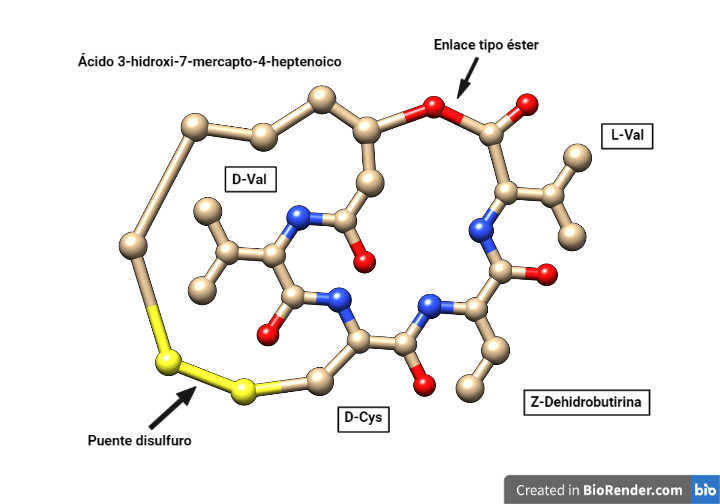
Figura 1. Estructura plana de la romidepsina. Estructura obtenida de PubChem y modificada con Chimera y Biorender
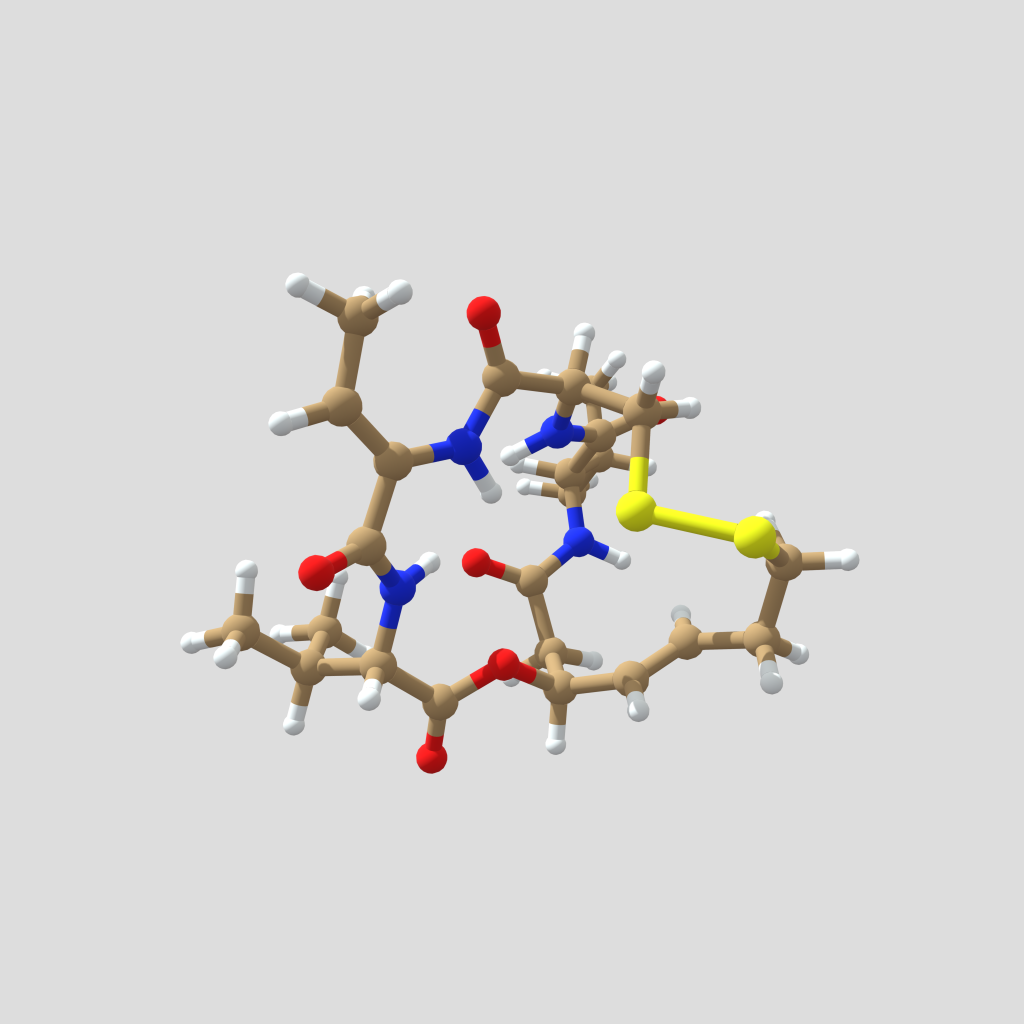
Figura 2. Modelo tridimensional de la romidepsina natural. Estructura obtenida de Drugbank. Creado con Sketchfab, clicar en la imagen para interactuar con el modelo.
3. EPIGENÉTICA
Los mecanismos epigenéticos son aquellos que regulan la expresión génica sin modificar la secuencia de bases nitrogenadas del DNA. Estos mecanismos se pueden clasificar en 3 grupos: metilación de bases, interferencia con RNA no codificante y modificaciones de la cromatina (4).
La metilación de bases consiste en la adición de un grupo metilo al carbono 5 de nucleótidos de citosina por acción de DNA metiltransferasas. Las citosinas metiladas son reconocidas por factores supresores de transcripción y se unen más difícilmente a los factores de transcripción, además de favorecer un mayor empaquetamiento y heterocromatinización del ADN; es decir, impiden la transcripción (5).
Por su parte, los RNAs no codificantes (ncRNAs)son moléculas de RNA que se transcriben de secuencias específicas de DNA pero no se traducen a proteínas y no intervienen de manera directa en su síntesis, a diferencia del mRNA, el rRNA o el tRNA. Hay varios tipos, aunque los más estudiados y relevantes son los miRNA, siRNA y lncRNA. Sus mecanismos de acción son variados, pudiendo destruir o silenciar moléculas de mRNA, favoreciendo la modificación de histonas, provocando cambios en el splicing, etc (4).
Finalmente, las modificaciones de la cromatina pueden deberse a cambios tanto en el DNA (por ejemplo, la metilación de citosinas) como en las histonas, aunque son mucho más habituales las modificaciones de este segundo tipo.
Estas modificaciones suelen tener como objetivo modificar la carga positiva que tienen las histonas, de modo que su relación con el DNA sea menos estrecha y que pueda acceder a él más fácilmente la maquinaria de transcripción, aunque hay algunos cambios que actúan a otros niveles.
Hay varios tipos de modificaciones, aunque los 3 más destacables son la metilación, la fosforilación y la acetilación de histonas, habitualmente de sus colas N-terminales.
Por su parte, la metilación de histonas ocurre por acción de metiltransferasas de histonas específicas y no tiene un efecto único en la transcripción: puede tanto favorecer como inhibir la expresión génica. Esto es así porque las metilaciones no modifican la carga de las histonas, afectando directamente la relación entre el DNA y ellas, sino que las histonas metiladas cambian su conformación espacial, dando lugar a estructuras secundarias de la cromatina que atraen dominios proteicos específicos pertenecientes a proteínas implicadas en la transcripción (6).
Por otro lado, la fosforilación de histonas se lleva a cabo mediante la intervención de kinasas. La adición de ácidos fosfóricos aporta cargas negativas a la cola de la histona, lo que reduce su afinidad por el DNA, también cargado negativamente, y favorece la accesibilidad a la secuencia de nucleótidos por parte de la maquinaria de transcripción (6).
La acetilación y desacetilación de histonas (concretamente la desacetilación) son los procesos epigenéticos que la romidepsina tiene como dianas, y que explicaremos más en profundidad. Este proceso está regulado por dos familias de enzimas: las acetiltransferasas de histonas (HATs) y las desacetilasas de histonas (HDACs). Las HATs toman el grupo acetilo de un acetil-CoA y lo transfieren a la cadena lateral de una lisina, anulando su carga positiva y reduciendo su afinidad por el DNA.
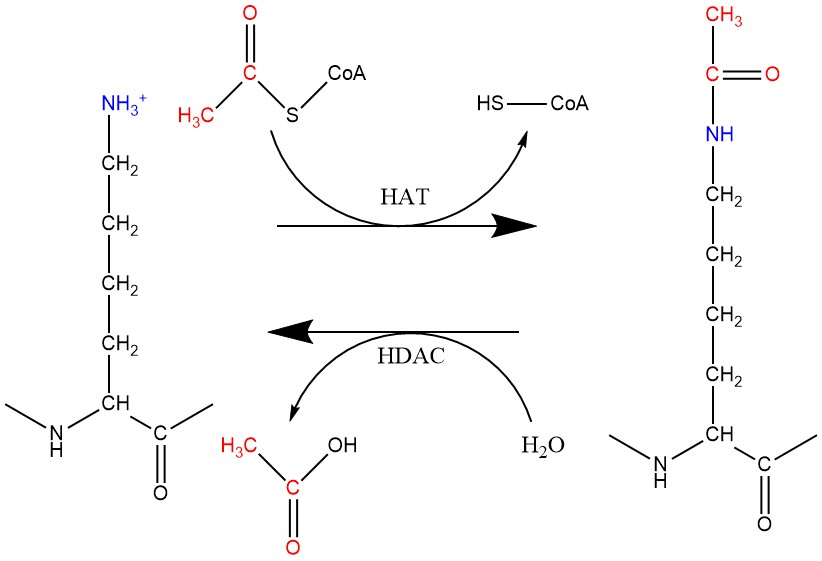
Figura 3. Reacciones de acetilación y desacetilación de lisinas llevadas a cabo por las enzimas HAT y HDAC, respectivamente. Creado con ChemDraw.
La desacetilación de histonas está a cargo de las HDACs, que toman el grupo acetilo que se había agregado a las lisinas y lo retiran, devolviéndole a la histona su carga positiva y permitiendo una unión más fuerte con el DNA.
En cuanto a la naturaleza de las HDAC, existen un total de 18 tipos diferentes en mamíferos, los cuales se agrupan en 4 clases: I, II, III y IV. Las dianas principales de la romidepsina son la HDAC1 y la HDAC2, ambas dentro de la clase I, y también tiene cierto efecto sobre las HDAC de clase II.
Figura 4. Modelo tridimensional de HDAC1 con un grupo acetato en su centro activo (PDB: 4BKX, Chain B). Estructura modificada con Chimera y creada con Sketchfab, clicar en la imagen para interactuar con el modelo.
El centro catalítico de las HDAC clase I lo forman un catión Zn2+, tres residuos de histidina, un residuo de tirosina y dos de aspartato. La tirosina forma un puente de hidrógeno con el oxígeno del grupo acetato a eliminar, mientras que una molécula de agua se une a las histidinas y ataca el enlace entre el grupo acetilo y el grupo amino de la cadena lateral de la lisina, formándose un intermediario de reacción estabilizado por el catión Zn2+ y, finalmente, rompiéndose el enlace amida para dar lugar a una lisina por un lado y a un grupo acetato libre por el otro (4, 6).

Figura 5. Modelo tridimensional de la superficie y del Zn pocket de la HDAC1 (PDB: 4BKX, Chain B). Estructura modificada con Chimera y creada con Sketchfab, clicar en la imagen para interactuar con el modelo.
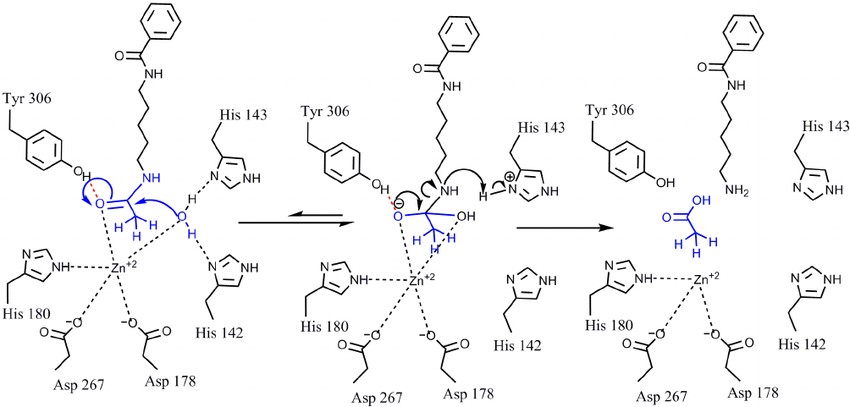
Figura 6. Mecanismo de acción de las HDAC, desacetilación del fármaco AHA por la HDAC8. Imagen tomada de Bonomi, R., Mukhopadhyay, U., Shavrin, A., Yeh, H. H., Majhi, A., Dewage, S. W., Najjar, A., Lu, X., Cisneros, G. A., Tong, W. P., Alauddin, M. M., Liu, R. S., Mangner, T. J., Turkman, N., & Gelovani, J. G. (2015). Novel Histone Deacetylase Class IIa Selective Substrate Radiotracers for PET Imaging of Epigenetic Regulation in the Brain. PloS one, 10(8), e0133512.
Cabe destacar que tan solo un 2-5% del genoma se encuentra regulado por la acetilación/desacetilación de histonas, dentro del cual encontramos genes muy importantes reguladores del ciclo celular o factores de apoptosis. Es por esto por lo que se convierte las HDAC en un target farmacológico muy interesante.
4. MECANISMO DE ACCIÓN
A rasgos generales, la romidepsina es un inhibidor de HDACs, lo que favorece que aumente el número de proteínas acetiladas, incluyendo proteínas histónicas y no histónicas. Al permanecer acetiladas las histonas, se mantiene una estructura de la cromatina más laxa y activa transcripcionalmente. Además, muchas proteínas citoplásmicas y nucleares también permanecen acetiladas, aunque se desconoce cómo afecta a la célula.
La romidepsina natural es un profármaco, y al entrar en las células el glutatión (GSH) reduce el puente disulfuro, dando un péptido monocíclico con dos grupos sulfhidrilo. El sulfhidrilo unido a la cadena de cuatro carbonos es capaz de introducirse en el sitio activo y quelar el Zn2+, lo que inhibe a la enzima de manera que ya no se puede unir el sustrato (acetato de Lys en colas de histonas) (7). La figura 7 es un estudio de modelado computacional que representa la interacción del fármaco con la enzima (8).
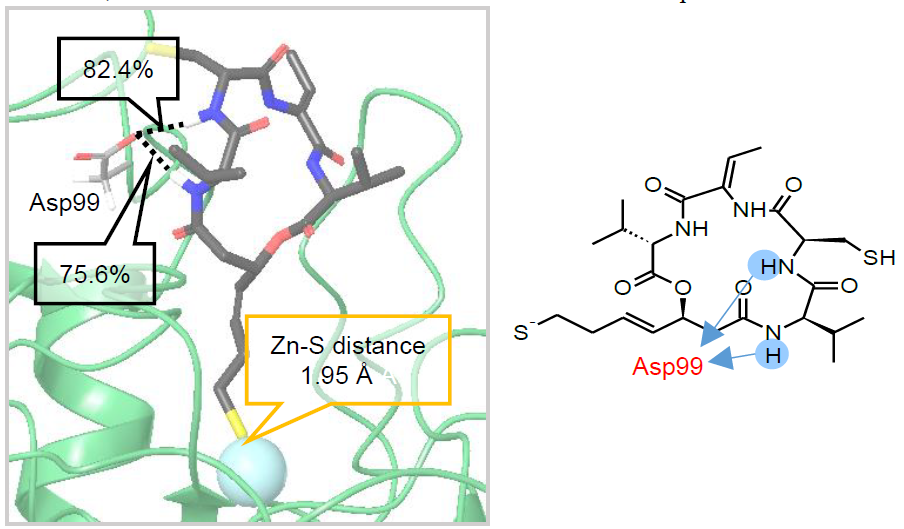
Figura 7. Interacciones entre romidepsina y HDAC1. Imagen tomada de Oda, A., Kato, K., Morino, M., Nakayoshi, T., Fukuyoshi, S., Saijo, K., Ishioka, C., & Kurimoto, E. (2018). Prediction of the three-dimensional structures of histone deacetylase 1 complexed with Romidepsin and FK-A5. Journal of Physics: Conference Series, 1136, 012019.
Como no hemos encontrado en el Protein Data Bank ninguna HDAC acomplejada con romidepsina, hemos seleccionado con fines didácticos una en la que se inhibe por Vorinostat o SAHA, otro inhibidor de HDAC que muestra un mecanismo parecido, y también aprobado frente al CTCL.
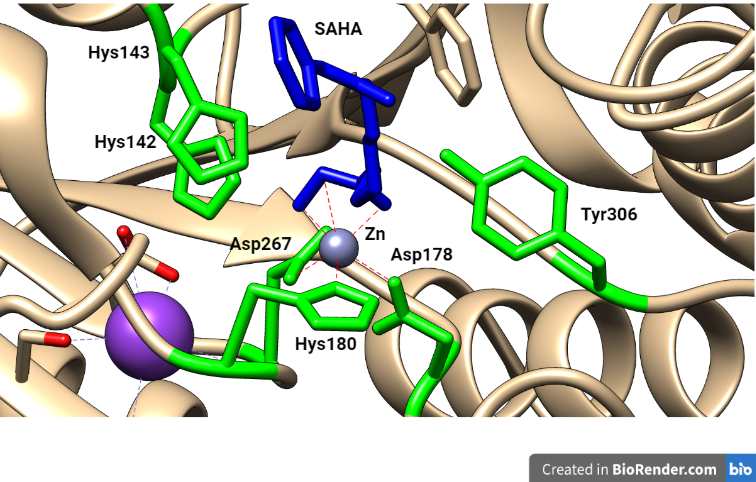
Figura 8. Centro activo de HDAC acomplejado con SAHA: en verde se muestran los aminoácidos del centro activo y en azul el inhibidor unido al Zn2+. Estructura modificada con Chimera (PDB:1ZZ1)
Figura 9. Modelo tridimensional de un homólogo bacteriano de HDAC inhibido con SAHA (PDB: 1ZZ1). Estructura modificada con Chimera y creada con Sketchfab, clicar en la imagen para interactuar con el modelo.
Inicialmente se pensó que el grupo sulfhidrilo se unía covalentemente a una cisteína del sitio activo (Cys151 en HDAC1), pero un experimento en el que se sustituyó esta por una serina puso de manifiesto que seguía existiendo inhibición, aunque era necesaria una concentración mayor. Por tanto, sumado al hecho de que la inhibición es reversible, se piensa que esta cisteína tiene un papel regulador en la afinidad por la romidepsina (9).
La efectividad del tratamiento epigenético se cree que se debe a la hipótesis de la “vulnerabilidad epigenética de las células cancerosas”, propuesta por Dawson y Korazides (10). Esta nos dice que mientras las células normales tienen múltiples mecanismos epigenéticos, las células tumorales dependen de unos pocos, que al fallar (inhibición de HDAC por ejemplo), llevan a catástrofe celular. Se basa en la observación de que las células normales permanecen inalteradas por estos inhibidores epigenéticos, al contrario que las cancerosas. Además, en el desarrollo de tumores, muchas veces las HDACs se encuentran sobreexpresadas, inhibiendo la expresión de genes reguladores del ciclo y supresores de tumores. Por ello, son actualmente un target farmacológico en investigación para el diseño de inhibidores (HDACi), dando lugar a este nuevo tipo de quimioterapia. Por tanto, lo fundamental es conseguir restablecer la expresión de proteínas antitumorales que llevan a la célula maligna a la muerte.
La romidepsina ejerce múltiples efectos encaminados a disminuir la población tumoral: relajación de cromatina, aumento de la transcripción, interferencia con la función de chaperonas, generación de ROS (especies reactivas de oxígeno), daños en el DNA, aumento de inhibidores endógenos del ciclo celular y promover apoptosis, tanto por vía extrínseca como intrínseca.
La romidepsina consigue la detención del ciclo celular y apoptosis principalmente, pero también inhibición de angiogénesis, inducción de autofagia y diferenciación. Los mecanismos que llevan a la célula cancerosa hacia la muerte son múltiples y variados, y dependen del tipo celular y la dosis. La mayoría de estos mecanismo se desconocen a día de hoy (7, 11).
Apoptosis: se ha visto que puede estar mediada por daños en el DNA, aumento de ROS, expresión de factores proapoptóticos, disminución de factores de supervivencia, aumento de la permeabilidad de la membrana mitocondrial y por interacciones con receptores de señales de muerte. Según el tipo de cáncer aparecen unas u otras vías implicadas. Por ejemplo, en líneas de células T malignas, se ha visto in vitro la apoptosis relacionada con ROS y daño en el DNA, activación de vías de señalización de estrés SAPK/JNK (pertenecen a superfamilia de MAPK) y UPR (respuesta a proteínas desplegadas) e inhibición de las vías PI3K-AKT-mTOR y Wnt/β-cateninas, ambas relacionadas con la progresión del ciclo (12).
Detención del ciclo celular: está relacionada con la inducción de p21 y p53. p21 o CDKN1A (cyclin dependent kinase inhibitor) inhibe las ciclinas dependientes de kinasa (CDK) y defosforila la proteína de retinoblastoma, lo que detiene el ciclo en G1. Su expresión está regulada por p53, que compite con las HDAC en el promotor de p21, induciendo su expresión. Este proceso se ve favorecido gracias a la inhibición de las HDAC. Además, la acetilación de p53 prolonga su vida media, estimulando aún más el proceso (11).
En cuanto a la generación de ROS parece que se debe a la reacción de los grupos sulfhidrilos de la romidepsina con oxígeno, dando lugar al mismo puente disulfuro y anión superóxido. La romidepsina se volvería a reducir por el GSH para pasar a su forma activa e inhibir a la HDAC o volver a reaccionar con oxígeno. Por tanto, la romidepsina es un fármaco inhibidor de HDAC, pero también productor de ROS y consumidor de GSH celular. Debido a esto último, la célula tumoral se vuelve más vulnerable a los fármacos y radicales libres, en especial, aquellas que presentan quimiorresistencias mediadas por GSH. Las ROS son inductores de daño en el ADN, desnaturalización de proteínas y permeabilización de la membrana mitocondrial, lo que supone el inicio de la vía intrínseca de la apoptosis (9, 13).
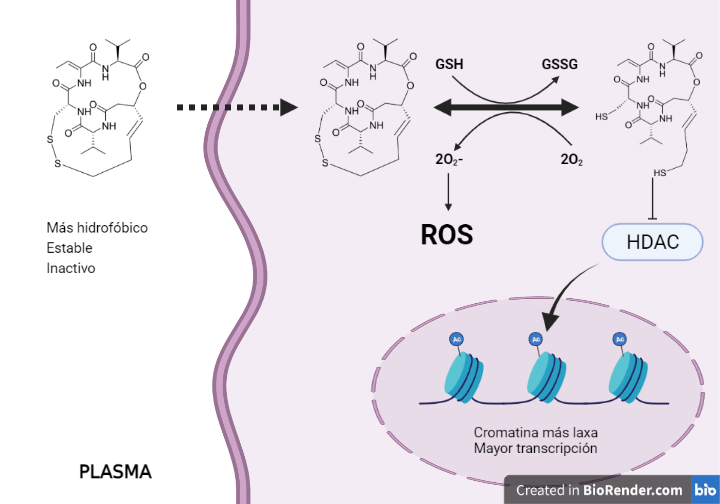
Figura 10. Relación de la romidepsina con GSH y ROS. Estructura de la romidepsina obtenida de Drugbank y modificada con Chemdraw.
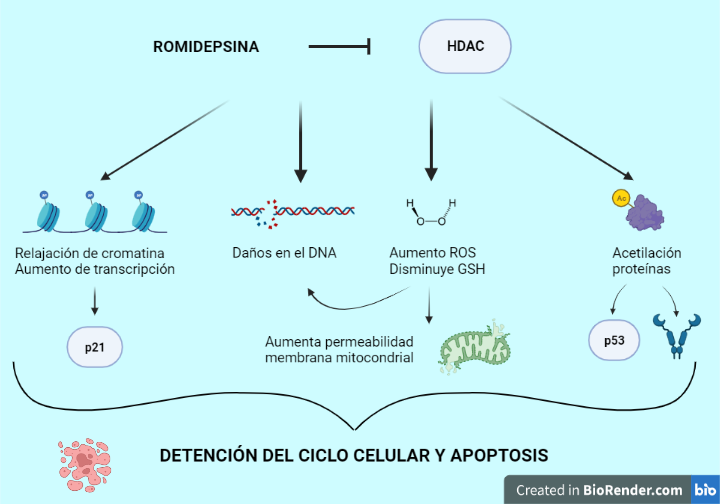
Figura 11. Efectos de la romidepsina. Se pueden observar algunos de los múltiples efectos del fármaco en las células tumorales. El conjunto de todas las vías activadas y desactivadas conducen a la célula hacia la detención del ciclo celular y apoptosis
5. USO CLÍNICO CONTRA LINFOMAS
La romidepsina se puede utilizar como tratamiento para dos tipos de cánceres, el linfoma cutáneo de células T (CTCL) y el linfoma periférico de células T (PTCL). Ambos son tipos de linfoma no Hodgkin (NHL), es decir, cánceres en los que proliferan y se malignizan linfocitos, en este caso linfocitos T (14).
Los CTCLs suponen un 4% de los NHLs y se dividen en varios tipos, dentro de los cuáles los principales son la micosis fungoide (más indolente) y el síndrome de Sézary (más agresivo), ambos caracterizados porque los linfocitos T malignos se concentran en la piel, aunque en el síndrome de Sézary también es habitual encontrarlos en la sangre (15, 22). Por su parte, los PTCLs son aproximadamente 10% de NHLs, son mucho más agresivos que los CTCLs y las células T cancerosas se encuentran principalmente en órganos linfoides secundarios, como los ganglios linfáticos o el bazo (16, 23).

Figura 12. Piel de un paciente de micosis fungoide. Imagen tomada de Ram-Wolff, C. (2014). Linfomas T Cutáneos de Tipo micosis fungoide/Síndrome de Sézary (incluida parapsoriasis). EMC – Dermatología, 48(2), 1–12. https://doi.org/10.1016/s1761-2896(14)67581-6
En 2009, la FDA (la Administración de Medicamentos y Alimentos de EEUU) aprobó el uso de la romidepsina como tratamiento para pacientes de CTCL y en 2011 para pacientes de PTCL que ya hayan recibido al menos una terapia previa. Sin embargo, hay estudios recientes que cuestionan su eficacia como tratamiento para los PCTLs, llevando a algunas compañías farmacéuticas a dejar de indicarla como tratamiento para estos cánceres (17).
Se administra de manera intravenosa, idealmente perfundiendo durante 4 horas una dosis total de 14mg/m2 los días 1, 8 y 15 de un ciclo de 28 días. Tras el final de cada ciclo se inicia uno nuevo, pudiendo modificarse la cantidad de dosis o los días de administración dependiendo de la respuesta del paciente al tratamiento. La romidepsina es transportada por la sangre unida a proteínas hasta llegar a sus tejidos diana, hasta que finalmente es metabolizada a nivel hepático por enzimas de la familia citocromo P450 (14, 24).
En cuanto a los efectos secundarios, puede producir algunos leves, como náuseas o vómitos, y otros algo más graves, como linfopenia, anemia o trombocitopenia, que pueden llegar a afectar hasta al 40% de los tratados (14, 24).
6. OTRAS APLICACIONES
Aparte de su eficacia contra los CTCLs y PTCLs, la romidepsina también parece tener cierta actividad antineoplásica en otros cánceres, aunque su uso clínico no está todavía aprobado. Algunos de los cánceres que parece que responderían a un tratamiento con romidepsina serían el cáncer de pulmón de células no pequeñas al combinarse con bortezomib (inhibidor del proteosoma 26S) (18), el cáncer de mama inflamatorio al combinarse con paclitaxel (inhibidor de polimerización de microtúbulos) (19) y ciertos tipos de cáncer de ovario al combinarse con inhibidores de las ciclooxigenasas, como la aspirina (20). Parece que las posibilidades de la romidepsina con otros fármacos son infinitas.
Por otro lado, también se tiene en cuenta a la romidepsina como un posible tratamiento contra el SIDA. Esto se debe a que la romidepsina es un agente reversor de la latencia, por lo que activa a los virus que estaban en estado latente, ocultos dentro de los linfocitos, y los expone a tratamientos antivirales o al sistema inmunitario, que debe haber sido previamente estimulado con una vacuna (21).
Para finalizar, nos gustaría lanzar una pregunta: ¿por qué la romidepsina es más eficaz en cánceres de sangre que en tumores sólidos? ¿Cuál es la diferencia fundamental para que uno sea mucho más susceptible que el otro a las modificaciones epigenéticas? La complejidad genética de los tumores sólidos o el medio en el que se encuentran expuestos al fármaco son algunas diferencias que podrían influir, pero aún se desconoce mucho de esta terapia. Por eso, “further research is needed!”.
BIBILIOGRAFÍA
- Ueda, H., Nakajima, H., Hori, Y., Fujita, T., Nishimura, M., Goto, T., & Okuhara, M. (1994). FR901228, a novel antitumor bicyclic depsipeptide produced by Chromobacterium violaceum No. 968. I. Taxonomy, fermentation, isolation, physico-chemical and biological properties, and antitumor activity. The Journal of antibiotics, 47(3), 301–310. https://doi.org/10.7164/antibiotics.47.301
- Ueda, H., Manda, T., Matsumoto, S., Mukumoto, S., Nishigaki, F., Kawamura, I., & Shimomura, K. (1994). FR901228, a novel antitumor bicyclic depsipeptide produced by Chromobacterium violaceum No. 968. III. Antitumor activities on experimental tumors in mice. The Journal of antibiotics, 47(3), 315–323. https://doi.org/10.7164/antibiotics.47.315
- Shigematsu, N., Ueda, H., Takase, S., Tanaka, H., Yamamoto, K., & Tada, T. (1994). FR901228, a novel antitumor bicyclic depsipeptide produced by Chromobacterium violaceum No. 968. II. Structure determination. The Journal of antibiotics, 47(3), 311–314. https://doi.org/10.7164/antibiotics.47.311
- Al Aboud, N. M., Tupper, C., & Jialal, I. (2021). Genetics, Epigenetic Mechanism. In StatPearls. StatPearls Publishing.
- Moore, L. D., Le, T., & Fan, G. (2013). DNA methylation and its basic function. Neuropsychopharmacology : official publication of the American College of Neuropsychopharmacology, 38(1), 23–38. https://doi.org/10.1038/npp.2012.112
- Bannister, A. J., & Kouzarides, T. (2011). Regulation of chromatin by histone modifications. Cell research, 21(3), 381–395. https://doi.org/10.1038/cr.2011.22
- VanderMolen, K. M., McCulloch, W., Pearce, C. J., & Oberlies, N. H. (2011). Romidepsin (Istodax, NSC 630176, FR901228, FK228, depsipeptide): a natural product recently approved for cutaneous T-cell lymphoma. The Journal of antibiotics, 64(8), 525–531. https://doi.org/10.1038/ja.2011.35
- Oda, A., Kato, K., Morino, M., Nakayoshi, T., Fukuyoshi, S., Saijo, K., Ishioka, C., & Kurimoto, E. (2018). Prediction of the three-dimensional structures of histone deacetylase 1 complexed with Romidepsin and FK-A5. Journal of Physics: Conference Series, 1136, 012019. https://doi.org/10.1088/1742-6596/1136/1/012019
- Furumai, R., Matsuyama, A., Kobashi, N., Lee, K. H., Nishiyama, M., Nakajima, H., Tanaka, A., Komatsu, Y., Nishino, N., Yoshida, M., & Horinouchi, S. (2002). FK228 (depsipeptide) as a natural prodrug that inhibits class I histone deacetylases. Cancer research, 62(17), 4916–4921.
- Dawson, M. A., & Kouzarides, T. (2012). Cancer epigenetics: from mechanism to therapy. Cell, 150(1), 12–27. https://doi.org/10.1016/j.cell.2012.06.013
- Eckschlager, T., Plch, J., Stiborova, M., & Hrabeta, J. (2017). Histone Deacetylase Inhibitors as Anticancer Drugs. International journal of molecular sciences, 18(7), 1414. https://doi.org/10.3390/ijms18071414
- Valdez, B. C., Brammer, J. E., Li, Y., Murray, D., Liu, Y., Hosing, C., Nieto, Y., Champlin, R. E., & Andersson, B. S. (2015). Romidepsin targets multiple survival signaling pathways in malignant T cells. Blood cancer journal, 5(10), e357. https://doi.org/10.1038/bcj.2015.83
- Mizutani, H., Hiraku, Y., Tada-Oikawa, S., Murata, M., Ikemura, K., Iwamoto, T., Kagawa, Y., Okuda, M., & Kawanishi, S. (2010). Romidepsin (FK228), a potent histone deacetylase inhibitor, induces apoptosis through the generation of hydrogen peroxide. Cancer science, 101(10), 2214–2219. https://doi.org/10.1111/j.1349-7006.2010.01645.x
- Yang L. P. (2011). Romidepsin: in the treatment of T-cell lymphoma. Drugs, 71(11), 1469–1480. https://doi.org/10.2165/11207170-000000000-00000
- Bagherani, N., & Smoller, B. R. (2016). An overview of cutaneous T cell lymphomas. F1000Research, 5, F1000 Faculty Rev-1882. https://doi.org/10.12688/f1000research.8829.1
- Xie, C., Li, X., Zeng, H., & Qian, W. (2020). Molecular insights into pathogenesis and targeted therapy of peripheral T cell lymphoma. Experimental hematology & oncology, 9(1), 30. https://doi.org/10.1186/s40164-020-00188-w
- Bachy, E., Camus, V., Thieblemont, C., Sibon, D., Casasnovas, R. O., Ysebaert, L., Damaj, G., Guidez, S., Pica, G. M., Kim, W. S., Lim, S. T., André, M., García-Sancho, A. M., Penarrubia, M. J., Staber, P. B., Trotman, J., Hüttmann, A., Stefoni, V., Re, A., Gaulard, P., … Delarue, R. (2021). Romidepsin Plus CHOP Versus CHOP in Patients With Previously Untreated Peripheral T-Cell Lymphoma: Results of the Ro-CHOP Phase III Study (Conducted by LYSA). Journal of clinical oncology : official journal of the American Society of Clinical Oncology, JCO2101815. Advance online publication. https://doi.org/10.1200/JCO.21.01815
- Schrump, D. S., Fischette, M. R., Nguyen, D. M., Zhao, M., Li, X., Kunst, T. F., Hancox, A., Hong, J. A., Chen, G. A., Kruchin, E., Wright, J. J., Rosing, D. R., Sparreboom, A., Figg, W. D., & Steinberg, S. M. (2008). Clinical and molecular responses in lung cancer patients receiving Romidepsin. Clinical cancer research : an official journal of the American Association for Cancer Research, 14(1), 188–198. https://doi.org/10.1158/1078-0432.CCR-07-0135
- Robertson, F. M., Chu, K., Boley, K. M., Ye, Z., Liu, H., Wright, M. C., Moraes, R., Zhang, X., Green, T. L., Barsky, S. H., Heise, C., & Cristofanilli, M. (2013). The class I HDAC inhibitor Romidepsin targets inflammatory breast cancer tumor emboli and synergizes with paclitaxel to inhibit metastasis. Journal of experimental therapeutics & oncology, 10(3), 219–233.
- Son, D. S., Wilson, A. J., Parl, A. K., & Khabele, D. (2010). The effects of the histone deacetylase inhibitor romidepsin (FK228) are enhanced by aspirin (ASA) in COX-1 positive ovarian cancer cells through augmentation of p21. Cancer biology & therapy, 9(11), 928–935. https://doi.org/10.4161/cbt.9.11.11873
- Mothe, B., Rosás-Umbert, M., Coll, P., Manzardo, C., Puertas, M. C., Morón-López, S., Llano, A., Miranda, C., Cedeño, S., López, M., Alarcón-Soto, Y., Melis, G. G., Langohr, K., Barriocanal, A. M., Toro, J., Ruiz, I., Rovira, C., Carrillo, A., Meulbroek, M., Crook, A., … BCN02 Study Investigators (2020). HIVconsv Vaccines and Romidepsin in Early-Treated HIV-1-Infected Individuals: Safety, Immunogenicity and Effect on the Viral Reservoir (Study BCN02). Frontiers in immunology, 11, 823. https://doi.org/10.3389/fimmu.2020.00823
- https://es.oncolink.org/tipos-de-cancer/linfomas/linfoma-cutaneo-de-celulas-t/all-about-cutaneous-t-cell-lymphoma-ctcl
- https://lymphoma.org/aboutlymphoma/nhl/ptcl/
- https://packageinserts.bms.com/pi/pi_istodax.pdf
- https://go.drugbank.com/drugs/DB06176
- https://pubchem.ncbi.nlm.nih.gov/compound/5352062


 Entrada siguiente
Entrada siguiente