Iniciación de la replicación
DnaA en procariotas
La replicación del DNA genómico en bacterias comienza con la unión de moléculas de la proteína DnaA a una serie de secuencias específicas llamadas DnaA box o cajas DnaA; la unión de DnaA a las cajas da lugar a la formación de una estructura nucleoproteica que contiene de 20 a 30 monómeros de la proteína DnaA (en E. coli). La formación del complejo nucleoproteico induce la fusión de una región próxima rica en pares AT, abriendo la burbuja de replicación, a la que se unirán la helicasa DnaB y otros factores, para formar el primosoma.
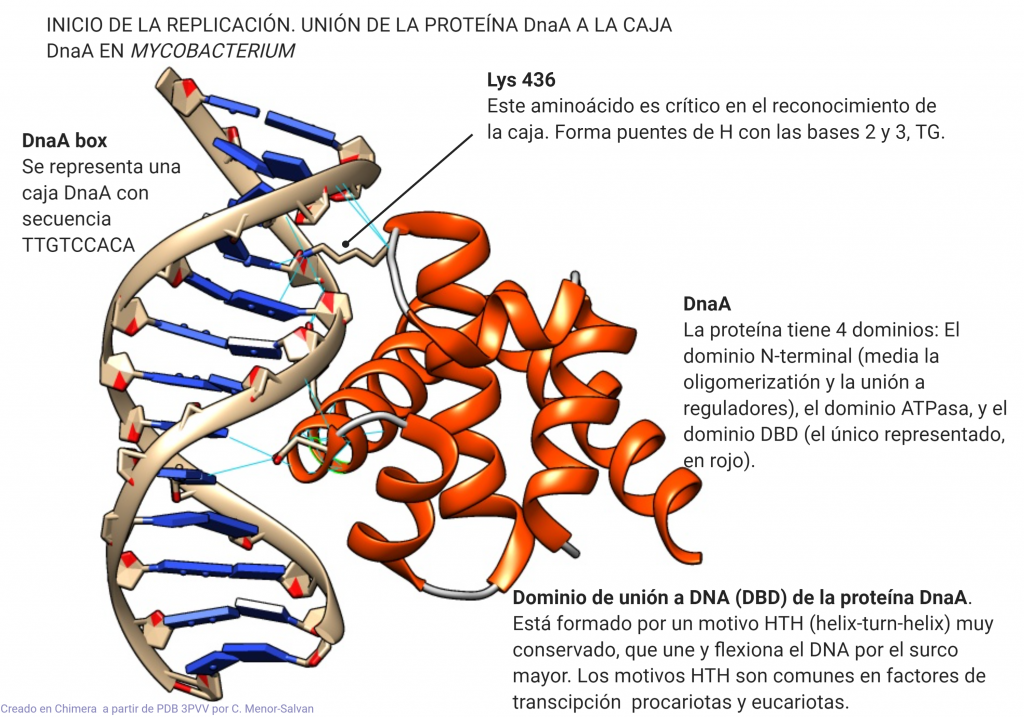
¿cómo reconoce específicamente la proteína DnaA a la caja a la que se va a unir? la clave la tiene una secuencia de 9 bp en la caja.
El dominio de unión a DNA está formado por un motivo de hélices alfa característico llamado «hélice-giro-hélice» (HTH). Este motivo se presenta comúnmente en proteínas de unión a DNA, principalmente factores de transcripción, tanto en procariotas como eucariotas. El motivo HTH interacciona fácilmente con la doble hélice de DNA, accediendo a las bases por el surco mayor. La clave del reconocimiento de la caja está en la secuencia de aminoácidos del motivo HTH: una serie de aminoácidos específica forma puentes de hidrógeno e interacciones hidrófobas con las 9 bases implicadas en el reconocimiento de la caja.
En cualquier otra región del DNA, la proteína interacciona con muy baja o ninguna afinidad, debido a que no se forman suficientes puentes. En cambio con la secuencia de la caja DnaA, la unión es muy estable y muestra muy alta afinidad. En la figura anterior se muestran una de las interacciones clave: un residuo de lisina forma puentes de hidrógeno con un par TG altamente conservado en la secuencia de la caja. Una vez que la DnaA ha reconocido la secuencia de la caja y se ha unido a ella, se forma el agregado nucleoproteico que conduce a la fusión de la región rica en AT, promoviendo el siguiente paso de la replicación.
Taq DNA polimerasa de Thermus aquaticus
Taq polymerase by cmenorsalvan on Sketchfab
DNA polimerasas dependientes de DNA (DdDP)
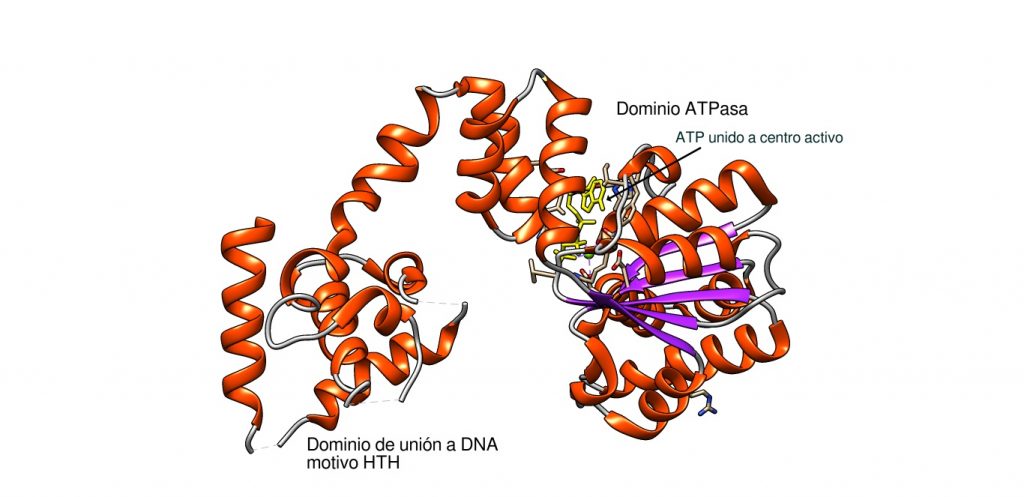
Este tipo de polimerasas, responsables de la replicación y procesos de reparación en procariotas y eucariotas, se denominan «polimerasas de mano derecha» o right handed polymerases. Son metaloproteínas dependientes de zinc y magnesio y ésta denominación se debe a la analogía (con cierta imaginación) de la estructura con una mano derecha abierta:
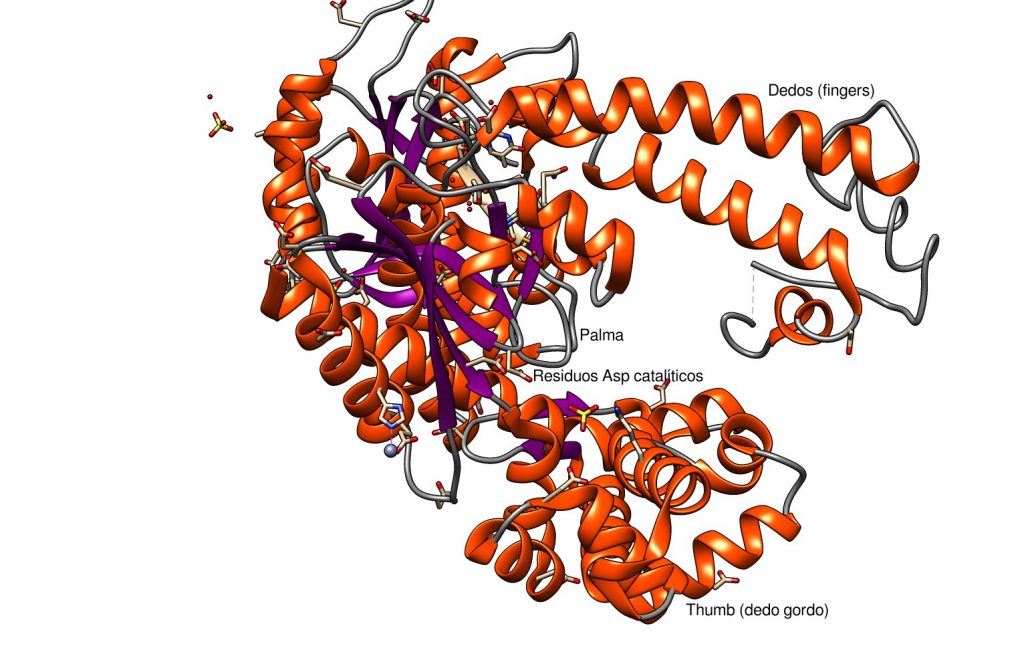
Veamos la estructura en 3D:
DNA pol I (Klenow). E. coli. by cmenorsalvan on Sketchfab
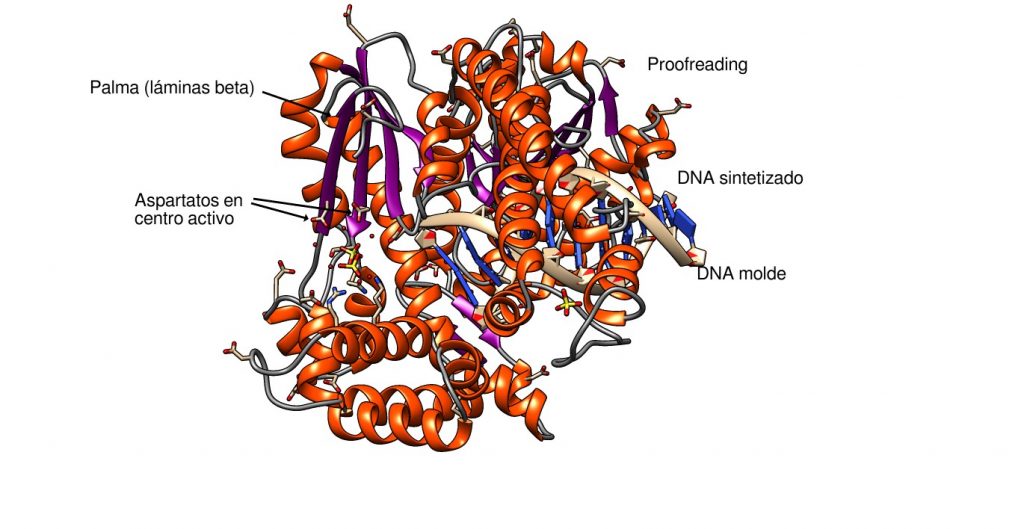
Ocultando las hélices alfa, se puede visualizar mejor la posición relativa del DNA respecto al centro catalítico, la palma de láminas beta. Esta estructura está altamente conservada en la evolución.
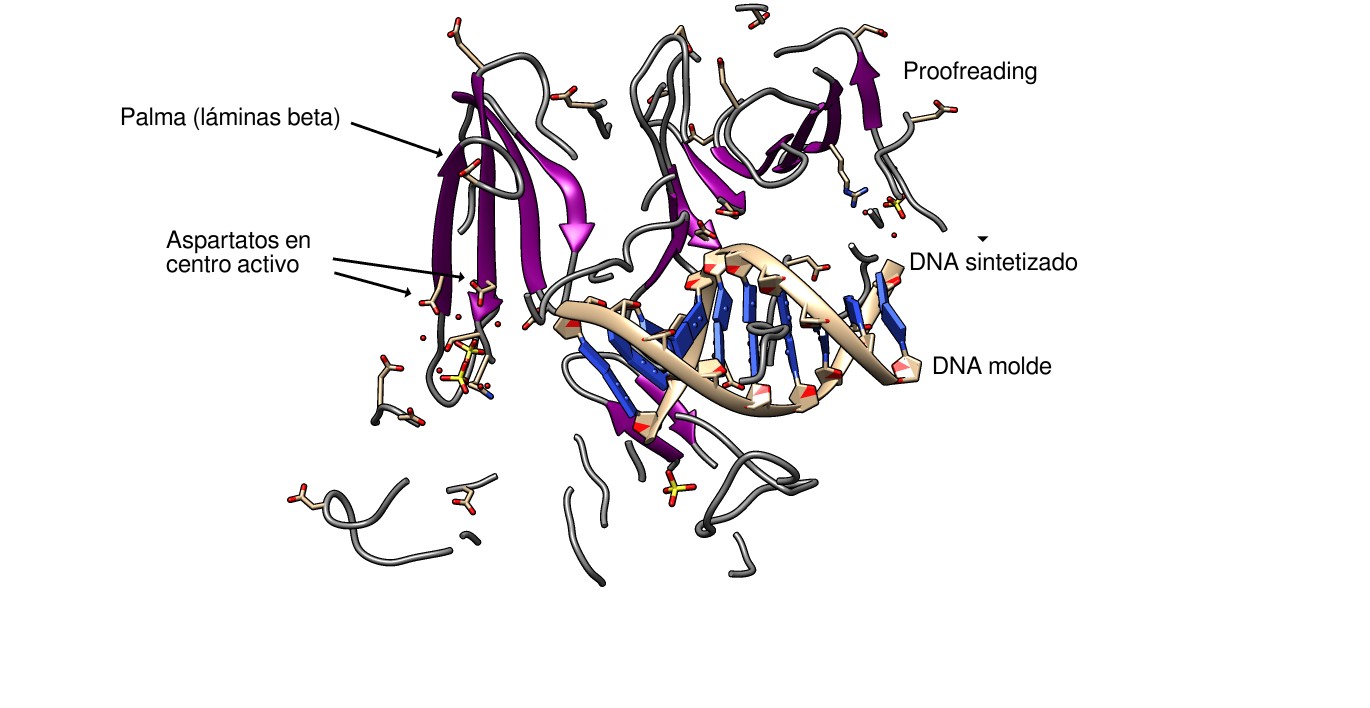
El alineamiento estructural es una técnica que pone de manifiesto la relación entre la conservación de la estructura y su función. Es el resultado de la conexión evolutiva entre proteínas en diferentes especies o entre diferentes proteínas que derivan de un origen común. Por ejemplo, la DNA polimerasa I de E. Coli y la Taq polimerasa del organismo termófilo Thermus aquaticus, tienen una estructura equivalente y perfectamente superponible en su dominio polimerasa y proofreading. El dominio proofreading de la Taq polimerasa tiene un cambio estructural, visible en el siguiente modelo 3d, que lo hace inactivo. En el modelo es evidente, por ejemplo, la coincidencia espacial de los residuos aspárticos en el centro activo.
DNA pol I vs Taq pol by cmenorsalvan on Sketchfab
Referencias
Erzberger, J. P., Mott, M. L., & Berger, J. M. (2006). Structural basis for ATP-dependent DnaA assembly and replication-origin remodeling. Nature Structural and Molecular Biology, 13(8), 676–683. https://doi.org/10.1038/nsmb1115
Tsodikov, O. V., & Biswas, T. (2011). Structural and thermodynamic signatures of DNA recognition by mycobacterium tuberculosis DnaA. Journal of Molecular Biology, 410(3), 461–476. https://doi.org/10.1016/j.jmb.2011.05.007