El sistema de ubiquitinas y la eliminación de proteínas
Ignacio Moratilla Rivera y Miryam Muñoz Martín
3º Biología Sanitaria
Introducción:
Si pensamos en degradación de materiales en la célula en general, siempre nos viene a la cabeza los lisosomas como máximo exponente de esta función. No vamos a negar su imprescindible papel en las células, pero tampoco es el único responsable de realizar estas funciones.
Existe otro sistema que es el responsable de la degradación de proteínas citosólicas contribuyendo a la proteostasis celular. Este sistema está constituido por unas pequeñas proteínas denominadas ubiquitinas y unos complejos proteicos conocidos como proteosomas. Ambos junto a otras enzimas constituyen un proceso en cadena en el que finalmente las proteínas serán degradas.
Este mecanismo no solo se va a limitar a la eliminación de proteínas mal plegadas o aberrantes, sino que también va a desempeñar un papel crucial en la regulación del ciclo celular y fallos en él pueden llevar a cataclismos fisiológicos serios como el Parkinson, Alzheimer o enfermedades cardiovasculares.
Ubiquitinas: las proteínas omnipresentes.
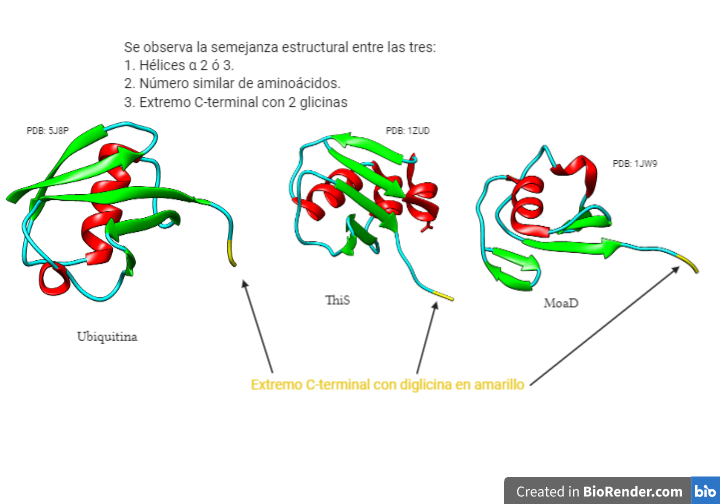
Las ubiquitinas son unas proteínas monoméricas que se encuentra presenten en todos los organismos eucariotas (de ahí su nombre, ya que son ubicuas). No se han encontrado en procariotas, pero tienen proteínas con motivos estructurales similares, ThiS y MoaD que son sus posibles antecesores. Una evidencia de ello son las dos glicinas del extremo C-terminal, que están presentes en estas proteínas y en las ubiquitinas.
Lo que más llama la atención es su alto grado de conservación, lo que pone en evidencia su importante papel dentro de la célula. Presentan 76 residuos de aminoácidos, y el ser humano solo se diferencia en 3 de estos con las levaduras.
Estas pequeñas proteínas pueden ser vistas como “chivatas” ya que marcan por unión covalente las proteínas que deben ser degradas en proteosomas.
El residuo que produce la unión es una Gly situada en el extremo C-terminal y que es común en todas las ubiquitinas. La glicina se une a la proteína diana por un enlace isopeptídico, que se diferencia del enlace peptídico en que uno de los grupos (sea NH2 o COOH) forma parte de un grupo R. En este caso ocurre entre en el grupo COOH de la Gly terminal con el grupo NH2 de la cadena lateral de una Lys de la proteína diana en gran número de ocasiones, pero también se ha visto que pueden unirse con grupos tiol de la Cys y grupos hidroxilo de Ser y Thr.
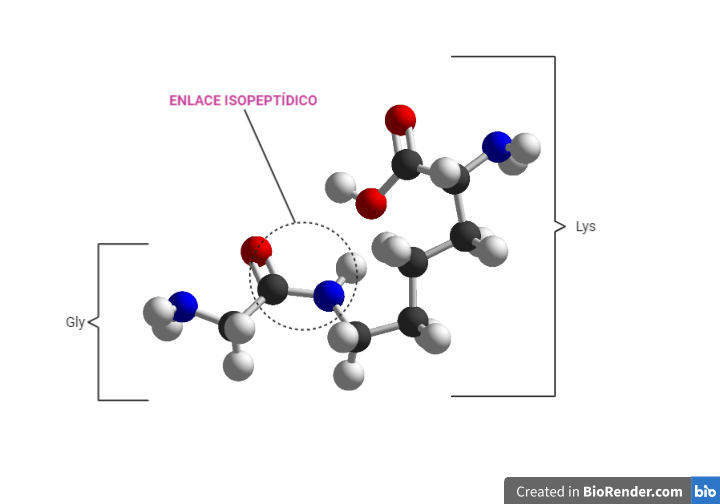
Las ubiquitinas no actúan solas en los procesos de marcaje de las proteínas a degradar, sino que estás se unen unas con otras formando cadenas. A este proceso se le conoce como poliubiquitinación y es clave para el reconocimiento de la proteína en el proteosoma. En la formación de la cadena cada eslabón de unión está formado por una Lys de la ubiquitina proximal y la Gly C-terminal de la distal.
A parte de las ubiquitinas, dentro de la célula podemos encontrar unas proteínas muy similares denominadas modificadores similares a ubiquitinas o Ubl (ubiquitin-like modifer proteins). Estas a parte de tener mecanismos comunes de unión con Lys, las Gly C-terminal y producir modificaciones en las proteínas, tienen un plegamiento casi idéntico al de la ubiquitina.
Proceso en cadena: E1, E2 y E3.
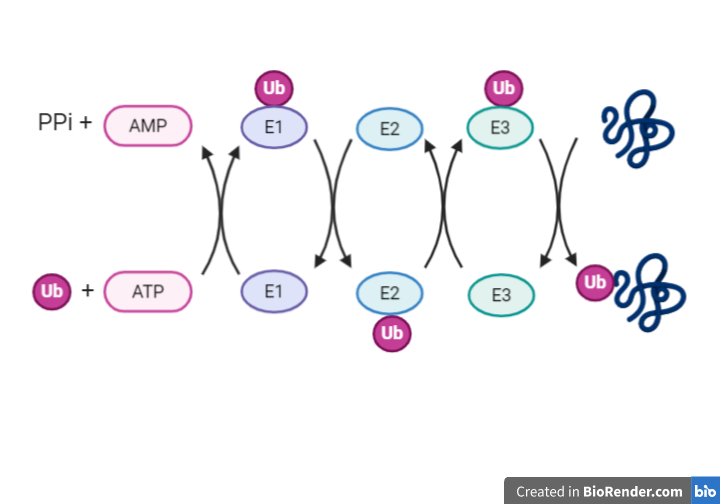
Hasta que la ubiquitina llega a marcar la proteína a degradar, tienen que actuar una serie de enzimas que la activen y unan.
E1: ubiquitina activasa
Esta proteína es la responsable de activar la ubiquitina mediante la unión a su extremo C-terminal de un residuo de cisteína (enlace tioéster). Para la formación de este enlace se requiere de ATP, que forma un intermediario con la ubiquitina antes de unirse a la enzima. La reacción de adenilación está muy conservada ya que se ha visto también en la proteína MoaD de bacterias.
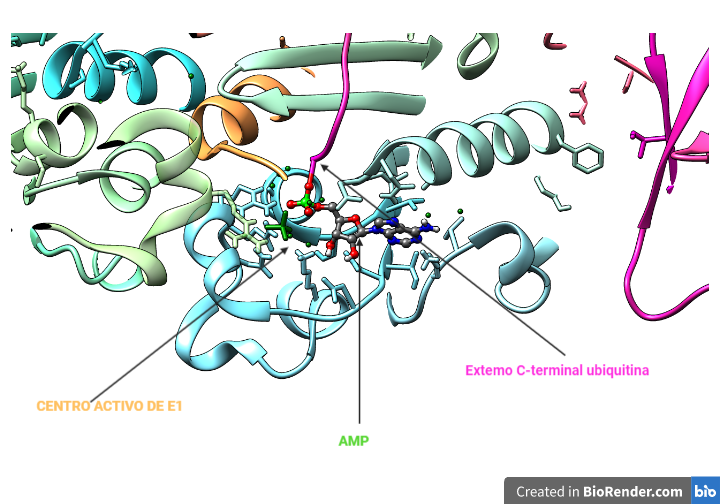
El intermediario Ub-AMP se sitúa en el centro activo de la E1, cerca de donde se encuentra la cisteína de unión. La estructura del nucleótido se estabiliza por puentes de hidrógeno con la enzima E1, reduciendo la entropía y favorecer el proceso de formación del enlace tioéster.
E2: enzima conjugadora
Las E2 unen las ubiquitinas que se habían unida a E1 también con un enlace en un residuo de cisteína.
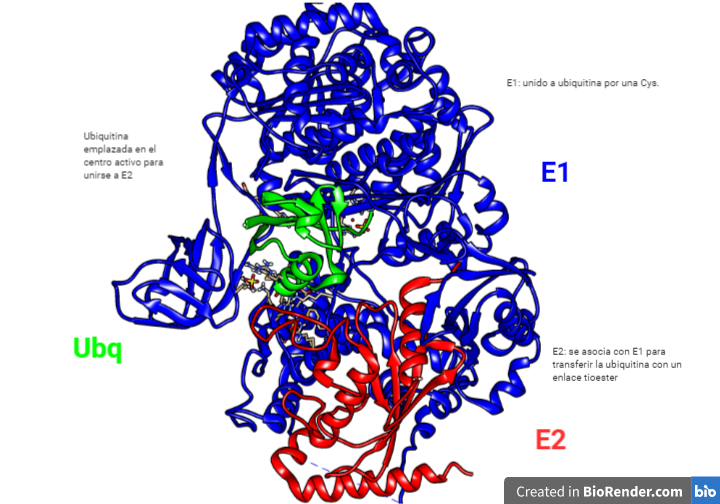
No es una única enzima E2, sino que son varias cada una con diferentes funciones. Estas después serán reconocidas por diferentes. El papel que puede tener E2 se discute, ya que podría ser más sencillo que E3 ligue directamente la ubiquitina desde E1 hasta la proteína diana.
Como es habitual, los eucariotas preferimos darle un vuelta de tuerca a todos los procesos moleculares. La presencia de E2 constituye un punto de regulación importante en la ubiquitinación y en la especificidad de los sustratos que se etiquetan.
La cisteína de E2 se encuentra en una hendidura de la estructura en la que debe entrar el complejo E1-Ubq para que se produzca la transferencia de una a otra.
E3: ubiquitina ligasa
La E3 tiene la habilidad de tomar la ubiquitina de la E2 y transferirla a la proteína diana. ¿Quiénes pueden ser estas proteínas dianas? Pues una gran diversidad de ellas. Es posible gracias a la existencia de varias enzimas E3 capaces de unir la ubiqutina a diferentes sustratos.
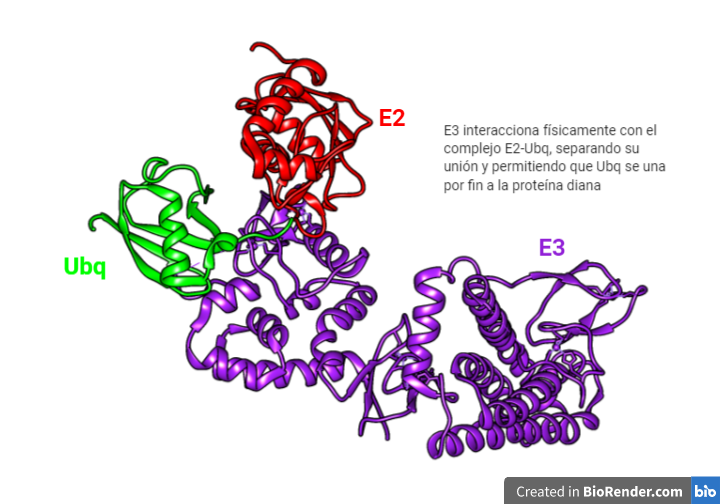
Dicha unión ocurre entre el C-terminal de la ubiquitina y una lisina de la proteína a degradar. Pero tienen la función también de enlazar unas ubiquitinas con otras para formar cadenas de poliubiquitinas.
Las tres clases de ubiquitina ligasa existentes se diferencian en función de su dominio activo: RING finger, HECT y U-box. Dentro de cada clase a su vez existen subclases, esto permite incrementar la especificidad del reconocimiento de las proteínas que queremos ubiquitinar. Para ello cada tipo de E3 cuanta con dominios de reconocimiento hacia determinadas proteínas y hacia diferentes tipos de E2.
RESUMEN DEL PROCESO ENCADENADO
Proteosomas: toneles de degradación
El sistema del proteosoma de ubiquitina (UPS) es un complejo multiproteico muy conservado que se encarga de la degradación enzimática de las proteínas. Está formado por una partícula o core central 20S. Este núcleo presenta dos anillos exteriores, con siete subunidades alfa, y dos anillos interiores con siete subunidades beta. Tiene forma de barril, en cuyo interior hay seis centros activos con actividad proteasa. El extremo amino-terminal de las subunidades alfa pueden abrirse para favorecer el paso de la proteína al interior del proteosoma.
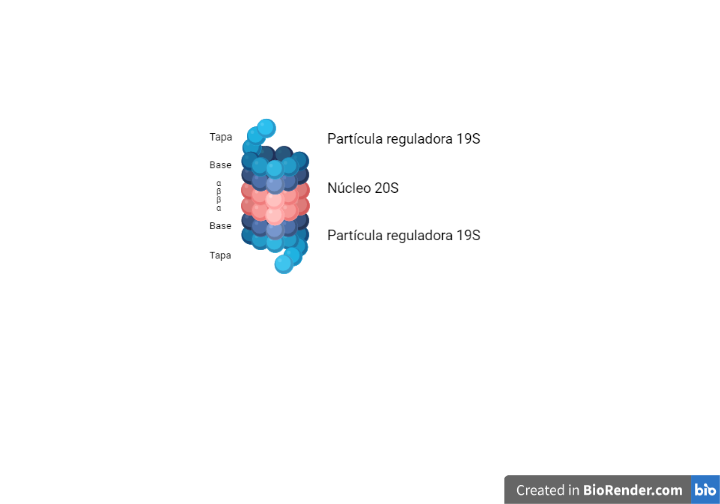
Además de la partícula central 20S, presenta partículas reguladoras 19S, compuestas por una tapa y una base. Esta última presenta seis moléculas de ATPasa, cuyos extremos carboxi-terminales encajan entre las subunidades alfa, provocando en ellas el cambio conformacional que abre la compuerta.
La ubiquitinación de las diferentes lisinas de las proteínas desencadena diferentes funciones, desde degradación de proteínas, hasta señales para reparar el DNA, activación de factores de transcripción, endocitosis…
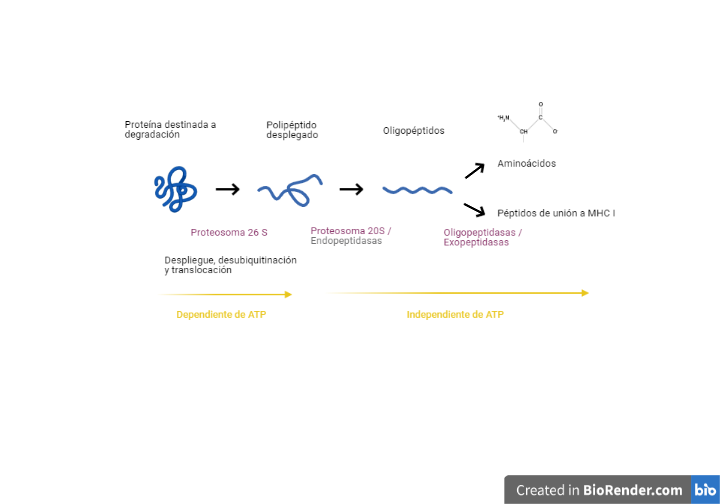
En el proceso de liberación de aminoácidos de la proteína se distinguen dos partes, según la utilización de energía metabólica: hay una parte del proceso que es dependiente de ATP, mientras que otra parte es independiente de ATP.
Las proteínas son desplegadas por reguladores mediante cambios conformacionales provocados gracias a la hidrólisis de ATP. Después, las proteínas desplegadas son traslocadas a los centros activos con actividad proteasa del núcleo central, dando lugar a pequeños péptidos de tres a quince aminoácidos. Por último, estos péptidos son degradados de forma independiente de ATP por endopeptidasas, carboxipeptidasas y aminopeptidasas.
Comprender la estructura y el funcionamiento del proteosoma ha permitido conocer su asociación con enfermedades cardiovasculares, diabetes, enfermedades neurológicas y cáncer. El gen que codifica el proteosoma se localiza en el cromosoma 6, cerca de la región donde se codifican las moléculas del MHC-II.
En la actualidad, se han descrito cuatro tipos de proteosomas:
- Proteosoma constitutivo, clásico o proteosoma 26S.
Es una estructura grande, macromolecular y multiproteica que tiene carácter enzimático. Se localiza principalmente en el citosol, aunque también en el núcleo de todos los eucariotas, por lo que es el principal complejo proteolítico celular, además, es la misma vía que la célula usa para presentar los péptidos en la superficie en el contexto de MHC-I.
Reconoce los sustratos de modo dependiente de ubiquitina, de modo mediado por un adaptador y de manera independiente de ubiquitina, en las tres formas requiere de ATP, siendo la vía dependiente de ubiquitina la más usada por microorganismos eucariotas.
- Inmunoproteosoma
Fue descubierto en células WEHI-3 al ser tratadas con IFN-gamma. Se localiza en las células dendríticas de la médula del timo y en las células epiteliales tímicas medulares.
Se observó que presentan baja actividad tipo caspasas y una actividad alta de tipo tripsina en comparación con el proteosoma 26S, por lo que podría producir diferentes tipos de péptidos, debido a su tendencia a producir escisión detrás de aminoácidos hidrofóbicos o básicos. Además, optimiza la respuesta efectora frente a determinados antígenos mediante la activación selectiva de clones de linfocitos CD8+.
Su estudio nos permite comprender mejor los mecanismos de aparición de determinados tipos de cánceres y enfermedades autoinmunes.
- Proteosomas intermedios
Es un proteosoma intermedio entre el proteosoma clásico y el inmunoproteosoma. Se ha observado en tumores del hígado, colon, intestino delgado y células dendríticas. El hallazgo de estos proteosomas intermedios en células dendríticas podría servir como herramienta para la inmunoterapia.
- Timoproteosoma
Son los proteosomas que se encuentran en el timo. Son necesarios para que pueda llevarse a cabo de manera correcta el mecanismo de selección positiva de los linfocitos CD8+, aunque no se sabe muy bien cómo lo hace. Genera de forma constitutiva ligandos de péptidos alterados durante el mecanismo de selección positiva, lo cual jugará un rol importante en el proceso de la tolerancia inmune.
Importancia del sistema: ciclo celular, enfermedades neurodegenerativas y cáncer.
Esta función que presentan los proteosomas los hace especialmente importantes a la hora de controlar el ciclo celular. Las kinasas dependientes de ciclinas (CDK) controlan la progresión del ciclo celular tras ser activadas por ciclinas. Cuando ya han llevado a cabo su función es necesario que sean eliminadas. Un mal funcionamiento de estas CDK puede producir un proceso tumoral.
La ciclina será poliubiquitinizada y, posteriormente, degradada por el proteosoma, para que pueda continuar el ciclo celular. En concreto, para que la célula finalice la mitosis, requiere que el componente regulador ciclina B se disocie. En las células de vertebrados esta disociación será dependiente de los proteosomas.
En el cáncer, la mutación de ciertos genes provoca que las células sean inmortales. En estas células (sea por mutación o activación) habrá una sobreexpresión del proteosoma, el cual estará degradando la ciclina en exceso. Esto lo que provoca es que la célula nunca pare de dividirse.
Teniendo esto en cuenta, muchos fármacos y quimioterapias tienen como diana la alteración o inhibición de los proteosomas, haciendo que las células entren en apoptosis y el cáncer se frenaría.
Hay muchos tipos de ciclinas que se unen a CDK específicos, por ello, los CDK-inhibidores (p21 y p27) efectúan su acción cuando hay algún daño celular o cromosómico. Estos CDK-inhibidores inhiben el ciclo celular en estos casos siempre y cuando el proteosoma esté inhibido o regulado, ya que en células tumorales se encontraría sobreexpresado y degradando CDK-inhibidores, impidiendo la regulación de este punto de control.
Otro punto de control del ciclo celular que da lugar a procesos cancerígenos es llevado a cabo por factores de supervivencia celular (ikB y NFKB). A este nivel, la inhibición del proteosoma también provocaría la apoptosis celular al no haber señales de supervivencia.
El inmunoproteosoma participa en la regulación del desarrollo de tumores. Se ha observado en pacientes humanos con leimoisarcoma uterino, que carecen de una de las subunidades del inmunoproteosoma. También se ha demostrado su sobreexpresión en leucemias agudas. Por lo que se sugiere que, dependiendo del tipo de cáncer, el inmunoproteosoma puede ser una consecuencia de la enfermedad o puede actuar como un factor de desarrollo. Por ello, se ha convertido en un objetivo importante para luchar contra el cáncer, ya que las células cancerígenas son más subceptibles a los inhibidores de proteosomas. Por ejemplo, en el Mieloma Múltiple, que es un trastorno del plasma sanguíneo debido a la proliferación de células tumorales en la médula ósea, se utilizan inhibidores del proteosoma, estimulando así señales apoptóticas.
Las enfermedades neurodegenativas se caracterizan por la acumulación de proteínas aberrantes que tienden a unirse entre sí. El origen de muchas enfermedades neurodegenerativas se debe a un mal funcionamiento del inmunoproteosoma.
En la enfermedad del Parkinson, las neuronas dopaminérgicas dañadas producen un exceso de proteínas anómalas que interaccionan entre sí formando “placas amiloideas”. La célula trata de eliminarlas mediante la vía ubiquitina-proteosoma. Llegada a una edad, estos mecanismos no funcionan bien, provocando la apoptosis neuronal generando la enfermedad.
La Enfermedad de Alzheimer es un proceso neurodegenerativo de la neocorteza y el hipocampo. En ella se definen dos tipos principales de lesiones: las placas neuríticas y las marañas neurofibrilares. Ambos representan los productos de un trastorno del plegamiento de proteínas que se caracterizan por la proteolisis. Esta acumulación está relacionada con disfunciones de la actividad proteolítica del proteosoma.
La acumulación de presenilina-2 debido al mal funcionamiento del proteosoma en enfermedad de Alzheimer puede ser asociado con la acumulación de β-amioloide, lo que dificultaría la función del proteosoma con el consecuente daño celular.
REFERENCIAS
Pickart, C. M. and Eddins, M. J. (2004) ‘Ubiquitin: Structures, functions, mechanisms’, Biochimica et Biophysica Acta – Molecular Cell Research, 1695(1–3), pp. 55–72. doi: 10.1016/j.bbamcr.2004.09.019.
Strous, G. J. and Govers, R. (1999) ‘The ubiquitin-proteosome system and endocytosis’, Journal of Cell Science, 112(10), pp. 1417–1423.
Tanaka, K. (2009) ‘The proteasome: Overview of structure and functions’, Proceedings of the Japan Academy Series B: Physical and Biological Sciences, 85(1), pp. 12–36. doi: 10.2183/pjab.85.12.
Amm, I., Sommer, T. and Wolf, D. H. (2014) ‘Protein quality control and elimination of protein waste: The role of the ubiquitin-proteasome system’, Biochimica et Biophysica Acta – Molecular Cell Research. Elsevier B.V., 1843(1), pp. 182–196. doi: 10.1016/j.bbamcr.2013.06.031.
Pick, E., & Berman, T. S. (2013). Formation of alternative proteasomes: Same lady, different cap? FEBS Letters, 587(5), 389–393. https://doi.org/10.1016/j.febslet.2013.01.014
Esser, C., Scheffner, M., & Höhfeld, J. (2005). The chaperone-associated ubiquitin ligase CHIP is able to target p53 for proteasomal degradation. Journal of Biological Chemistry, 280(29), 27443–27448. https://doi.org/10.1074/jbc.M501574200
Cellular protein degradation occurs in different cellular. (n.d.).
Redondo, C. L. (2015). Papel del inmunoproteosoma en enfermedades neurodegenerativas y cáncer Revisión bibliográfica.
Barrio, R., & Izquierdo, M. (n.d.). Aplicaciones terapéuticas de la ubiquitina.
Ubiquitinación, U., Un Un Sistema Sistema Sistema Sistema Sistema De De De De De Regulación Regulación Regulación, U. U., Manuel Zamudio-Arroyo, J., Teresa Peña-Rangel, M., Rafael Riesgo-Escovar María Teresa Peña-Rangel, J., & Rafael Riesgo-Escovar, J. (n.d.).
Erales, J., & Coffino, P. (2014, January). Ubiquitin-independent proteasomal degradation. Biochimica et Biophysica Acta – Molecular Cell Research. https://doi.org/10.1016/j.bbamcr.2013.05.008
Zelada Valdés, A. (2019). The proteasome: subtypes and involvement in central tolerance. Revista Cubana de Investigaciones Biomédicas (Vol. 38).
Hershko, A., & Ciechanover, A. (1998). THE UBIQUITIN SYSTEM. Annu. Rev. Biochem (Vol. 67).