Un RNA funcional: XIST, lionización del cromosoma X, corpúsculo de Barr
Ana Belén Alonso Aguado, Irene Chavarría Cubel y Rodrigo Díaz Muñoz. 3º Biología Sanitaria, UAH.
La lionización debe su nombre a la genetista Mary Lyon y consiste en el proceso de inactivación de uno de los cromosomas X en las células de las hembras de mamíferos. Para ello, se producen una serie de modificaciones epigenéticas que conducen a la aparición de la cromatina sexual o corpúsculo de Barr, un cuerpo de heterocromatina adherido a la envoltura nuclear.
Estas modificaciones se desencadenan gracias a un RNA no codificante funcional, producido por el gen Xist situado en la región XIC del cromosoma X, el cual permite el reclutamiento de los factores implicados en la reorganización de la cromatina.
El objetivo de este fenómeno es equiparar los niveles de expresión de los genes ligados al sexo, ya que las hembras presentan dos cromosomas X, mientras que los machos poseen sólo uno (XY).
Antecedentes históricos: corpúsculo de Barr e hipótesis de Lyon
En 1949, Bertran y Barr introdujeron el término cromatina sexual o corpúsculo de Barr tras observar en neuronas del asta anterior de la médula espinal de gatos, junto al nucléolo y bien distinguido de éste, un corpúsculo de cromatina densa (heterocromatina), que estaba adherido a la envoltura nuclear. Esta estructura solo aparecía en hembras, por lo que pensaron que estaría relacionado con el sexo.
En 1959, Susumu Ohno demostró que el corpúsculo de Barr se corresponde con un cromosoma X heterocromatizado y propuso que uno de los dos cromosomas X está inactivo en cada célula somática (células epiteliales, fibroblastos, leucocitos, etc.).
En 1966, Mary Lyon propuso la hipótesis de Lyon para la cromatina sexual o corpúsculo de Barr:
- La cromatina sexual es genéticamente inactiva.
- La heterocromatización ocurre en la embriogénesis temprana (hacia el día 16 del desarrollo embrionario en la mujer).
- El proceso de inactivación del cromosoma X en cada célula de una hembra se produce al azar, es decir, puede inactivarse el cromosoma X materno o paterno.
- Se trata de un proceso irreversible y heredable, por lo que en un linaje celular se mantendrá el mismo patrón de inactivación.
Como consecuencia, las hembras poseen mezclas de líneas celulares en las que se inactiva el cromosoma X paterno y líneas celulares en las que se inactiva el cromosoma X materno, manifestándose unos u otros factores genéticos ligados al sexo. De esta forma, una hembra heterocigótica para una característica o enfermedad ligada al cromosoma X es un mosaico (ver figura 2).
Además, el corpúsculo de Barr sigue la llamada “regla n-1” (ver figura 1): el número de corpúsculos de Barr de una célula es igual al número de cromosomas X que posee la célula (n) menos 1. Por lo tanto, en todas las células femeninas hay un único cromosoma X activo, mientras que el otro (cariotipo XX) o los otros cromosomas X (cariotipos anómalos) se encuentran heterocromatizados (inactivos). Solo en la meiosis de los ovocitos el cromosoma X heterocromatizado se reactiva, de manera que el patrón de inactivación no se mantiene en la descendencia.
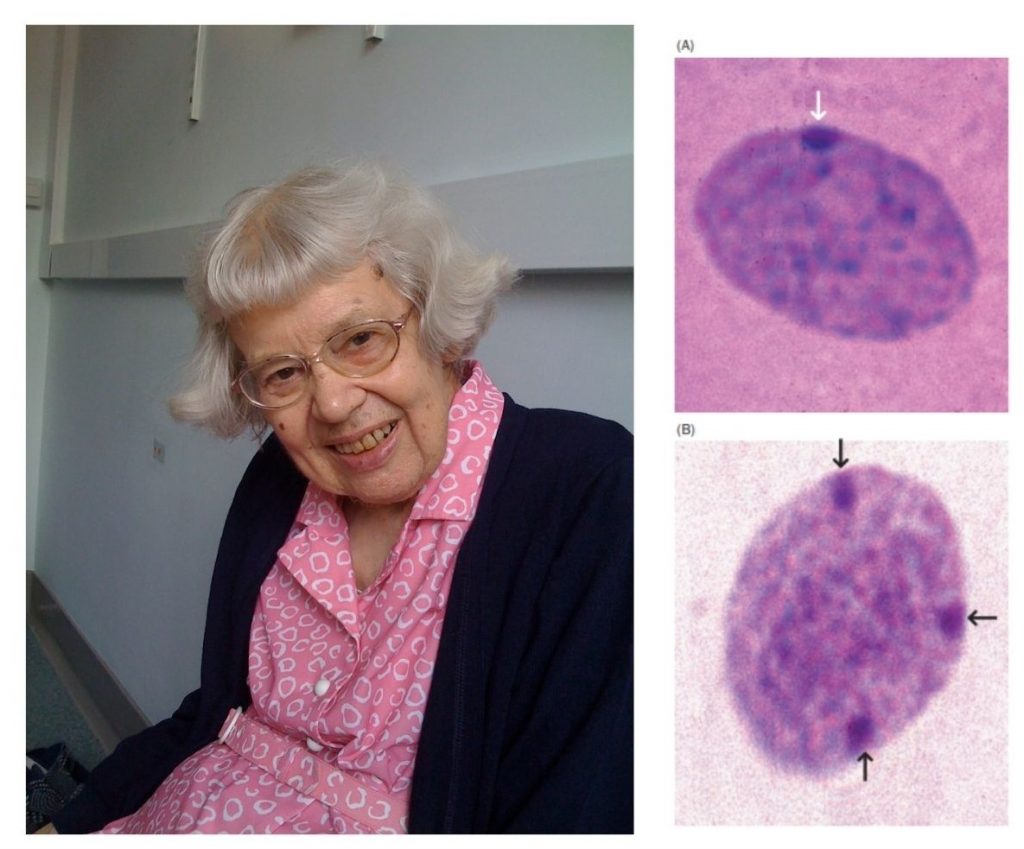
Figura 1. A la izquierda, imagen de Mary Lyon. A la derecha, corpúsculos de Barr al microscopio óptico. (A) Célula femenina con cariotipo 46 XX; presenta un cromosoma X inactivo y, por ende, un único corpúsculo de Barr (señalado con flecha blanca). (B) Célula masculina con cariotipo anómalo 49 XXXXY; presenta tres cromosomas X inactivos y, por tanto, muestra tres corpúsculos de Barr (flechas negras). [13]
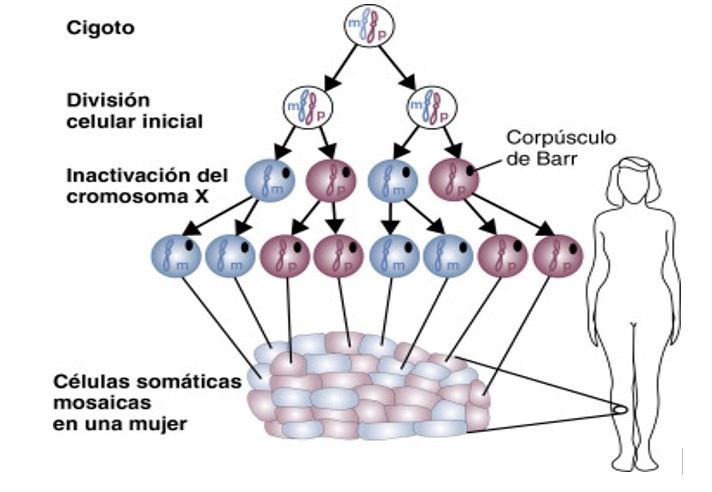
Figura 2. Si se ha inactivado el cromosoma X materno (m) o paterno (p) en una célula del embrión en las etapas tempranas del desarrollo embrionario, en todas las células que se originen a partir de ésta se inactivará siempre el cromosoma X materno o paterno, respectivamente. Como resultado, observamos células somáticas formando tejidos mosaicos. [12]
Además, el cromosoma X heterocromatizado no se inactiva por completo (ver figura 3). Se estima que la inactivación afecta al 65% de los genes del cromosoma en todas las células, mientras que un 20% se inactiva solo en algunas células y el 15% consigue escapar de este proceso. Por ello, las mujeres con síndrome de Turner (X0) presentan un fenotipo particular. Si el cromosoma X se inactivara completamente, el fenotipo de estas mujeres sería idéntico al de las mujeres XX.
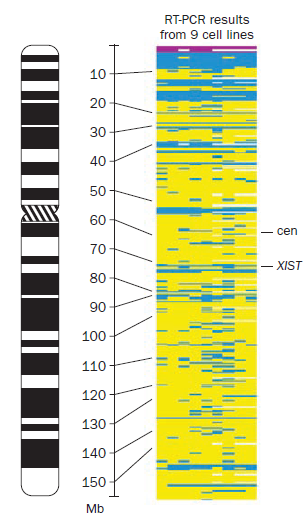
Figura 3. El esquema muestra los genes que se expresan (azul) y no se expresan (amarillo) en el cromosoma X inactivo (Xi). Estos resultados se obtuvieron mediante RT-PCR. [13]
Ejemplos de fenotipos mosaico ligados al cromosoma X
- Color del pelaje en los gatos calicó: el pelaje de las gatas puede ser naranja, negro o parcheado, mientras que el de los gatos es totalmente negro o naranja (ver figura 4).
- Enfermedades dermatológicas: algunas de estas enfermedades, como la displasia ectodérmica anhidrótica, presentan un patrón en mosaico.
- Isoformas de glucosa-6-fosfato deshidrogenasa: se observó la presencia de una única isoforma en fibroblastos aislados de mujeres heterocigotas para los genes de las isoenzimas A y B. Esto confirmó la hipótesis de Lyon, ya que, si no hubiera inactivación del cromosoma X y ésta no fuese al azar, se deberían observar dos isoformas distintas o la misma en todas las células (suponiendo uno de los alelos dominante).
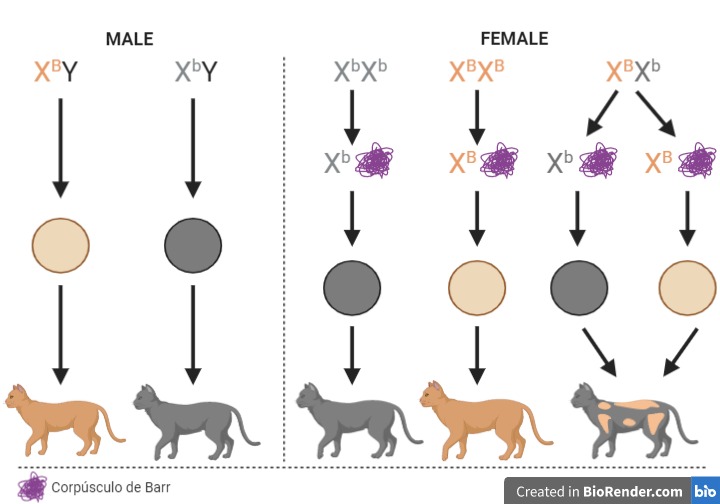
Figura 4. El color de las gatos calicó depende del gen B que se encuentra localizado en el cromosoma X; el alelo B da lugar a una coloración naranja y el b a una coloración negra. Como los machos solamente tienen un cromosoma X, serán naranjas o negros (B o b). Sin embargo, podremos encontrar hembras homocigotas para el alelo b (todas sus células independientemente del cromosoma que se inactive presentarán el alelo b y serán negras), homocigotas para B (naranjas) o heterocigotas (mosaicos). En este último caso, las zonas naranjas proceden de las células en las que se inactivó el cromosoma X portador del alelo b, mientras que las zonas negras estarán formadas por las células en las que se inactivó el cromosoma X portador del alelo B. Creada con BioRender.
Mecanismo de inactivación del cromosoma X y modificaciones en la cromatina
El proceso de inactivación del cromosoma X consta de cuatro pasos: contaje (cociente entre el número de cromosomas X y autosomas), selección (inactivación del cromosoma X materno o paterno), iniciación de la inactivación y mantenimiento en las siguientes generaciones de un mismo linaje celular.
La heterocromatización se inicia en un punto del cromosoma X llamado XIC (centro de inactivación de X), el cual está situado en el locus multifuncional Xq13 y se extiende hacia ambos extremos del cromosoma (ver figura 5).
Principales elementos de XIC implicados:
- Gen Xist (transcrito específico para la inactivación de X): produce un transcrito primario que, tras ser procesado por splicing y poliadenilación, da lugar a un RNA no codificante y funcional de 15-17 kb necesario para iniciar el silenciamiento del cromosoma X. Este RNA se dispone a lo largo del cromosoma X que se debe inactivar y recluta una serie de factores, los cuales producen modificaciones epigenéticas que conducen a la condensación de la cromatina.
- Locus Xce (elemento regulador del cromosoma X): los genes incluidos en este locus están implicados en los mecanismos de contaje y selección del cromosoma X que se va a inactivar.
- Genes reguladores de la expresión del gen Xist: activadores (Jpx, Ftx, Rnf12) y represores (Tsix en ratones). El gen Jpx y Ftx producen RNAs no codificantes que activan la expresión del gen Xist, mientras que el gen Rnf12 produce una proteína con actividad ubiquitina-ligasa que parece degradar un inhibidor del gen Xist. Por el contrario, el gen Tsix en ratones produce un RNA antisentido no codificante (complementario al Xist RNA) que inhibe la expresión del gen Xist y es fundamental en la selección del cromosoma X que se va a inactivar.
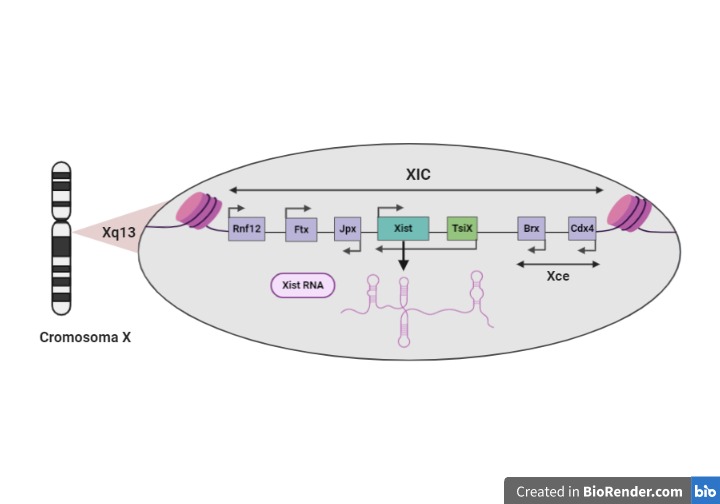
Figura 5. La figura muestra un esquema de la región XIC en ratones. Creada con BioRender.
En el comienzo del desarrollo embrionario, los factores de pluripotencialidad (NanoG, Sox2 y Oct-4) interaccionan con regiones promotoras del gen Xist, manteniendo bajos los niveles de expresión de este gen, tanto en el cromosoma X de origen materno como en el paterno. Además, estos factores regulan positivamente la expresión del gen Tsix (represor).
Llega un momento, aun en la embriogénesis temprana, en el que se produce un apareamiento transitorio entre algunas regiones de las secuencias XIC de dos cromosomas X. Este proceso parece constituir un mecanismo de recuento de cromosomas X y se repetirá hasta que solo quede uno activo (en caso de cariotipos anómalos con más de dos cromosomas X) (ver figura 6).
Como resultado de la interacción, se produce la activación del gen Tsix (represor) al azar en uno de los cromosomas (“competencia”). Esto conducirá al cese de la expresión del gen Xist en uno de los cromosomas X, mientras que aumentará significativamente en el otro (futuro cromosoma X inactivo o Xi).
En humanos también se da este apareamiento, pero Tsix produce un RNA antisentido no codificante truncado en el extremo 5’, incapaz de actuar como represor de Xist (en este caso, Tsix parece ser un vestigio evolutivo).
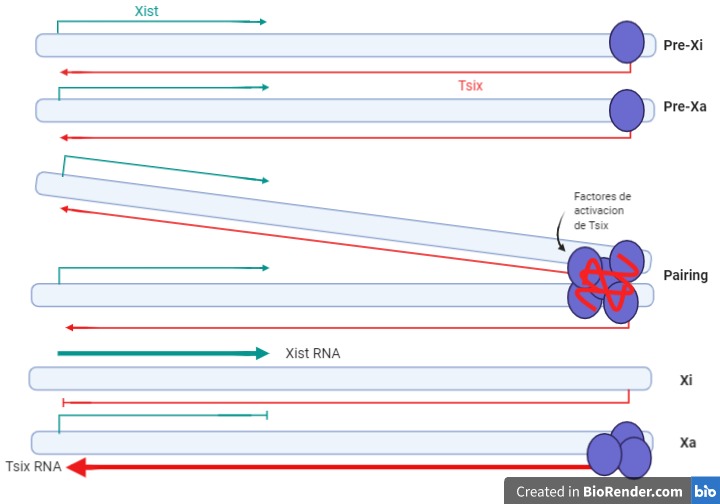
Figura 6. Apareamiento y papel del gen Tsix en la inactivación del cromosoma X en ratones. La expresión de Xist determinará el futuro cromosoma inactivo (Xi), mientras que la expresión de Tsix determinará el futuro cromosoma activo (Xa). Creado con BioRender.
A continuación, comienza el silenciamiento del cromosoma X seleccionado. Para ello, en el futuro cromosoma Xi, el gen Xist produce un transcrito primario que será procesado mediante splicing y poliadenilación para dar lugar a un RNA no codificante funcional que recubre el cromosoma Xi. Este es el Xist RNA, requerido para iniciar y estabilizar la inactivación del cromosoma X, pero no para mantenerla.
Se ha observado que, al eliminar el gen Xist del cromosoma X, éste no se inactiva. Sin embargo, si se coloca en cualquier cromosoma autosómico, automáticamente se produce la inactivación de dicho cromosoma.
Los elementos LINE (elementos nucleares dispersos largos) son secuencias repetidas a lo largo del cromosoma X, principalmente en regiones cercanas a XIC (forman un 30% del cromosoma). Al parecer, estas secuencias favorecen una rápida expansión del Xist RNA, facilitando el proceso de inactivación.
Una vez situado sobre el futuro cromosoma Xi, el Xist RNA recluta directa o indirectamente una serie de factores, como los complejos proteicos Polycomb PRC1 y PRC2, que reorganizan la cromatina para que ésta adquiera una conformación de heterocromatina facultativa (cromatina condensada transcripcionalmente inactiva).
El mantenimiento de la inactivación del cromosoma X se debe a las siguientes modificaciones epigenéticas que producen la condensación de la cromatina: trimetilación en las lisinas 9 y 27 de la histona H3 (H3K9me3 y H3K27me3, respectivamente), desmetilación de la lisina 4 de la H3 (no H3K4me3), aumento de nucleosomas con la variante macroH2A de la histona H2A, desacetilación de histonas y metilación de las islas CpG (residuos de citosina unidos a guanina) en los promotores de los genes que se van a inactivar (en los genes activos estas islas no están metiladas). Como consecuencia, se producirá una exclusión de la RNA polimerasa II y otros factores, así como una replicación tardía en la fase S (ver figura 7).
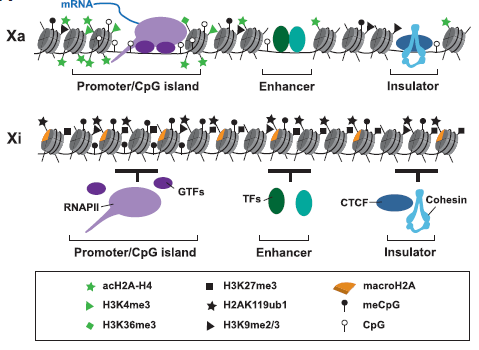
Figura 7. Diferencias a escala nucleosomal en las modificaciones epigenéticas del cromosoma X activo (Xa) e inactivo (Xi). RNAPII: RNA Polimerasa II; GTFs: Factores transcripcionales generales; TFs: Factores transcripcionales de unión a intensificadores; CTCF: Proteína aisladora. Enhancer: secuencia intensificadora; Insulator: secuencia aisladora. Nomenclatura modificaciones en el recuadro inferior: ac (acetilación), H (histona), K (lisina), me (metilación), me2 (dimetilación), me3 (trimetilación), ub (ubiquitinación), CpG (islas Cpg), meCpG (metilación islas CpG). [5]
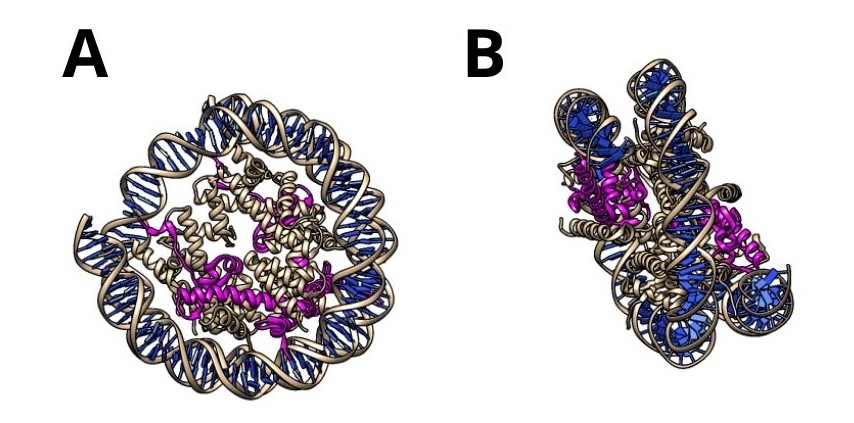
Figura 8. En morado se observan las histonas macroH2A y en azul el DNA, formando un nucleosoma. Las figuras A y B muestran distintas perspectivas. PDB: 2F8N. Creado con UCSF Chimera.
Xist RNA y factores reclutados implicados en la remodelación de la cromatina
El Xist RNA presenta unas regiones formadas por repeticiones en tándem de secuencias consenso, las cuales se clasifican de la A a la F. Las repeticiones A-D y F se encuentran en el exón 1, mientras que las repeticiones E se encuentran en el exón 7 (ver figuras 9, 10 y 13).
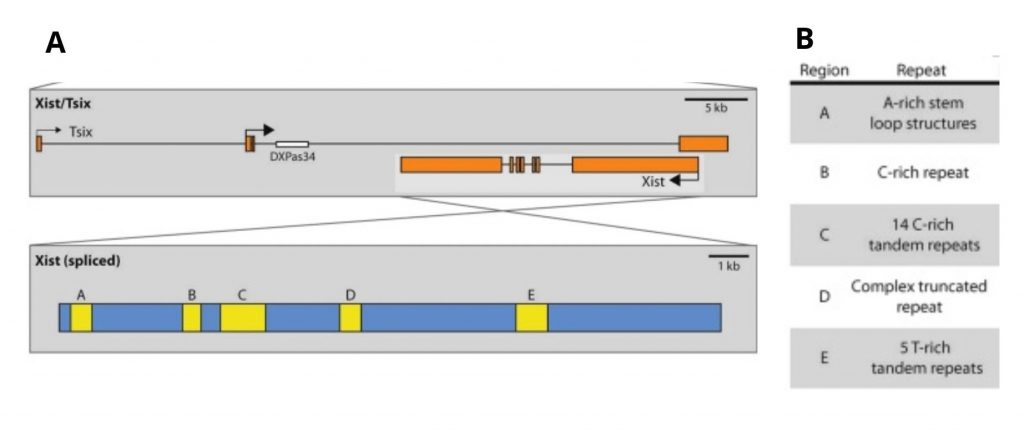
Figura 9. (A) El esquema superior muestra los genes Xist y Tsix localizados en la región XIC del cromosoma X. El esquema inferior muestra el Xist RNA tras el splicing, donde se ilustran en amarillo las repeticiones en tándem de secuencias consenso (A-E). (B) Tabla que describe las secuencias repetidas. [8]
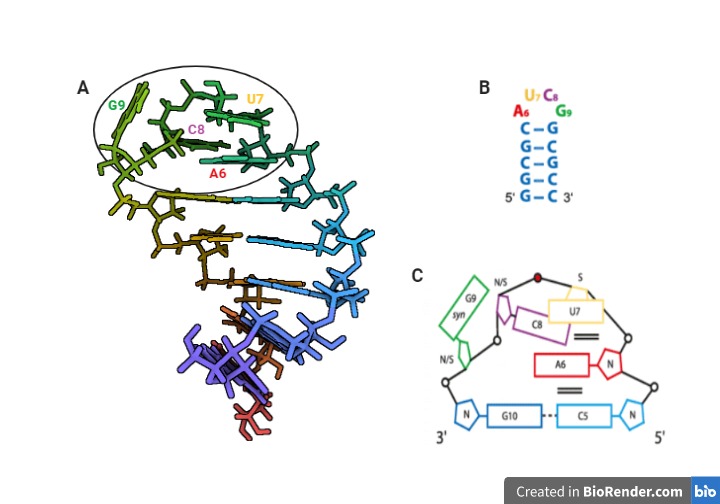
Figura 10. Tetraloops AUCG en las repeticiones A de Xist RNA. (A) Estructura secundaria en solución de las repeticiones A del Xist RNA en humanos. PDB: 2Y95. El círculo negro marca el tetraloop AUCG. Las letras de colores indican los nucleótidos del tetraloop, que se corresponden con los indicados en los esquemas B y C. (B) Esquema de la estructura ilustrada en A. (C) Esquema del tetraloop AUCG, rodeado en la ilustración A con un círculo negro. Creada con BioRender y [15].
Como ya sabemos, Xist RNA inicia el silenciamiento del cromosoma X mediante el reclutamiento directo e indirecto de distintos factores implicados en la remodelación de la cromatina. Estos factores interaccionan con las repeticiones mencionadas anteriormente y se conocen como proteínas de unión al RNA (RBPs). Actualmente, se sabe que las principales RBPs que participan en el silenciamiento son: SPEN (SHARP en humanos), RBM15, hnRNPK y LBR.
SPEN (SHARP en humanos)
SPEN es una proteína con capacidad de interacción con el RNA y otras proteínas gracias a cuatro dominios RRM presentes en el extremo N-terminal y un dominio SPOC en el extremo C-terminal, respectivamente (ver figura 11).
Por un lado, los RRM 2-4 (sobre todo RRM 3) interactúan directamente con las repeticiones A del Xist RNA, mientras que el dominio SPOC recluta el complejo de desacetilación de histonas (NCoR-HDAC3).
De esta forma, se llevará a la desacetilación de histonas en el cromosoma X que se está inactivando. Como ya sabemos, esta desacetilación es una de las modificaciones que favorecen la condensación de la cromatina y, por tanto, la disminución de la accesibilidad de la RNA polimerasa II a los promotores de los genes.
Parece que otra función de SPEN es secuestrar un complejo coactivador de la metiltransferasa H3K4 (KMT2D) mediante el dominio SPOC, de manera que disminuirá la actividad de la metiltransferasa H3K4 (recordemos que la metilación de H3K4 es característica del cromosoma X activo) (ver figura 7).
Además, algunos estudios sugieren que SPEN podría ser responsable de los bajos niveles de RNA polimerasa II (RNAPII) sobre el cromosoma Xi, ya que parece interactuar con esta enzima y algunos cofactores asociados, secuestrándolos.
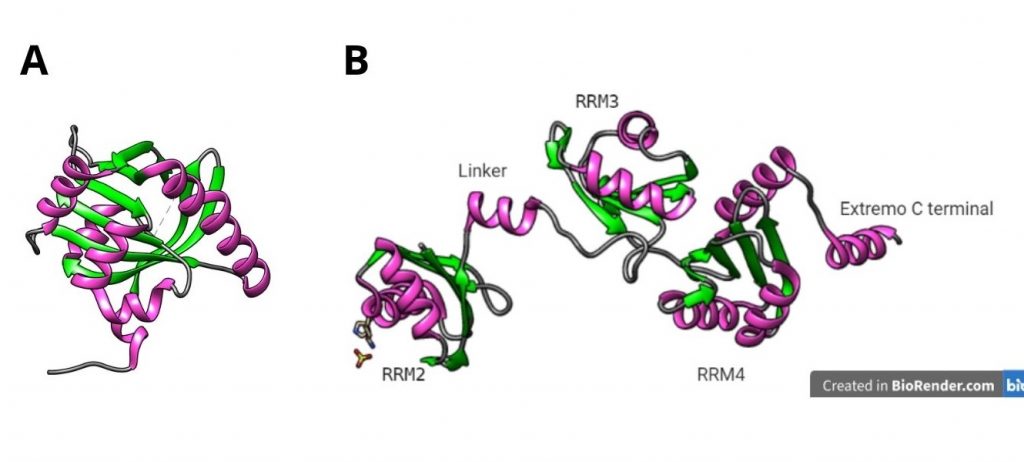
Figura 11. (A) Dominios SPOC (PDB: 1OW1) y (B) RRM 2-4 de la proteína SPEN (PDB: 4P6Q). Creado con UCSF Chimera y BioRender.
RBM15
RBM15 es una proteína perteneciente a la misma familia que SPEN, por lo que presenta tres dominios conservados RRM (unión a RNA) en el extremo N-terminal y un dominio SPOC (unión a otras proteínas) en el extremo C-terminal.
Los dominios RRM, al igual que en la proteína SPEN, interactúan con las repeticiones A del Xist RNA. Sin embargo, el dominio SPOC va a interactuar con el complejo proteico METTL3/14 que lleva a cabo la metilación del N6 de adenosinas (m6A). Esta modificación parece estabilizar el RNA, siendo muy abundante en RNAs no codificantes, en este caso Xist RNA, y también en mRNAs.
Asimismo, al igual que en SPEN, el dominio SPOC de RBM15 puede secuestrar otro complejo coactivador de la metiltransferasa H3K4 (SET1B o KMTD2G). De esta forma, disminuye la metilación de la lisina 4 en la histona 3 del cromosoma Xi.
hnRNPK
hnRNPK es otra de las RBPs que interacciona con Xist RNA, en este caso con las repeticiones B/C a través de tres dominios KH (KH1, KH2, KH3) (ver figura 12). Asimismo, a través de su dominio KI cercano al extremo C-terminal, recluta el complejo Polycomb PRC1.
Los complejos multienzimáticos Polycomb catalizan las modificaciones de histonas que conducen a la remodelación de la cromatina.
El complejo PRC1 cataliza la ubiquitinación de la lisina 119 de la histona H2A (H2AK119ub1). Esta modificación promueve la concentración del complejo PRC2 sobre el cromosoma Xi, así como de otros complejos Polycomb. En concreto, el complejo PRC2 produce la trimetilación de la lisina 27 de la histona 3 (H3K27me3).
El mecanismo de acción de los complejos Polycomb aún se desconoce, pero parece que el silenciamiento génico podría ser resultado de:
- La ubiquitinación y metilación de histonas podría reclutar otras proteínas implicadas en el silenciamiento de los genes.
- Como resultado de la modificación de las histonas, la cromatina se reorganiza de manera que limita la accesibilidad a los factores de transcripción y la RNA polimerasa II.
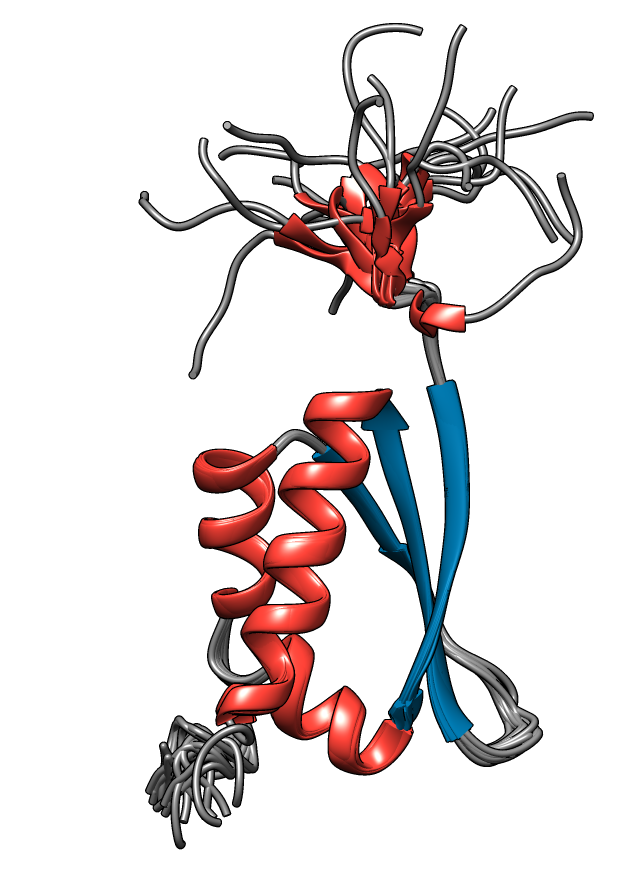
Figura 12. Dominio KH de hnRNPK. PDB: 1KHM. Creado con UCSF Chimera.
LBR
LBR es un receptor de la lámina nuclear que interactúa directamente con Xist RNA, por lo que se incluye dentro de las RBPs. Esta unión se produce principalmente entre el dominio SR (tramo de arginina-serina) de LBR y los sitios LBS en las repeticiones F de Xist RNA.
Por lo tanto, la interacción de LBR con Xist RNA permite la unión del cromosoma Xi a la lámina nuclear (por ello, el corpúsculo de Barr se observa en la periferia nuclear). En estudios recientes, se ha observado que esto podría mejorar la tasa de silenciamiento.
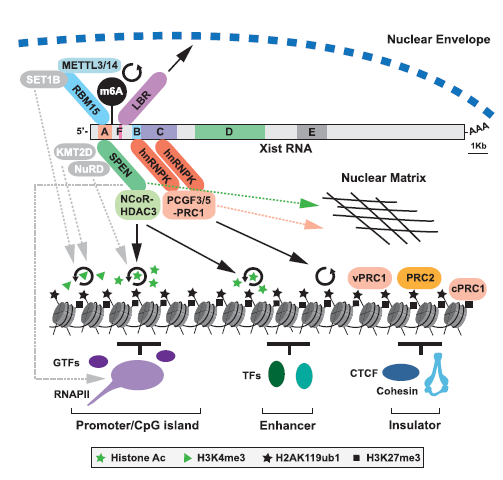
Figura 13. Esquema resumen de las interacciones de las RBPs con las repeticiones de Xist RNA (A, F, B, C, D, E), así como los factores reclutados por cada una de ellas que ejercen diversos efectos sobre la estructura de la cromatina y la localización del cromosoma Xi en el núcleo celular. [5]
Xist RNA antagonista del complejo SWI/SNF
El complejo SWI/SNF se encarga de la remodelación de la cromatina y, normalmente, actúa cuando se requiere una activación de la transcripción. En estudios recientes, se ha propuesto el posible papel de Xist RNA como antagonista de este complejo.
Curiosamente, la deleción del gen Xist produce un aumento de la accesibilidad de la cromatina regulado por BRG1, una subunidad ATPasa del complejo remodelador de la cromatina SWI/SNF.
Por tanto, parece que la la unión de Xist RNA al cromosoma Xi inhibe la actividad de remodelación del nucleosoma de BRG1 y da como resultado la expulsión del complejo SWI/SNF del Xi.
Bibliografía
1. Jégu T, Blum R, Cochrane JC, Yang L, Wang C-Y, Gilles M-E, et al. Xist RNA antagonizes the SWI/SNF chromatin remodeler BRG1 on the inactive X chromosome. Nat Struct Mol Biol. 2019 Feb 21;26(2):96–109.
2. Balderman S, Lichtman MA. A History of the Discovery of Random X Chromosome Inactivation in the Human Female and its Significance. Rambam Maimonides Med J. 2011 Jul 29;2(3).
3. Acosta Lobo ME, Vásquez Araque NA, Londoño Franco LF. Inactivación del cromosoma X en el desarrollo embrionario mamífero. CES Med Vet y Zootec. 2013;8(2):108–19.
4. Migeon BR. Is Tsix repression of Xist specific to mouse? Nat Genet. 2003 Mar 1;33(3):337–337.
5. Brockdorff N, Bowness JS, Wei G. Progress toward understanding chromosome silencing by Xist RNA. Genes Dev. 2020 Jun 1;34(11–12):733–44.
6. Maclary E, Hinten M, Harris C, Kalantry S. Long nonoding RNAs in the X-inactivation center. Chromosom Res. 2013 Dec 3;21(6–7):601–14.
7. Froberg JE, Yang L, Lee JT. Guided by RNAs: X-Inactivation as a Model for lncRNA Function. J Mol Biol. 2013 Oct 9;425(19):3698–706.
8. Pontier DB, Gribnau J. Xist regulation and function eXplored. Hum Genet. 2011 Aug;130(2):223–36.
9. Duszczyk MM, Wutz A, Rybin V, Sattler M. The Xist RNA A-repeat comprises a novel AUCG tetraloop fold and a platform for multimerization. RNA. 2011 Nov;17(11):1973–82.
10. Arieti F, Gabus C, Tambalo M, Huet T, Round A, Thore S. The crystal structure of the Split End protein SHARP adds a new layer of complexity to proteins containing RNA recognition motifs. Nucleic Acids Res. 2014 Jun 2;42(10):6742–52.
11. Paniagua R. Biología celular y molecular. 4ª ed. Madrid: McGraw-Hill; 2017.
12. Jorde L, Carey J, Bamshad M. Medical genetics. 5th. ed. Sant Lake City: Elsevier; 2015.
13. Strachan T, Read A. Human molecular genetics. 5th. ed. Florida: Garland Science; 2019.


