El boom de los miRNA
Ana Paola Cano Morris, María del Rosario García Sánchez, Luna Guerra Núñez. Biología Sanitaria, UAH.
Índice
¿Qué son los miRNA?
Los microRNA son cadenas pequeñas de RNA no codificante formadas por unos 22 nucleótidos, que intervienen en la regulación de RNA. Al tener de diana a gran parte de los transcritos codificantes de proteínas, los miRNA están involucrados en casi todos los procesos patológicos y de desarrollo de organismos.
Algunos estudios plantean la hipótesis de que los miRNA provienen de una evolución de RNAs pequeños en eucariotas para suprimir material genético y transcritos no deseados.
¿Cómo se forman?
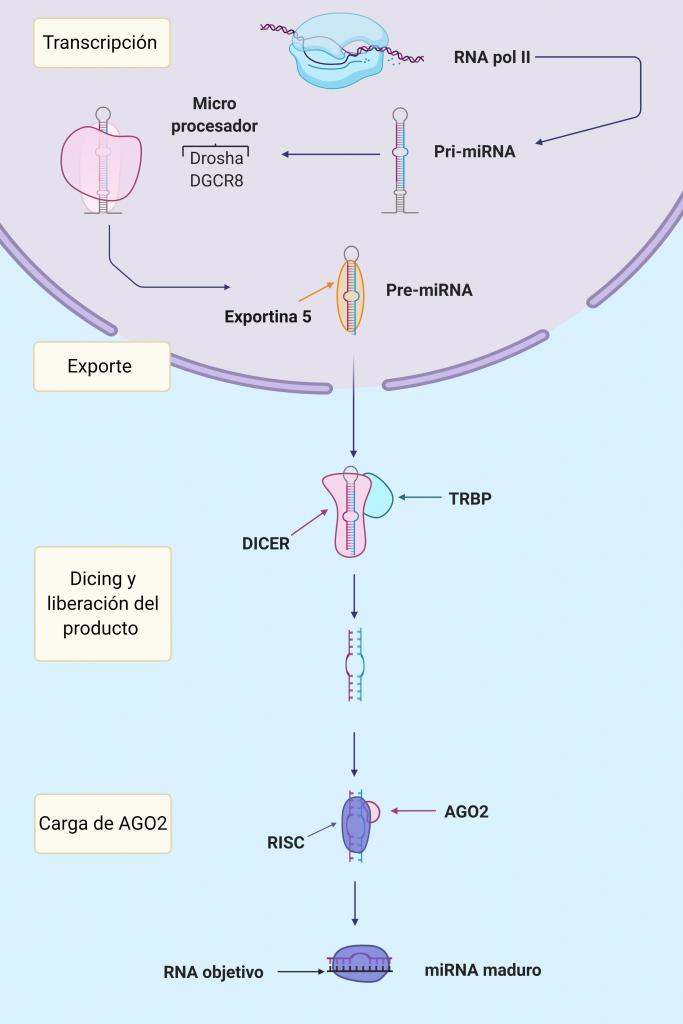
La mayoría de los miRNA son transcritos en el núcleo por la RNA polimerasa II como intrones o exones de RNAs no codificantes, siendo por tanto, su expresión regulada por factores de transcripción o reguladores epigenéticos. Como resultado se generará un primiRNA de unas 1000 bases que deberá sufrir una maduración hasta ser un miRNA funcional.
En estas secuencias aparecen pequeñas horquillas de unos 33-35 pares de bases en cuyos extremos hay un bucle y segmentos de cadena simple 5′ y 3′ que unirán las diferentes horquillas.
En el comienzo de la maduración participa la proteína Drosha, una endonucleasa RNasa-III cuya acción es sobre RNA de cadena doble, es decir, actuará directamente sobre la horquilla formada anteriormente. También colabora en este punto el cofactor DGCR8, cuya actividad principal es aumentar la interacción del Microprocesador que forman con el primiRNA.
Es importante que el tamaño de la horquilla sea regular para una correcta maduración. Este Microprocesador realiza un corte sobre el RNA de doble cadena aproximadamente a 11 pares de bases de la unión basal (lugar en el que comienza la doble cadena) y a 22 de la unión apical (lugar donde se sitúa el loop terminal de la horquilla). Si como hemos dicho antes, la horquilla tiene un tamaño de entre 33-35 pares de bases, el Microprocesador realizará un corte en el sitio correcto y se formará un premiRNA; este corte no es perfecto, sino que deja en el extremo 3′ dos nucleótidos de más que quedan desapareados. Es importante que esta nueva molécula también tenga en su extremo 5′ un monofosfato; ambas estructuras permitirán la correcta interacción con el resto de proteínas. El Microprocesador es susceptible de modificaciones que regulen tanto su expresión como su función, pudiendo ser la formación de miRNAs regulada en múltiples puntos.
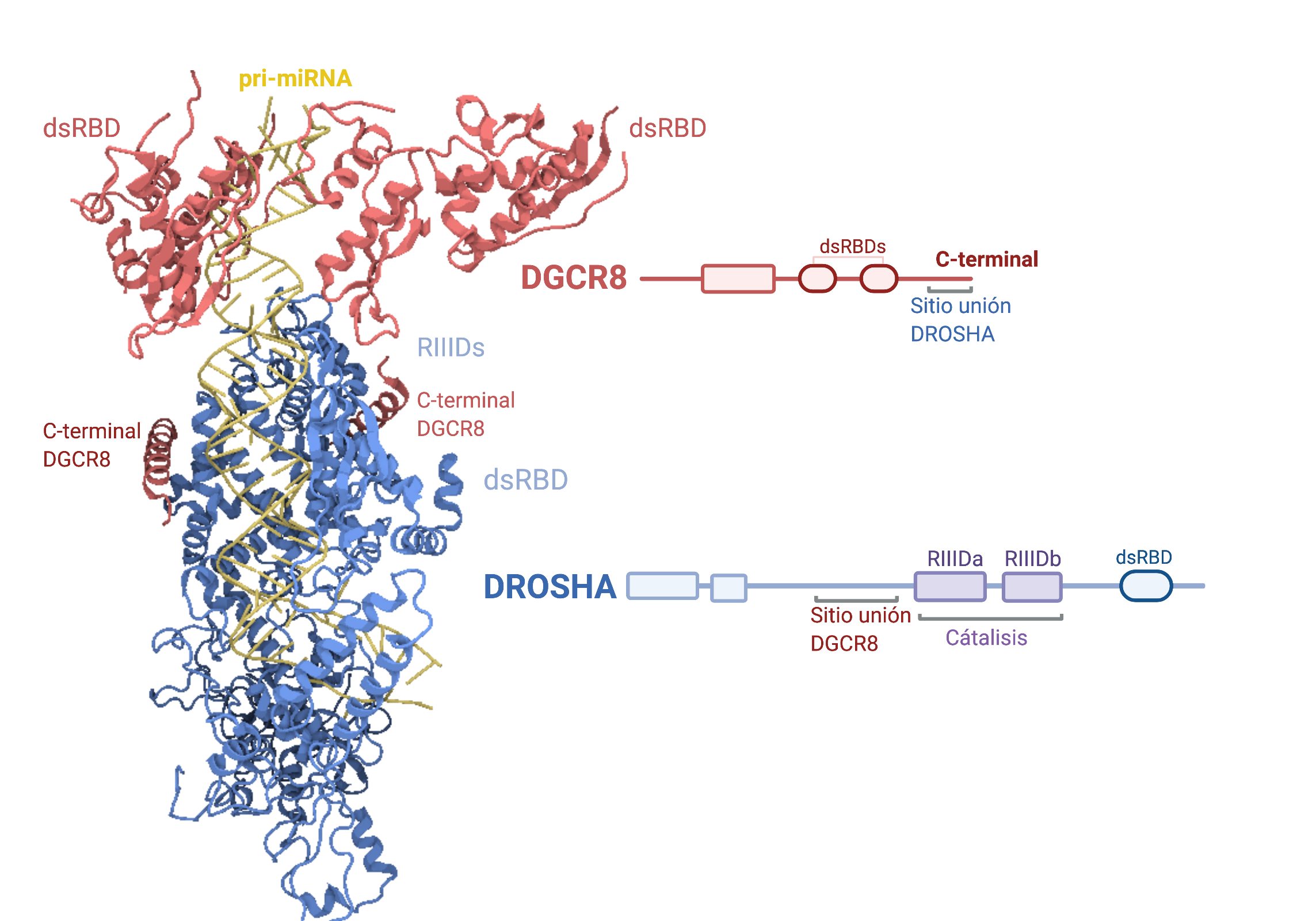
Imagen creada con Biorender.
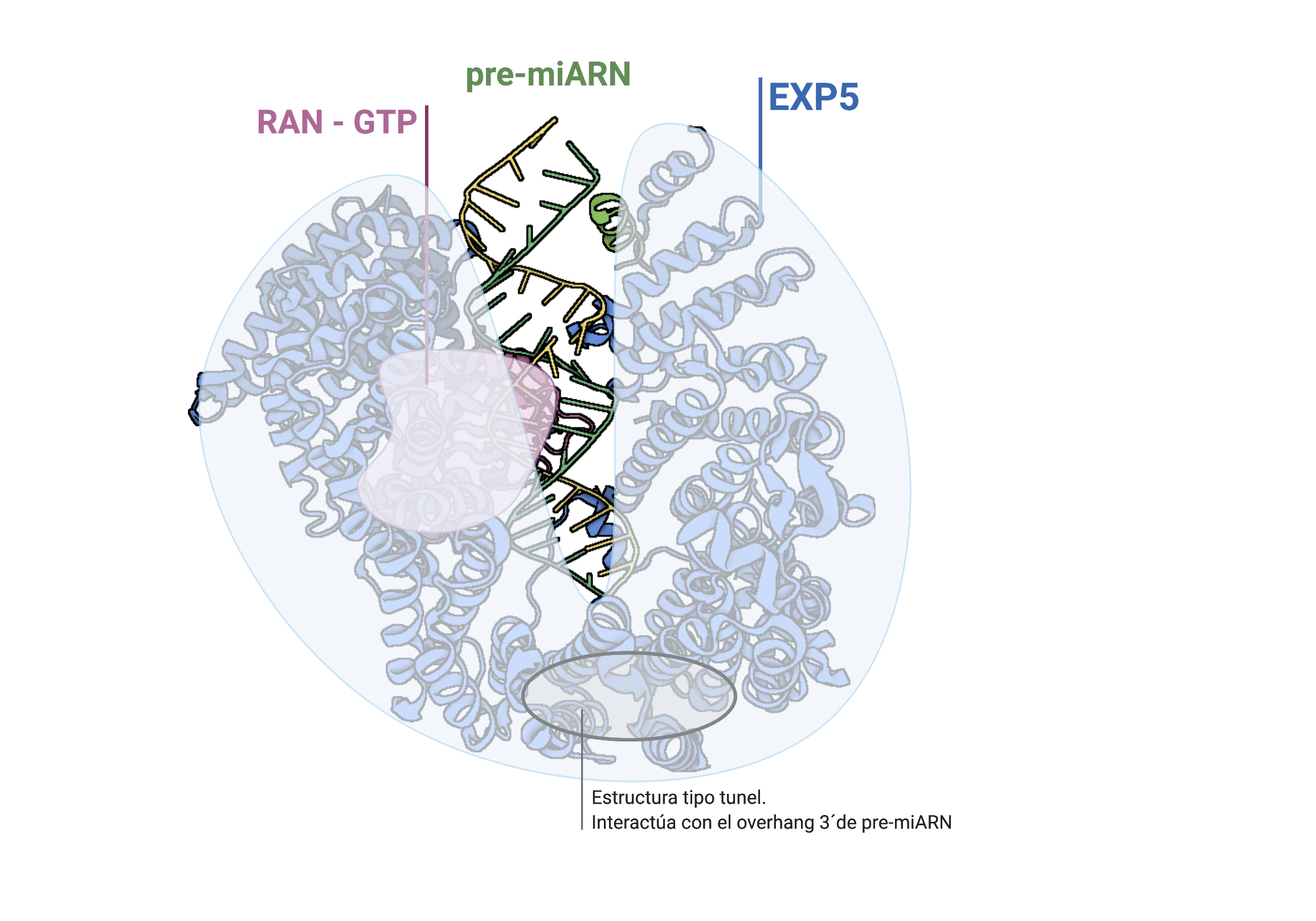
Una vez formado el premiRNA el resto de su maduración se realizará en el citoplasma. El exporte de la molécula es llevado a cabo por la proteína Exportina 5 (EXP5), que reconoce la estructura del RNA de doble cadena e interacciona con el saliente en su extremo 3′. Se forma un complejo en forma de canasta con la proteína RAN-GTP y el premiRNA que interacciona con el poro nuclear. El GTP es hidrolizado después de la translocación del complejo, de forma que este se desensambla y el premiRNA queda libre en el citoplasma. En algunos tumores la proteína EXP5 está truncada por una mutación en el gen que la codifica, impidiendo la maduración de miRNAs.
En el siguiente paso de la maduración, interviene la proteína Dicer acompañada de la proteína TRBP que aumenta su eficacia. Dicer, al igual que Drosha, es una endonucleasa RNasa III cuya función es realizar un corte en el premiRNA cerca del loop terminal de la horquilla. Así se liberará una corta cadena de RNA duplexo de unos 22 pares de bases, que será el futuro miRNA. La proteína Dicer necesitará de los extremos 5′ fosforilado y 3′ desapareado para su correcta interacción y funcionamiento.
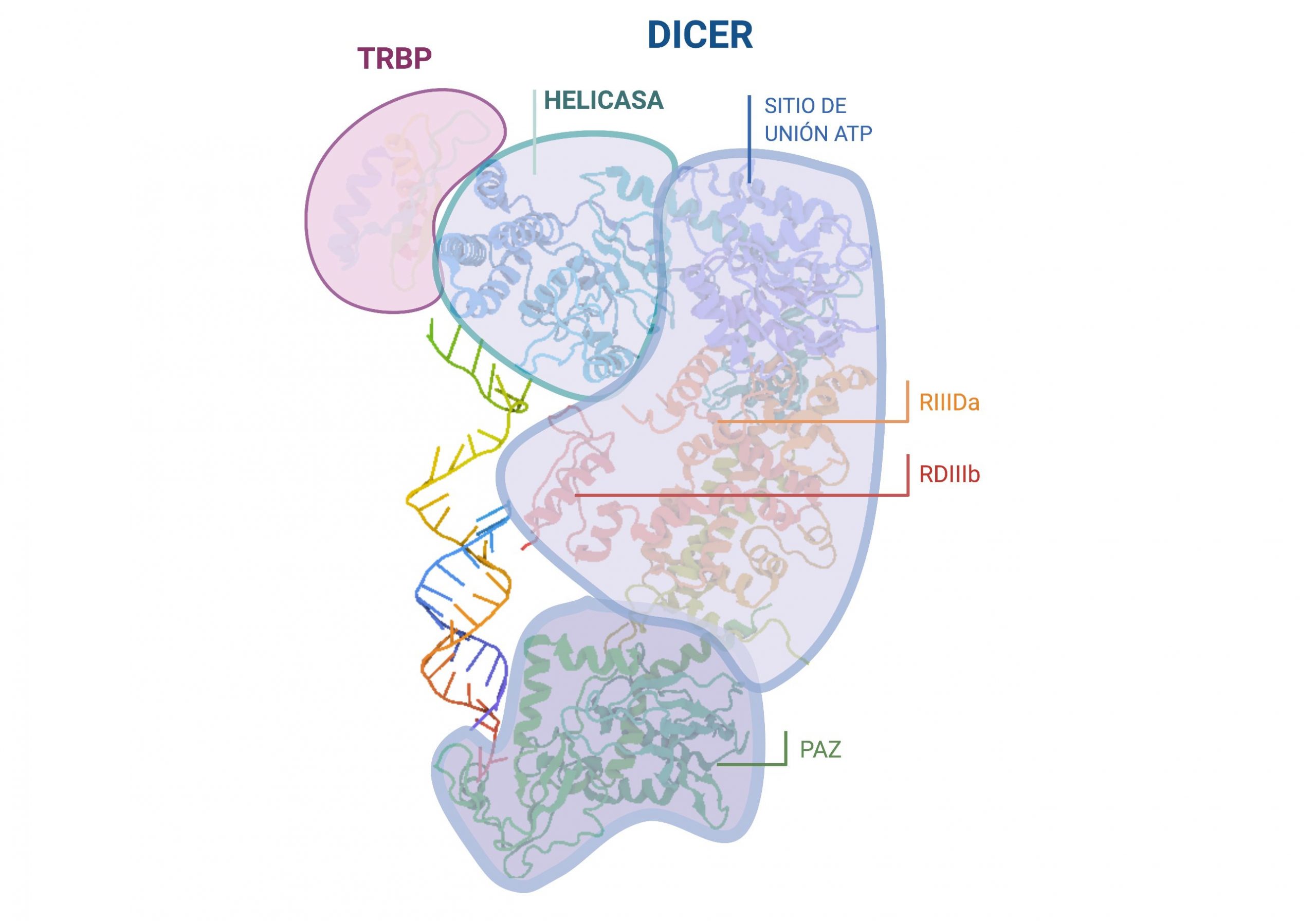
Imagen creada con Biorender.
La pequeña doble cadena de RNA se asocia a continuación con a proteína AGO, formando el complejo silenciador inducido por RNA (RISC). Será otra vez necesario que los extremos 5′ y 3′ estén correctamente formados para una apropiada interacción con la proteína. Para la carga del miRNA en la proteína AGO, es necesaria la formación un complejo intermedio denominado RLC compuesto por el miRNA, la proteína Dicer explicada antes y la nueva AGO. En humanos existen hasta 4 tipos de la proteína AGO, pero se unen indistintamente a los miRNAs para la formación del complejo.
Por el momento seguimos teniendo el miRNA como una cadena doble, por lo que debemos referirnos al complejo como pre-RISC. Por último, se deberá eliminar la cadena que no vaya a funcionar como guía en el complejo por medio de la endonucleasa C3PO, teniendo ya así el complejo RISC con un miRNA funcional. La selección de la cadena guía se realiza en función de la estabilidad termodinámica de los extremos de la cadena, normalmente es elegida la que posee un extremo 5′ menos estable; la cadena descartada será rápidamente degradada (será una pequeña cadena de RNA sin ningún tipo de protección). No siempre es elegida la misma cadena, ya que existe cierta preferencia hacia una de ellas en un 96-99% de los casos. La cadena de miRNA menos frecuente, aún siendo también activa, será menos efectiva y aparece señalada como miRNA*.
Se acaba de explicar el funcionamiento de la biogénesis de miRNA de forma canónica. Por el contrario, no es la única vía y existen otras vías alternativas. Destacaremos los mirtrons, los cuales se forman desde los intrones de mRNAs inmaduros y también forman una horquilla; producto del splicing que sufren los mRNA en su maduración. Estas secuencias de RNA serán similares a premiRNA y se introducirán en el proceso de síntesis de miRNA habiéndose saltado el procesamiento por Drosha y DGCR8.
¿Cómo funcionan?
Como se ha adelantado antes, la complementariedad de bases entre el complejo RISC y el mRNA a regular es crucial para que el miRNA pueda llevar a cabo su función. La regulación del producto codificado por el mRNA en cuestión puede llevarse a cabo por dos vías:
- Degradación del mensajero
- Represión de la traducción
Ambas dependerán en el grado de complementariedad al que se llegue entre el miRNA y el mRNA.
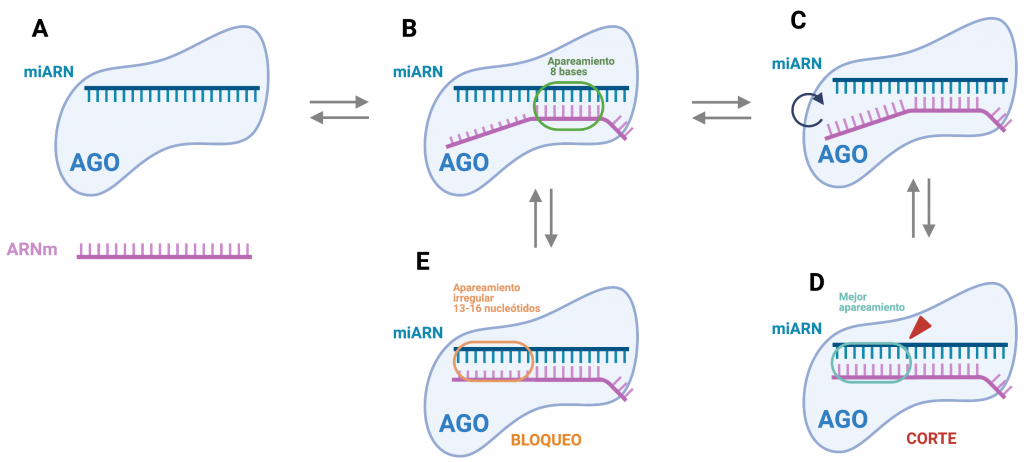
Imagen creada con Biorender. [2]
Principalmente interactúan los nucleótidos de 2-8 del miRNA, altamente conservados, en el extremo 5’ (conocida como secuencia “seed”) con la región 3’UTR del mRNA, aproximadamente 15 nucleótidos después del codón de paro y junto a una secuencia rica en AU. Actualmente se sabe que la secuencia diana puede también encontrarse en la región codificante (ORF) o 5’UTR, aunque menos frecuente y eficaz que la unión canónica.
Que sean esos nucleótidos presentados al mRNA no es algo casual, viene determinado por la disposición del complejo RISC que consigue presentar este fragmento concreto de la cadena guía en forma de hélice A, lo que aumenta la afinidad y especificidad con el mRNA. Se ha estudiado que un segmento mayor supondría un problema para la unión, mientras que uno menor no tendría la especificidad suficiente para su buen funcionamiento.
El miRNA debe estar totalmente cubierto por la proteína AGO para evitar su degradación por RNasas. Solo se muestra la secuencia “seed” que se unirá específicamente al mRNA. En este punto ocurre la represión de la traducción. Existen varias hipótesis sobre este mecanismo:
- Ralentización o paralización de los ribosomas en un punto posterior a la iniciación de la traducción.
- Marcaje del polipéptido para su posterior degradación
- Prevenir la interacción entre el promotor y su activador traduccional
Para que ocurra la degradación del mRNA es necesario alcanzar un grado mayor de complementariedad con el miRNA. Después de la unión típica del extremo 5’ hay un cambio conformacional del complejo que permite extender la interacción en las regiones central y 3’ de la cadena, después la proteína AGO vuelve a su conformación típica. Es en este momento cuando puede ocurrir la degradación, realizándose un corte entre los nucleótidos 10 y 11. Es la proteína AGO quien posee una actividad endonucleolítica capaz de realizar el corte. Los fragmentos del mRNA serán posteriormente degradados, mientras que el miRNA permanece intacto para poder seguir actuando.
En eucariotas, los complejos RISC se encuentran principalmente asociados a Cuerpos P, zonas electrodensas donde se acumulan enzimas implicadas en la degradación de mRNA, siendo así considerado un lugar para la regulación postranscripcional de mRNA.
Se sabe que existe una función cooperativa de los miRNA ya que aparecen múltiples sitios de unión a estas moléculas en los mRNA, multiplicando así las respuestas de cada sitio de unión trabajando por si mismo. De esta forma el mecanismo de acción es más sensible a cambios en los niveles de miRNA. Los miRNA que se unieran al mismo mRNA también podrían ser distintos, debiendo tener en cuenta todas las interacciones a la hora de determinar una patología o tratamiento.
Los miRNA normalmente no tienen un mensajero diana determinado, sino que pueden regular la expresión de varios genes. Esto es posible en primer lugar, gracias a la conservación de los sitios de unión en las regiones 3’UTR de modo que aparecen compartidos entre una familia de genes. Por otra parte, la complementariedad entre la región 3’UTR y el miRNA no es completa en la mayoría de casos, sino que solo se da en una pequeña secuencia de 7 nucleótidos, lo suficientemente corta como para poder estar en otros mRNA.
Transporte de miRNA
Tan importante como conocer su funcionamiento en el interior celular, es conocer su transmisión entre diferentes células. Dependiendo de los mecanismos de transporte intercelular que utilicen, su transmisión tendrá características diferentes, las cuales afectaran a los procesos fisiológicos y patológicos. En el caso de estos últimos, determinaran la capacidad de diseminación y extensión de la enfermedad en el organismo.
Las principales vías de comunicación intercelular son: la señalización dependiente de contacto célula-célula (uniones intercelulares), la señalización mediante moléculas solubles y la señalización mediante vesículas extracelulares.
En el caso de los miRNA se ha demostrado su transmisión mediante: microvesículas, HDL, lipoproteínas, riboproteínas, cuerpos apoptóticos, exosomas y gap junctions. Estos dos últimos procesos serán los que explicaremos en profundidad debido a la gran importancia que están teniendo en las últimas investigaciones sobre miRNA.
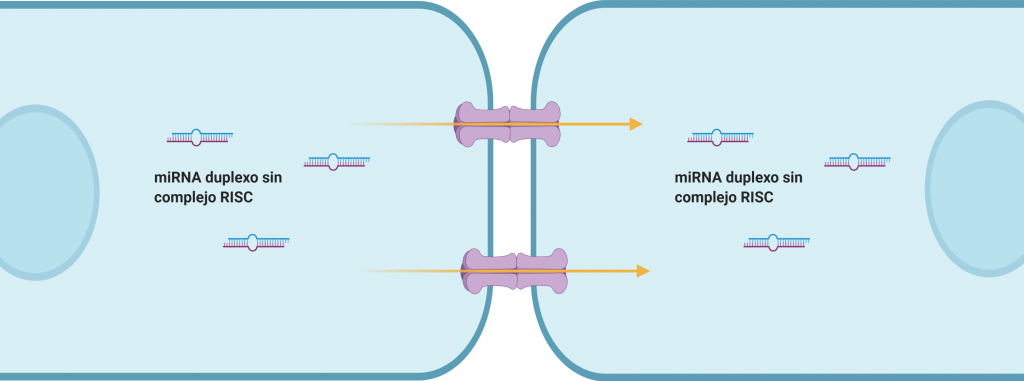
Gap-junctions
Son microdominios de la membrana plasmática de células adyacentes, en los cuales se encuentran conexinas (en los invertebrados se denominan inexinas) formando canales proteicos que permiten un intercambio rápido de sustancias entre ambas células mediante el transporte directo de moléculas de pequeño tamaño.
Diversos estudios han demostrado la existencia de un trasporte de miRNA desnudos, es decir, sin el complejo RISC. Sin embargo, aún se desconoce el mecanismo molecular de dicho transporte.
Exosomas:
Los exosomas fueron descritos por primera vez por Pan y Johnstone en 1983. Son vesículas extracelulares de entre 40 y 120 nm de diámetro constituidos por una bicapa lipídica. Pueden transportar moléculas como: lípidos, proteínas, RNA… Además, se han detectado en la mayoría de líquidos corporales, lo cual los convierte en potenciales biomarcadores para ciertas enfermedades.
La biogénesis de los exosomas se produce siguiendo el siguiente proceso: Primero se forman endosomas tempranos a partir de la invaginación de la membrana celular. Seguidamente, se generan invaginaciones de la membrana del endosoma, dando lugar a múltiples vesículas intraluminales (ILVs), lo que resulta en la formación de cuerpos multivesiculares (MVBs). El contenido es seleccionado e introducido en las ILVs, que cuando sean secretadas al exterior celular por exocitosis, pasarán a considerarse exosomas.
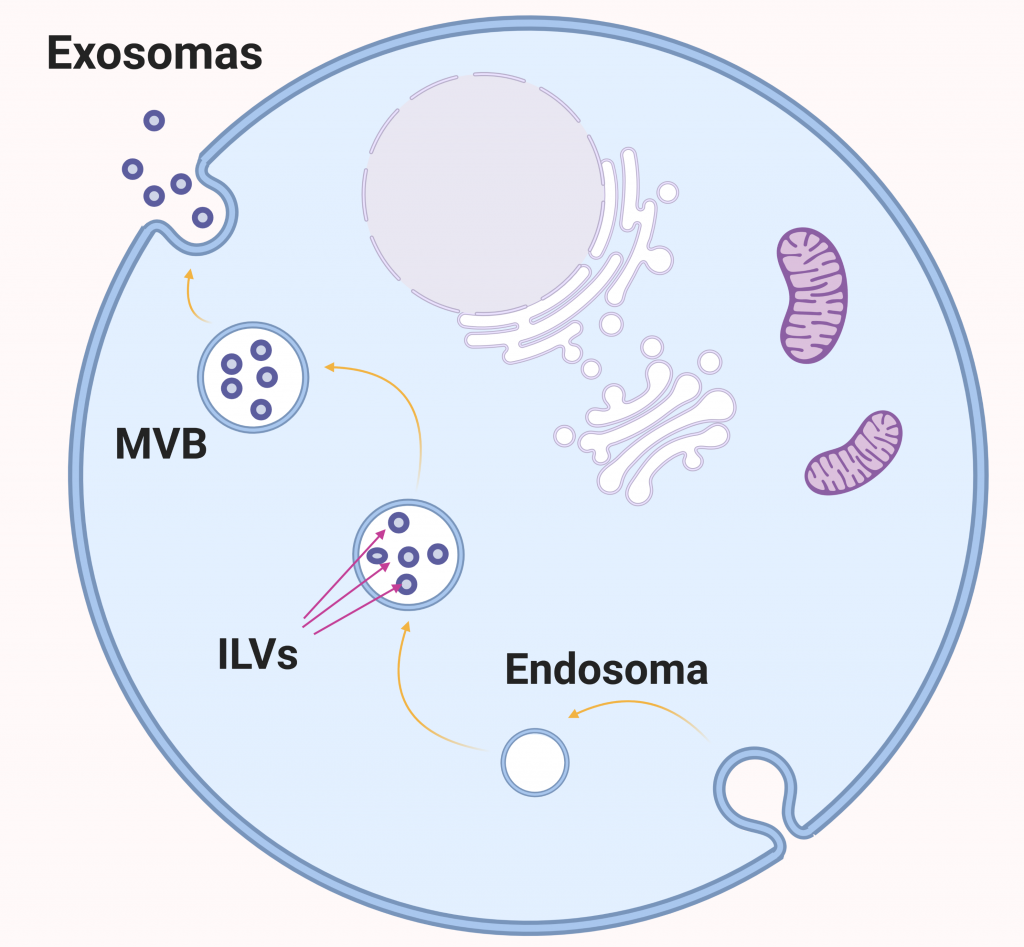
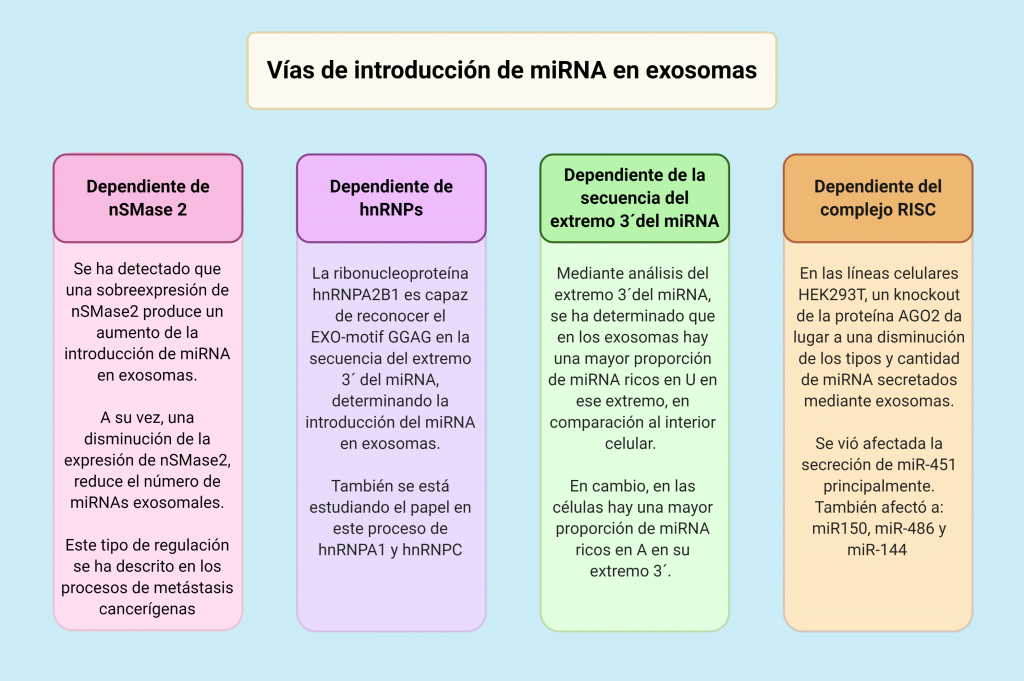
El mecanismo para la introducción de los miRNA en los exosomas aún no está completamente estudiado y descrito, siendo necesaria una mayor investigación en ese campo. Las cuatro vías que vamos a exponer a continuación han sido descritas en diversos estudios y actualmente son la principal hipótesis para explicar dicho proceso.
MiRNA en el ámbito clínico
Fallos en la regulación de la expresión y la función de los miRNA, ya sea por alteraciones genómicas o cambios en su biogénesis, han mostrado ser factores importantes en el desarrollo y la progresión de múltiples enfermedades.
Los miRNA son por lo tanto, un área de estudio importante para futuras técnicas de diagnóstico y tratamiento.
MiRNA como biomarcadores
Los miRNA tienen el potencial de ser el biomarcador perfecto; se expresan de forma ubicua, se pueden aislar fácilmente y cuantificar con gran sensibilidad y especificidad en tejidos o fluidos. Además, son estables, tienen especificidad celular y relevancia fisiológica en estados patológicos.
Sin embargo, aún no se logra una estandarización en cuanto a su análisis, y la expresión y función de los miRNA ha presentado variabilidad en distintas poblaciones.
Los principales test de diagnostico en el mercado son útiles para distintos tipos de cáncer y enfermedades relacionadas con la edad, esto mediante el análisis de varios miRNA. Aun así, hay proyectos en desarrollo que se centran en los niveles de un único miRNA.
En los últimos años, muchos estudios han propuesto el uso clínico de los exosomas con miRNA como posibles biomarcadores para la detección y seguimiento de enfermedades en las que participan. Esto se debe a las siguientes características de los miRNAS exosomales:
- No todos los miRNA son transportados por exosomas.
- Algunos miRNA solo han sido detectados en exosomas en estados patológicos.
- Se han detectado diferencias en los niveles de secreción de exosomas con miRNA, además de una diferente concentración de estos en su interior, tanto entre estado fisiológico normal y estado patológico, como en la evolución de la enfermedad.
Ante estos resultados, el departamento de Hematología/Oncología del Hospital Pediátrico Bambino Gesù ha propuesto el análisis de los miRNA exosomales mediante RT-qPCR para la detección del cáncer infantil y un mejor seguimiento del progreso de la enfermedad, además de su reacción a los diferentes tratamientos.
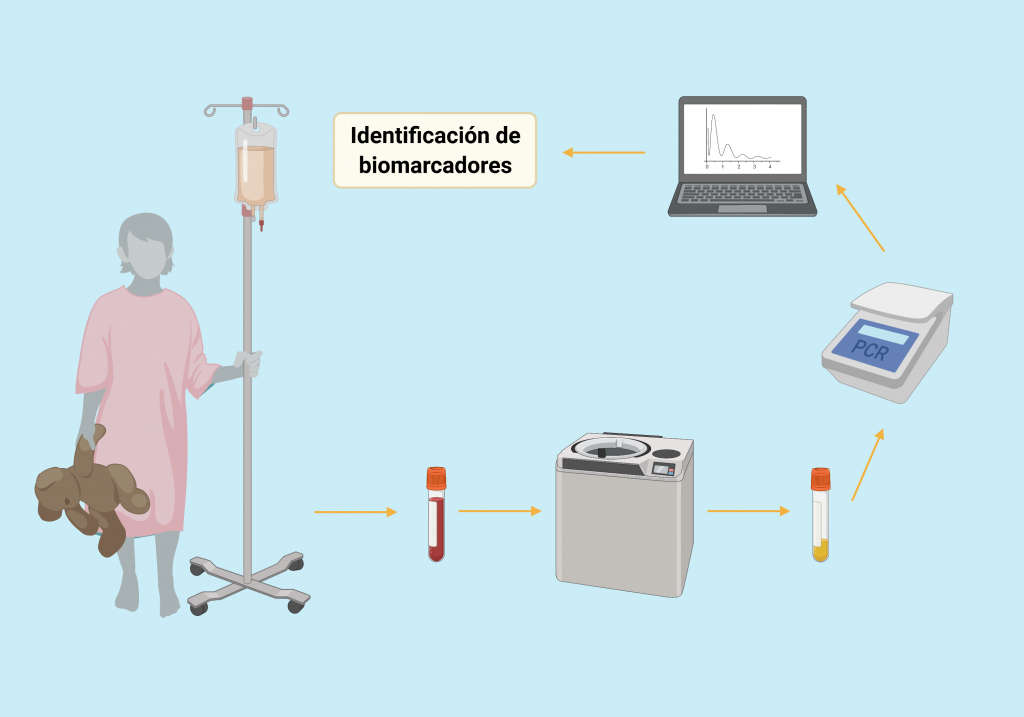
Diagnóstico con miRNA exosomal en pacientes oncológicos A partir de la extracción de sangre del paciente, se realiza un ultracentrifugado para la separación del plasma. Una vez obtenido la fracción con los exosomas, se realiza una RT-qPCR. Los datos se envían a un programa para su análisis, terminando en la identificación y cuantificación de los biomarcadores. [3]
MiRNA como terapia
Debido a sus patrones de expresión y su habilidad de unirse a varias dianas, usualmente en un mismo proceso biológico, los miRNA pueden potencialmente regular la expresión de múltiples elementos en rutas de señalización conocidas por estar afectadas en determinados estados patológicos.
Sin embargo, la baja especificidad de unión, aumenta el peligro de efectos secundarios importantes producto de la afectación de otras rutas fuera de la diana deseada.
Es indispensable así el desarrollo de herramientas que permitan una predicción más exacta de la unión miRNA – mRNA, validando su uso terapéutico.
MiRNA “mimics” y “AntagomiRs”
Distintas patologías se caracterizan por alteraciones en la expresión de miRNA; tanto sobreexpresión como infraexpresión. Es por ello que las terapias basadas en miRNA, buscan principalmente aumentar o reducir los niveles de un miRNA especifico, según sea el caso.
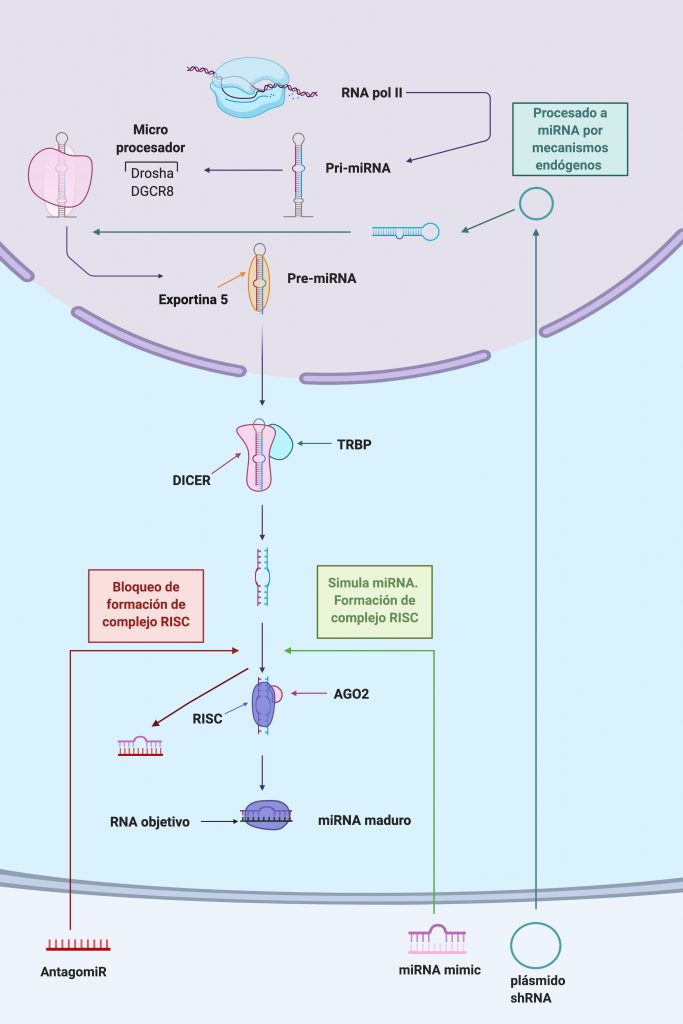
Imagen creada con Biorender. [1]
Infraexpresión de miRNA;
Para aumentar los niveles de un miRNA especifico, se utilizan “mimics” de miRNA. Son moléculas sintéticas de RNA que presentan la misma secuencia que el miRNA endógeno, pudiendo restablecer así los niveles y función normal de este.
También se utilizan “Short hairpin RNAs (shRNAs)”; moléculas sintéticas que son procesadas de forma endógena para dar miRNAs maduros. Los shRNAs han mostrado minimizar la toxicidad y permiten sintetizar varios miRNAs a partir de un solo promotor.
En cáncer, la mayoría de miRNAs se encuentran infraexpresados, muchos de estos regulan el ciclo celular y se consideran supresores de tumores, por lo que restablecer sus niveles puede complementar o potenciar la eficacia de otros tratamientos oncológicos.
Sobreexpresión de miRNA;
Para inhibir la función de un miRNA que se encuentra sobreexpresado, se utilizan “AntagomiRs”; moléculas sintéticas que se unen al miRNA endógeno, bloqueándolo y regulando su expresión.
Otra forma de inhibir la expresión de miRNAs, es utilizando sitios de unión competitivos. Estos sitios de unión suelen ser “Antisense oligonucleotides (ASOs)”. Estos ASOs se modifican para aumentar su resistencia a la degradación por nucleasas, su unión a proteínas plasmáticas, su complementariedad con el miRNA diana y su habilidad para activar el sistema inmune innato. Lo que tiene como resultado que los miRNAs tendrán mayor afinidad por estos ASOs sintéticos que por sus ARNm complementarios, viéndose inhibida su función.
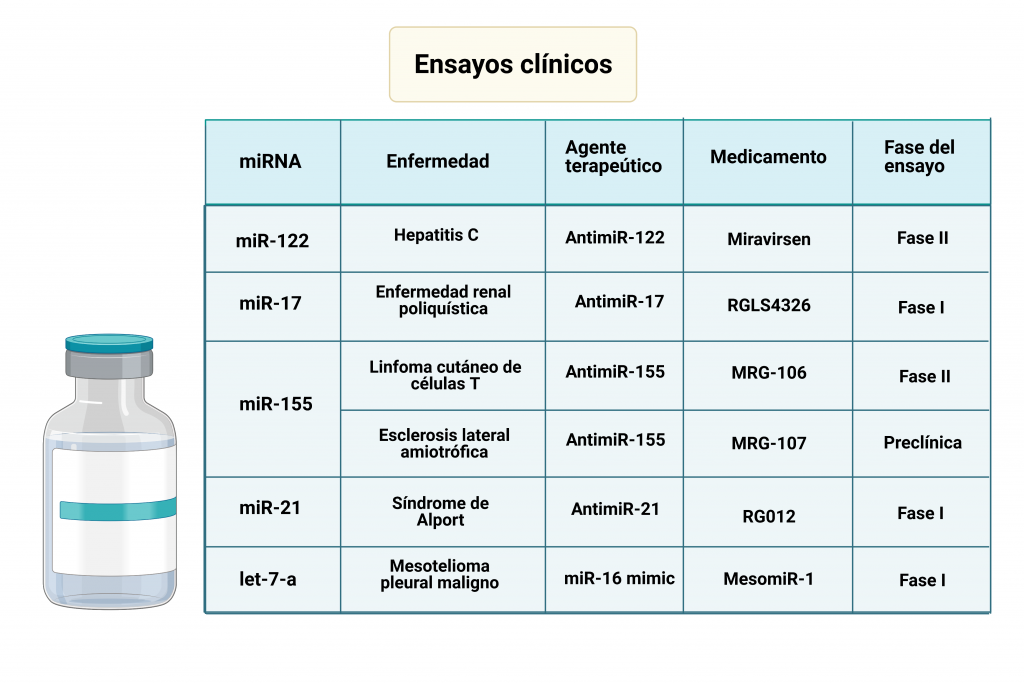
En la tabla se recogen datos de ensayos clínicos relacionados con el desarrollo de medicamentos basados en terapia mediante miRNA, exponiendo también su relación con algunas enfermedades. A continuación, vamos a exponer el funcionamiento de dos de ellos
- Miravirsen: La creación de dicho medicamento se basó en el funcionamiento del miR-122 en la infección por el virus de la hepatitis C (HCV). Este utiliza los miRNAs de las células infectadas durante su ciclo replicativo, siendo capaz de modular su expresión para favorecer la continuidad del virus. MicroRNA-122 (miR-122) es un miRNA específico del hígado, el cual se une a la región 5´UTR del genoma del HCV promoviendo la estabilidad de su RNA, produciéndose también su acumulación. El complejo formado entre el miR-122 y el HCV, protege el genoma del virus de la degradación por exonucleasas celulares, además de evitar una respuesta inmune innata. Al introducir el AntimiR-122, se inhibe el miR-122, evitándose el proceso anteriormente explicado. En los ensayos clínicos se ha visto que los niveles de miR-122 en plasma se reducen en gran medida tras la administración del medicamento.
- MRG-106 / Cobomarsen: La sobreexpresión de miR-155 está relacionada con el desarrollo algunos cánceres, debido a su papel en la proliferación y supervivencia celular y la inestabilidad del genoma de células malignizadas. Además, se han demostrado que algunos subtipos de este miRNA están implicados en el linfoma cutáneo de células T. La desregulacion de las vías JAK/STAT, NF-jB y PI3K/AKT tienen un papel muy importante en la patogénesis y progresión del linfoma cutáneo de células T. La activación de esas vías está relacionada con la sobreexpresión del miR-155. Se ha demostrado que el Cobomarsen o MRG-106 reduce la actividad de estas vías, siendo un potencial tratamiento de esta enfermedad.
Conclusión:
Desde el descubrimiento del primer microRNA su estudio se ha expandido considerablemente. Un mejor entendimiento del papel de los miRNA en el desarrollo y la enfermedad, los han convertido en herramientas y dianas atractivas para un enfoque terapéutico innovador. Es por esto que la comunidad científica tiene muchas esperanzas en el futuro desarrollo de tratamientos mediante medicina con miRNAs, ya que es notablemente más eficaz que la realizada con DNA. Sin embargo, la regulación mediada por miRNAs es una red muy compleja, de la cual todavía desconocemos gran parte de su funcionamiento y por lo tanto, aún ignoramos el verdadero alcance de su potencial, por lo que es necesaria una mayor investigación en este campo.
Bibliografía
[1] Bajan, S. and Hutvagner, G. (2020) ‘RNA-Based Therapeutics: From Antisense Oligonucleotides to miRNAs’, Cells, 9(1), pp. 1–27. doi: 10.3390/cells9010137.
[2] Bartel, D. P. (2009) ‘MicroRNAs: Target Recognition and Regulatory Functions’, Cell, 136(2), pp. 215–233. doi: 10.1016/j.cell.2009.01.002.
[3] Galardi, A., Colletti, M., Di Paolo, V., Vitullo, P., Antonetti, L., Russo, I. and Di Giannatale, A. (2019) ‘Exosomal MiRNAs in pediatric cancers’, International Journal of Molecular Sciences, 20(18). doi: 10.3390/ijms20184600.
[4] Ha, M. and Kim, V. N. (2014) ‘Regulation of microRNA biogenesis’, Nature Reviews Molecular Cell Biology. Nature Publishing Group, 15(8), pp. 509–524. doi: 10.1038/nrm3838.
[5] Monaghan, M., Pandit, A., Bartel, D. P., Lee, R. and Feinbaum, R. (2008) ‘MicroRNAs : Genomics , Biogenesis , Mechanism , and Function Genomics : The miRNA Genes’, Advanced Drug Delivery Reviews, 116(4), pp. 197–208.
[6] Rupaimoole, R. and Slack, F. J. (2017) ‘MicroRNA therapeutics: Towards a new era for the management of cancer and other diseases’, Nature Reviews Drug Discovery. Nature Publishing Group, 16(3), pp. 203–221. doi: 10.1038/nrd.2016.246.
[7] Saliminejad, K., Khorram Khorshid, H. R., Soleymani Fard, S. and Ghaffari, S. H. (2019) ‘An overview of microRNAs: Biology, functions, therapeutics, and analysis methods’, Journal of Cellular Physiology, 234(5), pp. 5451–5465. doi: 10.1002/jcp.27486.
[8] Seto, A. G., Beatty, X., Lynch, J. M., Hermreck, M., Tetzlaff, M., Duvic, M. and Jackson, A. L. (2018) ‘Cobomarsen, an oligonucleotide inhibitor of miR-155, co-ordinately regulates multiple survival pathways to reduce cellular proliferation and survival in cutaneous T-cell lymphoma’, British Journal of Haematology, 183(3), pp. 428–444. doi: 10.1111/bjh.15547.
[9] Van Der Ree, M. H., Van Der Meer, A. J., Van Nuenen, A. C., De Bruijne, J., Ottosen, S., Janssen, H. L., Kootstra, N. A. and Reesink, H. W. (2016) ‘Miravirsen dosing in chronic hepatitis C patients results in decreased microRNA-122 levels without affecting other microRNAs in plasma’, Alimentary Pharmacology and Therapeutics, 43(1), pp. 102–113. doi: 10.1111/apt.13432.
[10] Zhang, J., Li, S., Li, L., Li, M., Guo, C., Yao, J. and Mi, S. (2015) ‘Exosome and exosomal microRNA: Trafficking, sorting, and function’, Genomics, Proteomics and Bioinformatics, 13(1), pp. 17–24. doi: 10.1016/j.gpb.2015.02.001.


 Entrada siguiente
Entrada siguiente
Un estudio muy prometedor. Enhorabuena
me vino super bien este articulo, de verdad !me salvaron de un trabajo horrible!
muchas gracias por compartirlo 🙂